Сценарии моделирования
SimBiology.Scenarios - это объект, который позволяет создавать различные сценарии моделирования на основе различных значений проб величин модели. Можно объединить эти величины с различными дозами или вариантами и смоделировать различные сценарии для изучения моделей поведения в различных экспериментальных условиях и режимах дозирования.
sObj = SimBiology.Scenarios(name,content)Scenarios объект sObj с одной записью. name является именем количества модели или именем группы вариантов или доз для генерации сценария. content содержит соответствующие числовые значения для величины модели или вектора вариационных объектов или вектора дозовых объектов.
sObj = SimBiology.Scenarios(quantityNames,probDist,Name,Value)quantityNames из совместного распределения вероятностей probDist. Укажите дополнительные параметры для распределения вероятностей и метода выборки, используя один или несколько аргументов пары имя-значение. Для определения распределения вероятностей необходимо иметь Toolbox™ статистики и машинного обучения.
name - Наименование записиИмя записи, указанное как символьный вектор или строка.
Можно задать имя записи как имя количества модели (вида, параметра или отсека). Кроме того, можно определить имя для группы доз или вариантов, которые будут включены в генерацию образцов (сценариев).
Пример: "k1"
Типы данных: char | string
content - Модельные количественные значения или вектор доз или вариантовRepeatDose или ScheduleDose объекты | вектор объектов-вариантовЗначения количества модели или вектор доз или вариантов, указанный как числовой вектор, вектор RepeatDose или ScheduleDose объекты или вектор variant объекты.
Если указано наименование количества для name входной аргумент, набор content к числовому вектору.
Если указано имя для группы доз или вариантов, установите content к вектору дозовых объектов или вектору вариационных объектов.
Пример: [0.5,1,1.5]
quantityNames - Наименования величин моделиИмена величин модели для генерации образца (сценария), указанных как символьный вектор, строка, строковый вектор или массив ячеек символьных векторов.
Пример: ["k12","k21"]
Типы данных: char | string | cell
probDist - Распределения вероятностейРаспределения вероятностей для формирования выборочных значений для величин модели, заданных как вектор объектов распределения вероятностей, символьный вектор, строка, строковый вектор или массив ячеек символьных векторов, содержащих имена поддерживаемых распределений вероятностей. Для определения распределения вероятностей необходимо иметь панель инструментов статистики и машинного обучения.
Используйте makedist (Statistics and Machine Learning Toolbox) используется для создания объектов распределения. Список поддерживаемых распределений см. в разделе distname (Statistics and Machine Learning Toolbox).
Пример: [pd1,pd2]
Укажите дополнительные пары, разделенные запятыми Name,Value аргументы. Name является именем аргумента и Value - соответствующее значение. Name должен отображаться внутри кавычек. Можно указать несколько аргументов пары имен и значений в любом порядке как Name1,Value1,...,NameN,ValueN.
'Number',10 определяет создание 10 выборок.'Number' - Количество образцов[] (по умолчанию) | положительный скалярКоличество выборок, извлекаемых из вероятностных распределений, указанных как пара, разделенная запятыми, состоящая из 'Number' и положительный скаляр. Значение по умолчанию [] означает, что функция выводит количество выборок из других записей. Если номер не может быть выведен, ему присваивается значение 2.
Пример: 'Number',5
'RankCorrelation' - Матрица ранговой корреляции[] (по умолчанию) | числовая матрицаМатрица ранговой корреляции для совместного распределения вероятностей, определяемая как разделенная запятыми пара, состоящая из 'RankCorrelation' и числовую матрицу. Поведение по умолчанию заключается в том, что когда оба 'RankCorrelation' и 'Covariance' имеют значение [], SimBiology.Scenarios извлекает некоррелированные выборки из совместного распределения вероятностей.
Невозможно указать 'RankCorrelation' если 'Covariance' установлен. Число столбцов в матрице должно соответствовать числу указанных распределений. Матрица должна быть симметричной с диагональными значениями 1. Все его собственные значения также должны быть положительными.
Пример: 'RankCorrelation',[1 0.3;0.3 1]
'Mean' - Средние значенияСредние значения величин, указанные как разделенная запятыми пара, состоящая из 'Mean' и числовой вектор.
Можно указать средние значения только для обычных распределений. Число средних значений должно равняться числу указанных вероятностных распределений.
Пример: 'Mean',[0.5,1.5]
'Covariance' - Ковариационная матрица[] (по умолчанию) | числовая матрицаКовариационная матрица для совместного распределения вероятностей, указанная как разделенная запятыми пара, состоящая из 'Covariance' и числовую матрицу. Поведение по умолчанию таково, если оба 'RankCorrelation' и 'Covariance' имеют значение [], SimBiology.Scenarios извлекает некоррелированные выборки из совместного распределения вероятностей. Невозможно указать 'Covariance' при указании 'RankCorrelation'.
Ковариационную матрицу можно задать только для нормальных распределений. Число столбцов в матрице должно соответствовать числу указанных распределений. Все его собственные значения также должны быть неотрицательными.
Пример: 'Covariance',[0.25 0.15;0.15 0.25]
'SamplingMethod' - Метод отбора проб'random' (по умолчанию) | 'lhs' | 'copula' | 'sobol' | 'halton'Метод выборки, указанный как разделенная запятыми пара, состоящая из 'SamplingMethod' и символьный вектор или строку. В зависимости от того, распределения вероятностей с 'RankCorrelation' или нормальные распределения с 'Covariance' указаны, методы отбора проб различаются.
Если запись содержит (совместное) нормальное распределение с Covariance указаны следующие методы отбора проб:
'random' - Извлекать случайные выборки из указанного нормального распределения с помощью mvnrnd (Статистика и инструментарий машинного обучения).
'lhs' - Нарисуйте образцы латинского гиперкуба из указанных обычных распределений с помощью lhsnorm (Статистика и инструментарий машинного обучения). Дополнительные сведения см. в разделе Создание квазислучайных чисел (панель инструментов статистики и машинного обучения).
Если запись содержит (совместное) распределение без Covariance указаны следующие методы отбора проб:
'random' - Извлекать случайные выборки из указанных вероятностных распределений с помощью random (Статистика и инструментарий машинного обучения).
'lhs' - Рисование выборок латинского гиперкуба из указанных распределений вероятностей с использованием алгоритма, аналогичного lhsdesign (Статистика и инструментарий машинного обучения). Этот подход представляет собой более систематический подход к заполнению пространства, чем случайная выборка. Дополнительные сведения см. в разделе Создание квазислучайных чисел (панель инструментов статистики и машинного обучения).
'copula' - Нарисуйте случайные выборки с помощью пары (Statistics and Machine Learning Toolbox). Используйте эту опцию, чтобы наложить корреляции между образцами с помощью копул.
'sobol' - Использовать последовательность собол (sobolset (Statistics and Machine Learning Toolbox)), который преобразуется с помощью функции обратного кумулятивного распределения (icdf (Statistics and Machine Learning Toolbox)) указанных распределений вероятностей. Этот метод используется для планомерного заполнения пространства. Дополнительные сведения см. в разделе Создание квазислучайных чисел (панель инструментов статистики и машинного обучения).
'halton' - Использовать последовательность halton (haltonset (Statistics and Machine Learning Toolbox)), который преобразуется с помощью функции обратного кумулятивного распределения (icdf (Statistics and Machine Learning Toolbox)) указанных распределений вероятностей. Дополнительные сведения см. в разделе Создание квазислучайных чисел (панель инструментов статистики и машинного обучения).
Если нет Covariance указывается, SimBiology.Scenarios по существу выполняет два этапа. Первый этап состоит в генерировании проб с использованием одного из вышеупомянутых методов выборки. Для lhs, sobol, и halton генерируемые однородные выборки преобразуются в выборки из указанного распределения с использованием функции обратного кумулятивного распределения icdf (Статистика и инструментарий машинного обучения). Затем, в качестве второго шага, выборки коррелируются с использованием алгоритма Имана-Коновера, если RankCorrelation указывается. Для randomвыборки извлекаются непосредственно из указанных распределений, и затем выборки коррелируются с использованием алгоритма Имана-Коновера.
Пример: 'SamplingMethod','lhs'
Expression - Комбинированное выражениеЭто свойство доступно только для чтения.
Комбинационное выражение, суммирующее комбинацию элементов в объекте, указанное как символьный вектор. Плюс + знак обозначает элементарное сочетание, а крест x знак указывает декартову комбинацию. Дополнительные сведения см. в разделе Объединение сценариев моделирования в SimBiology.
Пример:
'(k1 + k2 + k3) x doses'
Типы данных: char
NumberOfEntries - Количество записейЧисло записей в объекте сценариев, указанное как положительное целое число.
Пример:
4
Типы данных: double
RandomSeed - Начальное или состояние для генерации случайных чисел[] (по умолчанию) | неотрицательное целое число меньше 232 | структура, возвращенная rngНачальное число для генерации случайных чисел для получения воспроизводимых сценариев, указанное как неотрицательное целое число, меньшее, чем 232 или структура, возвращенная rng определяет случайное состояние. Значение по умолчанию [] означает, что сгенерированные сценарии будут отличаться каждый раз, когда generate вызывается функция, если вы не задаете случайное начальное число перед вызовом функции или не используете воспроизводимые последовательности, такие как Соболь или Халтон.
Пример:
10
Типы данных: double | struct
add | Добавление значений количества, доз или вариантов к SimBiology.Scenarios объект |
getEntry | Получить содержимое записи из SimBiology.Scenarios объект |
updateEntry | Обновить содержимое записи из SimBiology.Scenarios объект |
rename | Переименовать запись из SimBiology.Scenarios объект |
remove | Удалить записи из SimBiology.Scenarios объект |
verify | Проверить SimBiology.Scenarios объект |
generate | Создание сценариев из SimBiology.Scenarios объект и таблица возврата |
getNumberScenarios | Количество возвращаемых сценариев из SimBiology.Scenarios объект |
В этом примере используются:
Загрузить модель глюкозно-инсулиновой реакции. Для получения подробной информации о модели см. раздел «Фон» в разделе «Моделирование реакции глюкоза-инсулин».
sbioloadproject('insulindemo','m1');
Модель содержит различные значения параметров и исходные состояния, которые представляют различные нарушения инсулина (такие как диабет 2 типа, низкая чувствительность к инсулину и так далее), хранящиеся в пяти вариантах.
variants = getvariant(m1)
variants = SimBiology Variant Array Index: Name: Active: 1 Type 2 diabetic false 2 Low insulin se... false 3 High beta cell... false 4 Low beta cell ... false 5 High insulin s... false
Подавление информационного предупреждения, выдаваемого во время моделирования.
warnSettings = warning('off','SimBiology:DimAnalysisNotDone_MatlabFcn_Dimensionless');
Выберите дозу, которая представляет собой однократный прием 78 граммов глюкозы.
singleMeal = sbioselect(m1,'Name','Single Meal');
Создать Scenarios Изобретение относится к медицине и может быть использовано для представления различных исходных условий в сочетании с дозой. То есть создать scenario объект, где каждый вариант спарен (или скомбинирован) с дозой, в общей сложности для пяти сценариев моделирования.
sObj = SimBiology.Scenarios; add(sObj,'cartesian','variants',variants); add(sObj,'cartesian','dose',singleMeal)
ans =
Scenarios (5 scenarios)
Name Content Number
________ ___________________ ______
Entry 1 variants SimBiology variants 5
x Entry 2 dose SimBiology dose 1
See also Expression property.
sObj содержит две записи. Используйте generate для объединения записей и создания пяти сценариев. Функция возвращает таблицу сценариев, где каждая строка представляет сценарий, а каждый столбец представляет запись Scenarios объект.
scenariosTbl = generate(sObj)
scenariosTbl=5×2 table
variants dose
________________________ ___________________________
[1x1 SimBiology.Variant] [1x1 SimBiology.RepeatDose]
[1x1 SimBiology.Variant] [1x1 SimBiology.RepeatDose]
[1x1 SimBiology.Variant] [1x1 SimBiology.RepeatDose]
[1x1 SimBiology.Variant] [1x1 SimBiology.RepeatDose]
[1x1 SimBiology.Variant] [1x1 SimBiology.RepeatDose]
Измените имя первой записи.
rename(sObj,1,'Insulin Impairements')ans =
Scenarios (5 scenarios)
Name Content Number
____________________ ___________________ ______
Entry 1 Insulin Impairements SimBiology variants 5
x Entry 2 dose SimBiology dose 1
See also Expression property.
Создать SimFunction объект для моделирования созданных сценариев. Используйте Scenarios объект в качестве входных данных и определение концентраций глюкозы и инсулина в плазме в качестве ответных сигналов (выходные данные функции, которая должна быть нанесена на график). Определить [] для входного аргумента дозы с момента Scenarios объект уже имеет информацию о дозировке.
f = createSimFunction(m1,sObj,{'[Plasma Glu Conc]','[Plasma Ins Conc]'},[])f =
SimFunction
Parameters:
Name Value Type Units
____________________________ ______ _____________ ___________________________________________
{'Plasma Volume (Glu)' } 1.88 {'parameter'} {'deciliter' }
{'k1' } 0.065 {'parameter'} {'1/minute' }
{'k2' } 0.079 {'parameter'} {'1/minute' }
{'Plasma Volume (Ins)' } 0.05 {'parameter'} {'liter' }
{'m1' } 0.19 {'parameter'} {'1/minute' }
{'m2' } 0.484 {'parameter'} {'1/minute' }
{'m4' } 0.1936 {'parameter'} {'1/minute' }
{'m5' } 0.0304 {'parameter'} {'minute/picomole' }
{'m6' } 0.6469 {'parameter'} {'dimensionless' }
{'Hepatic Extraction' } 0.6 {'parameter'} {'dimensionless' }
{'kmax' } 0.0558 {'parameter'} {'1/minute' }
{'kmin' } 0.008 {'parameter'} {'1/minute' }
{'kabs' } 0.0568 {'parameter'} {'1/minute' }
{'kgri' } 0 {'parameter'} {'1/minute' }
{'f' } 0.9 {'parameter'} {'dimensionless' }
{'a' } 0 {'parameter'} {'1/milligram' }
{'b' } 0.82 {'parameter'} {'dimensionless' }
{'c' } 0 {'parameter'} {'1/milligram' }
{'d' } 0.01 {'parameter'} {'dimensionless' }
{'Stomach Glu After Dosing'} 78 {'parameter'} {'gram' }
{'kp1' } 2.7 {'parameter'} {'milligram/minute' }
{'kp2' } 0.0021 {'parameter'} {'1/minute' }
{'kp3' } 0.009 {'parameter'} {'(milligram/minute)/(picomole/liter)' }
{'kp4' } 0.0618 {'parameter'} {'(milligram/minute)/picomole' }
{'ki' } 0.0079 {'parameter'} {'1/minute' }
{'[Ins Ind Glu Util]' } 1 {'parameter'} {'milligram/minute' }
{'Vm0' } 2.5129 {'parameter'} {'milligram/minute' }
{'Vmx' } 0.047 {'parameter'} {'(milligram/minute)/(picomole/liter)' }
{'Km' } 225.59 {'parameter'} {'milligram' }
{'p2U' } 0.0331 {'parameter'} {'1/minute' }
{'K' } 2.28 {'parameter'} {'picomole/(milligram/deciliter)' }
{'alpha' } 0.05 {'parameter'} {'1/minute' }
{'beta' } 0.11 {'parameter'} {'(picomole/minute)/(milligram/deciliter)'}
{'gamma' } 0.5 {'parameter'} {'1/minute' }
{'ke1' } 0.0005 {'parameter'} {'1/minute' }
{'ke2' } 339 {'parameter'} {'milligram' }
{'Basal Plasma Glu Conc' } 91.76 {'parameter'} {'milligram/deciliter' }
{'Basal Plasma Ins Conc' } 25.49 {'parameter'} {'picomole/liter' }
Observables:
Name Type Units
_____________________ ___________ _______________________
{'[Plasma Glu Conc]'} {'species'} {'milligram/deciliter'}
{'[Plasma Ins Conc]'} {'species'} {'picomole/liter' }
Dosed:
TargetName TargetDimension
__________ _____________________
{'Dose'} {'Mass (e.g., gram)'}
Моделирование модели в течение 24 часов и печать данных моделирования. Данные содержат пять прогонов, где каждый прогон представляет сценарий в объекте «Сценарии».
sd = f(sObj,24); sbioplot(sd)

ans =
Axes (SbioPlot) with properties:
XLim: [0 30]
YLim: [0 450]
XScale: 'linear'
YScale: 'linear'
GridLineStyle: '-'
Position: [0.0951 0.1100 0.2521 0.8150]
Units: 'normalized'
Show all properties
При наличии Toolbox™ «Статистика» и «Машинное обучение» можно также получить выборочные значения для величин модели из различных вероятностных распределений. Например, предположим, что параметры Vmx и kp3, которые известны низкой и высокой чувствительностью к инсулину, следуют логнормальному распределению. Из такого распределения можно создать образцы значений для этих параметров и выполнить сканирование для изучения поведения модели.
Определите объект распределения логнормальных вероятностей для Vmx.
pd_Vmx = makedist('lognormal')pd_Vmx =
LognormalDistribution
Lognormal distribution
mu = 0
sigma = 1
По определению, параметр mu - среднее логарифмических значений. Для изменения значения параметра вокруг базового (модельного) значения параметра установите mu кому log(model_value). Установите стандартное отклонение (сигма) равным 0,2. Для малого значения сигмы среднее логнормальное распределение приблизительно равно log(model_value). Дополнительные сведения см. в разделе Lognormal Distribution (Statistics and Machine Learning Toolbox).
Vmx = sbioselect(m1,'Name','Vmx'); pd_Vmx.mu = log(Vmx.Value); pd_Vmx.sigma = 0.2
pd_Vmx =
LognormalDistribution
Lognormal distribution
mu = -3.05761
sigma = 0.2
Аналогично определите распределение вероятности для kp3.
pd_kp3 = makedist('lognormal'); kp3 = sbioselect(m1,'Name','kp3'); pd_kp3.mu = log(kp3.Value); pd_kp3.sigma = 0.2
pd_kp3 =
LognormalDistribution
Lognormal distribution
mu = -4.71053
sigma = 0.2
Теперь определите совместное распределение вероятности, чтобы получить значения выборки для Vmx и kp3, с ранговой корреляцией, чтобы указать некоторую корреляцию между этими двумя параметрами. Следует отметить, что это допущение корреляции предназначено только для иллюстрации данного примера и может не быть биологически значимым.
Сначала удалите запись вариантов (запись 1) из sObj.
remove(sObj,1)
ans =
Scenarios (1 scenarios)
Name Content Number
____ _______________ ______
Entry 1 dose SimBiology dose 1
See also Expression property.
Добавьте запись, определяющую совместное распределение вероятности с помощью матрицы ранговой корреляции.
add(sObj,'cartesian',["Vmx","kp3"],[pd_Vmx, pd_kp3],'RankCorrelation',[1,0.5;0.5,1])
ans =
Scenarios (2 scenarios)
Name Content Number
____ ______________________ ___________
Entry 1 dose SimBiology dose 1
x (Entry 2.1 Vmx Lognormal distribution 2 (default)
+ Entry 2.2) kp3 Lognormal distribution 2 (default)
See also Expression property.
По умолчанию количество образцов для извлечения из распределения соединений устанавливается равным 2. Увеличьте количество образцов.
updateEntry(sObj,2,'Number',50)ans =
Scenarios (50 scenarios)
Name Content Number
____ ______________________ ______
Entry 1 dose SimBiology dose 1
x (Entry 2.1 Vmx Lognormal distribution 50
+ Entry 2.2) kp3 Lognormal distribution 50
See also Expression property.
Убедитесь, что Scenarios объект может быть смоделирован с помощью модели. verify функция выдает ошибку, если какая-либо запись не имеет уникального разрешения для объекта в модели, или содержимое записи имеет противоречивую длину (размеры выборки). Функция выдает предупреждение, если несколько записей разрешаются для одного и того же объекта в модели.
verify(sObj,m1)
Создайте сценарии моделирования. Постройте график значений образца с помощью plotmatrix. Вы можете видеть значение Vmx варьируется вокруг значения его модели 0,047 и значения kp3 около 0,009.
sTbl = generate(sObj); [s,ax,bigax,h,hax] = plotmatrix([sTbl.Vmx,sTbl.kp3]); ax(1,1).YLabel.String = "Vmx"; ax(2,1).YLabel.String = "kp3"; ax(2,1).XLabel.String = "Vmx"; ax(2,2).XLabel.String = "kp3";

Смоделировать сценарии с использованием той же SimFunction, что была создана ранее. Нет необходимости создавать новый объект SimFunction, даже если он был обновлен.
sd2 = f(sObj,24); sbioplot(sd2);

По умолчанию SimBiology использует метод случайной выборки. Вы можете изменить его на латинский гиперкуб (или соболь или халтон) для более систематического подхода заполнения пространства.
entry2struct = getEntry(sObj,2)
entry2struct = struct with fields:
Name: {'Vmx' 'kp3'}
Content: [2x1 prob.LognormalDistribution]
Number: 50
RankCorrelation: [2x2 double]
Covariance: []
SamplingMethod: 'random'
entry2struct.SamplingMethod = 'lhs'entry2struct = struct with fields:
Name: {'Vmx' 'kp3'}
Content: [2x1 prob.LognormalDistribution]
Number: 50
RankCorrelation: [2x2 double]
Covariance: []
SamplingMethod: 'lhs'
Теперь можно использовать обновленную структуру для изменения записи 2.
updateEntry(sObj,2,entry2struct)
ans =
Scenarios (50 scenarios)
Name Content Number
____ ______________________ ______
Entry 1 dose SimBiology dose 1
x (Entry 2.1 Vmx Lognormal distribution 50
+ Entry 2.2) kp3 Lognormal distribution 50
See also Expression property.
Визуализируйте значения образца.
sTbl2 = generate(sObj); [s,ax,bigax,h,hax] = plotmatrix([sTbl2.Vmx,sTbl2.kp3]); ax(1,1).YLabel.String = "Vmx"; ax(2,1).YLabel.String = "kp3"; ax(2,1).XLabel.String = "Vmx"; ax(2,2).XLabel.String = "kp3";

Смоделировать сценарии.
sd3 = f(sObj,24); sbioplot(sd3);

Восстановить параметры предупреждения.
warning(warnSettings);
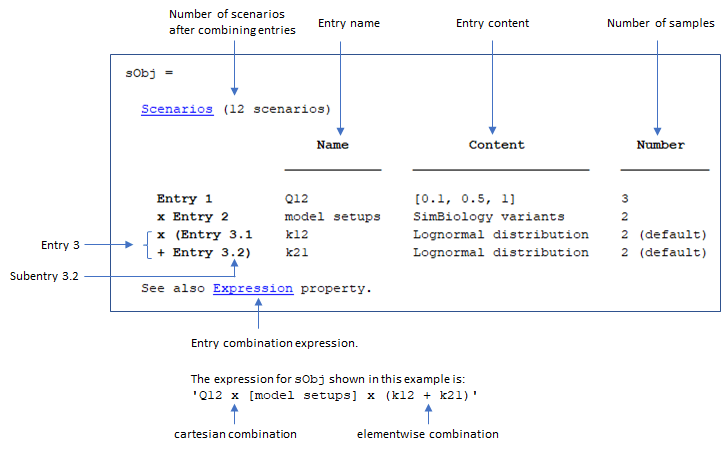
SimBiology.Scenarios ТерминологияЭтот раздел аннотирует отображение командной строки SimBiology.Scenarios и объясняет термины, показанные в выходных данных. В частности, она объясняет эти терминологии: Scenarios, Entry, Subentry, Name, Content, Number, Expression, inconsistent и Diagnosis.
Последовательное
Scenarios объект имеет записи, которые имеют правильное количество выборок, так что записи могут быть объединены без ошибок. Пример непротиворечивого Scenarios объект отображается следующим.
Несогласованный объект Scriptions имеет одну или несколько записей с неверным количеством образцов. Эти записи необходимо исправить перед использованием объекта для моделирования. Далее показан пример несогласованного объекта.

Diagnosis в столбце указывается, какие записи следует исправить для правильного количества образцов. Использовать updateEntry, rename, и remove для редактирования записей.
[1] Иман, Р. и У. Джей Коновер. 1982. Подход без распределения к индукции ранговой корреляции между входными переменными. Связь в статистике - моделирование и вычисление. 11(3):311–334.
Имеется измененная версия этого примера. Открыть этот пример с помощью изменений?
1. Если смысл перевода понятен, то лучше оставьте как есть и не придирайтесь к словам, синонимам и тому подобному. О вкусах не спорим.
2. Не дополняйте перевод комментариями “от себя”. В исправлении не должно появляться дополнительных смыслов и комментариев, отсутствующих в оригинале. Такие правки не получится интегрировать в алгоритме автоматического перевода.
3. Сохраняйте структуру оригинального текста - например, не разбивайте одно предложение на два.
4. Не имеет смысла однотипное исправление перевода какого-то термина во всех предложениях. Исправляйте только в одном месте. Когда Вашу правку одобрят, это исправление будет алгоритмически распространено и на другие части документации.
5. По иным вопросам, например если надо исправить заблокированное для перевода слово, обратитесь к редакторам через форму технической поддержки.