Класс: geneont
Найдите термины, которые являются предками заданного термина Gene Ontology (GO)
AncestorIDs = getancestors(GeneontObj, ID)
[AncestorIDs, Counts]
= getancestors(GeneontObj, ID)
... = getancestors(..., 'Height', HeightValue,
...)
... = getancestors(..., 'Relationtype', RelationtypeValue,
...)
... = getancestors(..., 'Exclude', ExcludeValue,
...)
AncestorIDs = getancestors(GeneontObj, ID)GeneontObj, объект geneont, для терминов GO, которые являются предками терминов GO, заданных ID, который является идентификатором термина GO или вектором идентификаторов. Возвращается AncestorIDsвектор идентификаторов терминов GO, включая ID. ID является неотрицательным целым числом или вектором, содержащим неотрицательные целые числа.
[ также возвращает количество раз, когда каждый предок найден. AncestorIDs, Counts]
= getancestors(GeneontObj, ID)Counts - вектор-столбец с таким же количеством элементов, как и условия в GeneontObj.
Совет
The Counts возвращаемое значение полезно, когда вы подсчитываете в исследованиях обогащения генов. Для получения дополнительной информации см. Gene Ontology Enrichment in Microarray Data.
... = getancestors (..., вызывает 'PropertyName', PropertyValue, ...)getancestors с необязательными свойствами, которые используют пары имя/значение свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должны быть заключены в одинарные кавычки и нечувствительны к регистру. Эти имена свойства/пары значения свойств следующие:
... = getancestors(..., 'Height', поиск по заданному количеству уровней, HeightValue,
...)HeightValue, в онтологии генов. HeightValue является положительным целым числом. По умолчанию это Inf.
... = getancestors(..., 'Relationtype', поиск заданных типов отношений, RelationtypeValue,
...)RelationtypeValue, в онтологии генов. RelationtypeValue является вектор символов. Варианты 'is_a', 'part_of', или 'both' (по умолчанию).
... = getancestors(..., 'Exclude', управляет, исключая ExcludeValue,
...)ID, исходный запрашиваемый термин (ы) из выхода AncestorIDs, если только термин не был достигнут при поиске генной онтологии. Варианты true или false (по умолчанию).
GeneontObj | Объект генеонта, такой как созданный geneont функция конструктора. |
ID | Идентификатор термина GO или вектор идентификаторов. |
HeightValue | Положительное целое число, определяющее количество уровней для поиска вверх в онтологии гена. |
RelationtypeValue | Вектор символов, задающий типы отношений для поиска в онтологии генов. Варианты:
|
ExcludeValue | Управляет, исключая ID, исходный запрашиваемый термин (ы) из выхода AncestorIDs, если только термин не был достигнут при поиске генной онтологии. Варианты true или false (по умолчанию). |
AncestorIDs | Вектор идентификаторов терминов GO, включая ID. |
Counts | Вектор-столбец с таким же количеством элементов, как и термины в GeneontObj, что указывает на количество раз, когда каждый предок обнаруживается. |
Загрузите текущую версию базы данных Gene Ontology из Web в объект генеонта в MATLAB® программное обеспечение.
GO = geneont('LIVE', true)MATLAB создает объект geneont и отображает количество терминов в базе данных.
Gene Ontology object with 24316 Terms.
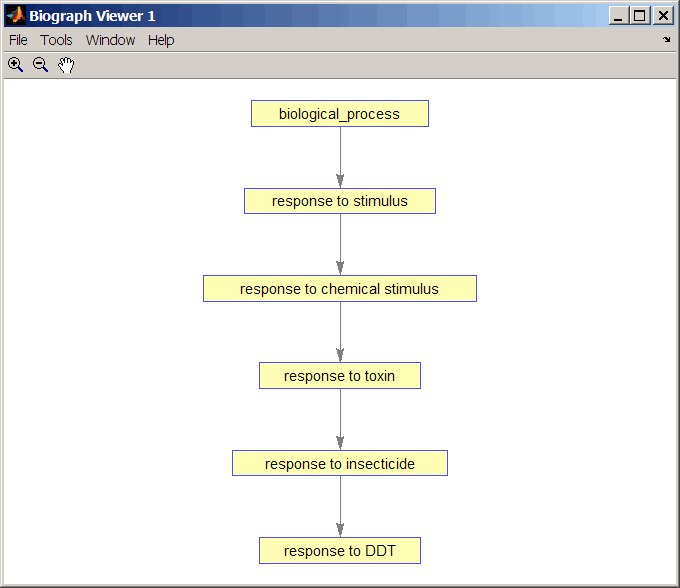
Найдите предков термина Gene Ontology с идентификатором 46680.
ancestors = getancestors(GO,46680)
ancestors =
8150
9636
17085
42221
46680
50896Создайте подчиненную онтологию генов.
subontology = GO(ancestors) Gene Ontology object with 6 Terms.
Создайте и отобразите отчет о подчиненных терминах Gene Ontology, который включает идентификатор и имя GO.
rpt = get(subontology.terms,{'id','name'})
[ 8150] 'biological_process'
[ 9636] 'response to toxin'
[17085] [1x23 char]
[42221] [1x29 char]
[46680] 'response to DDT'
[50896] [1x20 char]
Просмотрите отношения подчиненной онтологии генов при помощи getmatrix метод создания матрицы соединений для передачи в biograph функция.
cm = getmatrix(subontology); BG = biograph(cm, get(subontology.terms, 'name')); view(BG)
goannotread | num2goid | term