Преобразуйте структуру mzCDF в пиковый список
[Peaklist, Times]
= mzcdf2peaks(mzCDFStruct)
mzCDFStruct | MATLAB® структура, содержащая информацию из файла netCDF, например, созданную |
Peaklist | Одно из следующих:
|
Times | Скаляр вектора времени удерживания, сопоставленный с набором данных по жидкостной хроматографии/масс-спектрометрии (LC/MS) или газовой хроматографии/масс-спектрометрии (GC/MS). Если |
[ извлекает пиковую информацию из Peaklist, Times]
= mzcdf2peaks(mzCDFStruct)mzCDFStruct, структуру MATLAB, содержащую информацию из файла netCDF, например, созданную mzcdfread function, и создает Peaklist, одну матрицу или массив ячеек матриц, содержащих значения массы/заряда (m/z) и значения интенсивности ионов, и Timesскаляр или вектор времени удерживания, сопоставленный с набором данных жидкостной хроматографии/масс-спектрометрии (LC/MS) или газовой хроматографии/масс-спектрометрии (GC/MS).
mzCDFStruct содержит поля, которые соответствуют переменным и глобальным атрибутам в файле netCDF. Если переменная netCDF содержит локальные атрибуты, создается дополнительное поле с именем поля, содержащим имя переменной _attributes. Количество и имена полей будут варьироваться в зависимости от программного обеспечения масс-спектрометра, но обычно существуют mass_values и intensity_values поля.
В следующем примере файл results.cdf не предусмотрен.
Используйте mzcdfread функция для чтения файла netCDF в программное обеспечение MATLAB в качестве структуры. Затем извлеките пиковую информацию из структуры.
mzcdf_struct = mzcdfread('results.cdf');
[peaks,time] = mzcdf2peaks(mzcdf_struct)
peaks =
[7008x2 single]
[7008x2 single]
[7008x2 single]
[7008x2 single]
time =
8.3430
12.6130
16.8830
21.1530
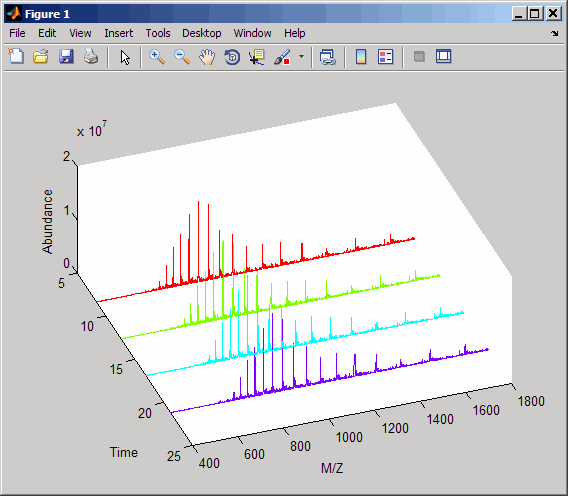
Создайте карту цветов, содержащую цвет для каждого пикового списка (время хранения).
colors = hsv(numel(peaks));
Создайте 3-D рисунок peaks и добавьте к ней метки.
figure
hold on
for i = 1:numel(peaks)
t = repmat(time(i),size(peaks{i},1),1);
plot3(t,peaks{i}(:,1),peaks{i}(:,2),'color',colors(i,:))
end
view(70,60)
xlabel('Time')
ylabel(mzcdf_struct.mass_axis_label)
zlabel(mzcdf_struct.intensity_axis_label)