Визуализируйте межмолекулярные расстояния в файле Protein Data Bank (PDB)
pdbdistplot(PDBid)
pdbdistplot(PDBid, Distance)
pdbdistplot(___,'Chain',ChainID)
pdbdistplot(___,'Model',ModelNum)
pdbdistplot(___,'Hetero',TF)
PDBid | Любое из следующих:
Примечание Каждая структура в базе данных PDB представлена четырехсимвольным алфавитно-цифровым идентификатором. Для примера, |
Distance | Пороговое расстояние в ангстремах показано на шпионском графике. По умолчанию это |
ChainID | Любое из следующих:
|
ModelNum | Положительное целое число, определяющее, какую модель учитывать. Значение по умолчанию является 1. |
pdbdistplot отображает расстояния между атомами и между остатками в структуре PDB.
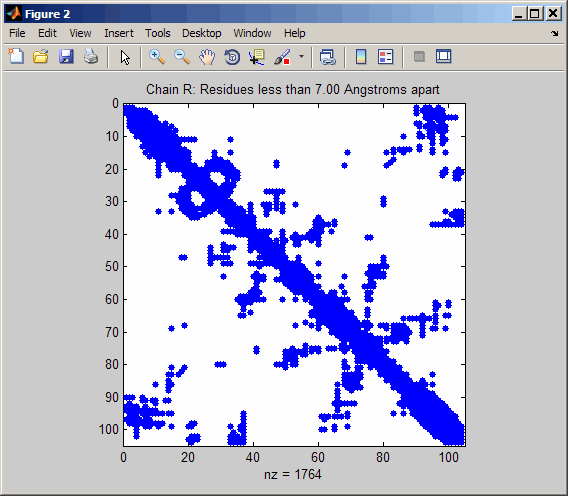
pdbdistplot( извлекает структуру, заданную в PDBid)PDBid из базы данных PDB и создает тепловую карту, показывающую расстояния между остатками и шпионский график, показывающий остатки, где минимальные расстояния друг от друга меньше 7 ангстремы. Если в PDBid присутствуют несколько цепей, создаются отдельные графики.
pdbdistplot( задает пороговое расстояние, показанное на шпионском графике. По умолчанию это PDBid, Distance)7.
pdbdistplot(___,'Chain', задает учитываемые цепи. По умолчанию рассматриваются все цепи, включенные в модель.ChainID)
pdbdistplot(___,'Model', определяет, какую структурную модель PDB следует учитывать. Значение по умолчанию является 1.ModelNum)
pdbdistplot(___,'Hetero', определяет, включать ли гетероатомы в график взаимодействий остатков. TF)TF является логическим, true или false. По умолчанию это false.
Отобразите тепловую карту расстояний между остатками и шпионский график на 7 ангстремы белкового цитохрома С из альбакорного тунца.
pdbdistplot('5CYT');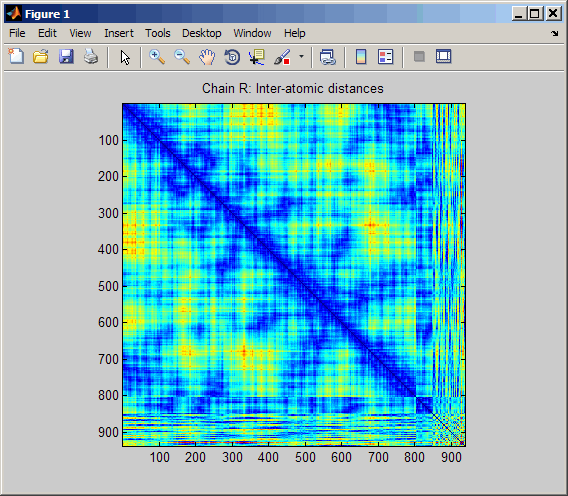
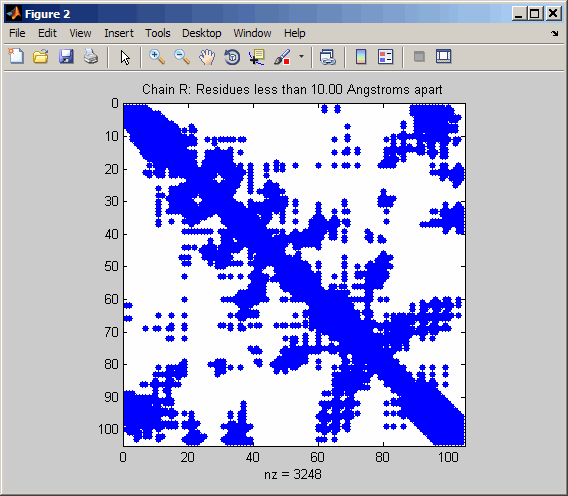
Отобразите шпионский график в 10 ангстремы той же структуры.
pdbdistplot('5CYT',10);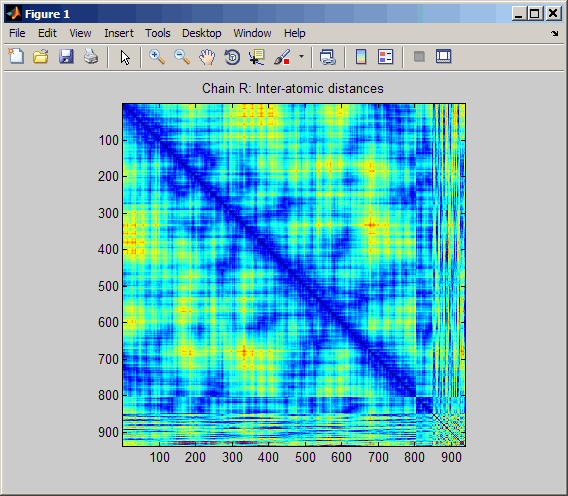
getpdb | molviewer | pdbread | proteinplot | ramachandran