Постройте график Рамачандрана для данных Protein Data Bank (PDB)
ramachandran(PDBid)
ramachandran(File)
ramachandran(PDBStruct)
RamaStruct = ramachandran(...)
ramachandran(..., 'Chain', ChainValue,
...)
ramachandran(..., 'Plot', PlotValue,
...)
ramachandran(..., 'Model', ModelValue,
...)
ramachandran(..., 'Glycine', GlycineValue,
...)
ramachandran(..., 'Regions', RegionsValue,
...)
ramachandran(..., 'RegionDef', RegionDefValue,
...)
PDBid | Вектор символов или строка, задающая уникальный идентификатор для записи структуры белка в базе данных PDB. Примечание Каждая структура в базе данных PDB представлена четырехсимвольным алфавитно-цифровым идентификатором. Для примера, |
File | Вектор символов или строка, указывающая имя файла или путь и имя файла. Файл-ссылка является файлом банка данных белка (PDB) в формате. Если вы задаете только имя файла, этот файл должен быть в MATLAB® путь поиска файлов или в текущей директории MATLAB. |
PDBStruct | Структура MATLAB, содержащая PDB-форматированные данные, такие как возвращенные getpdb или pdbread. |
ChainValue | Вектор символов, строка, строковый вектор или массив ячеек из векторов символов, который задает цепи (цепи ) для вычисления углов кручения и построения графика . Варианты:
|
PlotValue | Вектор символов или строка, задающая способ построения цепей. Варианты:
|
ModelValue | Целое число, которое задает учитываемую модель структуры. По умолчанию это 1. |
GlycineValue | Управляет подсветкой остатков глицина кругом на графике. Варианты true или false (по умолчанию). |
RegionsValue | Управление чертежом ссылочных областей Рамачандран на графике. Варианты Областями по умолчанию являются ядро, правая альфа, бета ядро, левая альфа, и разрешено, с основными областями, соответствующими точкам данных предпочтительных значений пар psi/phi угла, и разрешенными областями, соответствующими возможным, но неблагополучным значениям пар psi/phi угла, на основе простых энергетических факторов. Контуры этих областей по умолчанию основаны на расчетах Morris et al., 1992. Примечание При использовании палитры по умолчанию red = right-hand core alpha, core beta и core left-hand alpha, тогда как yellow = allowed. |
RegionDefValue | Структура MATLAB или массив структур (если задано несколько областей), содержащий информацию (имя, цвет и контуры) для пользовательских ссылочных областей на графике Рамачандрана. Каждая структура должна содержать следующие поля:
Совет Если вы задаете пользовательские опорные области, в которых меньшая область содержится или покрыта большей областью, сначала перечислите структуру для меньшей области в массиве, чтобы она была нанесена последней и видимой на графике. |
RamaStruct | Структура MATLAB или массив структур (если белок содержит несколько цепей). Каждая структура содержит следующие поля:
Описание полей см. в следующей таблице. |
График Ramachandran - график угла скрученности phi, Φ, (угол скрученности между C-N-CA-C атомы) против скрученности поворачивают psi, Ψ, (угол скрученности между N-CA-C-N атомы) для каждого остатка белковой последовательности.
ramachandran( генерирует график Рамачандрана для белка, заданного идентификатором базы данных PDB PDBid)PDBid.
ramachandran( генерирует график Рамачандрана для белка, заданного File)File, файл в формате PDB.
ramachandran( генерирует график Рамачандрана для белка, хранящегося в PDBStruct)PDBStruct, структуру MATLAB, содержащую PDB-форматированные данные, такие как возвращенные getpdb или pdbread.
RamaStruct = ramachandran(...)
| Область | Описание |
|---|---|
Angles | Матрица с тремя столбцами, содержащая скрученность, поворачивает phi (Φ), psi (Ψ), и омега (ω) для каждого остатка в последовательности, заказанной порядковым номером остатка. Количество строк в матрице равно количеству строк в Примечание The |
ResidueNum | Вектор-столбец, содержащий порядковые номера остатков из файла PDB. Примечание The
The Углы, перечисленные в |
ResidueName | Векторы-столбцы, содержащие имена остатков белка. |
Chain | Вектор символов или строка, задающая цепи в белке. |
HPoints | Указатель на точки данных на графике. |
рамачандран (..., вызывает 'PropertyName', PropertyValue, ...)ramachandran с необязательными свойствами, которые используют пары имя/значение свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должны быть заключены в одинарные кавычки и нечувствительны к регистру. Эти имена свойства/пары значения свойств следующие:
ramachandran(..., 'Chain', задает цепи (цепи ) для вычисления углов кручения и построения графиков. Варианты:ChainValue,
...)
'All' (по умолчанию) - Вычисляются и строятся углы кручения для всех цепей.
Вектор символов или строка, задающая идентификатор цепи, который чувствителен к регистру.
Массив ячеек из векторов символов или строкового вектора, задающий идентификаторы цепей, которые чувствительны к регистру.
ramachandran(..., 'Plot', задает способ построения графиков цепей. Варианты:PlotValue,
...)
'None' - Ничего не строит.
'Separate' - Строит графики углов кручения для всех указанных цепей на отдельных графиках.
'Combined' (по умолчанию) - строит графики углов кручения для всех заданных цепей на одном комбинированном графике.
ramachandran(..., 'Model', задает учитываемую модель структуры. По умолчанию это ModelValue,
...)1.
ramachandran(..., 'Glycine', управляет подсветкой остатков глицина кругом на графике. Варианты GlycineValue,
...)true или false (по умолчанию).
ramachandran(..., 'Regions', управляет чертежом ссылочных областей Рамачандрана на графике. Варианты RegionsValue,
...)true или false (по умолчанию).
Областями по умолчанию являются ядро, правая альфа, бета ядро, левая альфа, и разрешено, с основными областями, соответствующими точкам данных предпочтительных значений пар psi/phi угла, и разрешенными областями, соответствующими возможным, но неблагополучным значениям пар psi/phi угла, на основе простых энергетических факторов. Контуры этих областей по умолчанию основаны на расчетах Morris et al., 1992.
Примечание
Если используется палитра по умолчанию, то red = core right-hand alpha, core beta и core left-hand alpha, тогда как yellow = allowed.
ramachandran(..., 'RegionDef', задает информацию (имя, цвет и контур) для пользовательских ссылочных областей на графике Рамачандрана. RegionDefValue,
...)RegionDefValue - структура MATLAB или массив структур, содержащий следующие поля:
Name - Вектор символов или строка, задающая имя для области.
Color - Символьный вектор или строка или трехэлементный числовой вектор значений RGB, задающий цвет для области на графике.
Patch - матрица 2-by-N значений, первая строка, содержащая значения угла кручения phi (,) и вторая строка, содержащая значения угла кручения psi (,). Когда строятся пары углов psi/phi, точки данных задают контур для области. N - количество точек данных, необходимых для определения области.
Совет
Если вы задаете пользовательские опорные области, в которых меньшая область содержится или покрыта большей областью, сначала перечислите структуру для меньшей области в массиве, чтобы она была нанесена последней и видимой на графике.
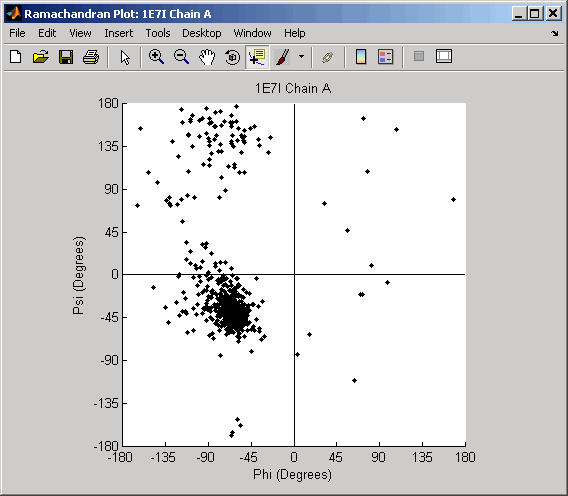
Нарисуйте график Рамачандрана для альбумина сыворотки человека, комплексного с октадекановой кислотой, которая имеет идентификатор базы данных PDB 1E7I.
ramachandran('1E7I')
Используйте getpdb функция для извлечения данных о структуре белка для гормона роста человека из базы данных PDB и сохранения информации в файл.
getpdb('1a22','ToFile','1a22.pdb');Вычислите углы кручения и нарисуйте график Рамачандрана для цепи А гормона роста человека, представленного в файле PDB 1a22.pdb.
ChainA1a22Struct = ramachandran('1a22.pdb','chain','A')
ChainA1a22Struct =
Angles: [191x3 double]
ResidueNum: [191x1 double]
ResidueName: {191x1 cell}
Chain: 'A'
HPoints: 370.0012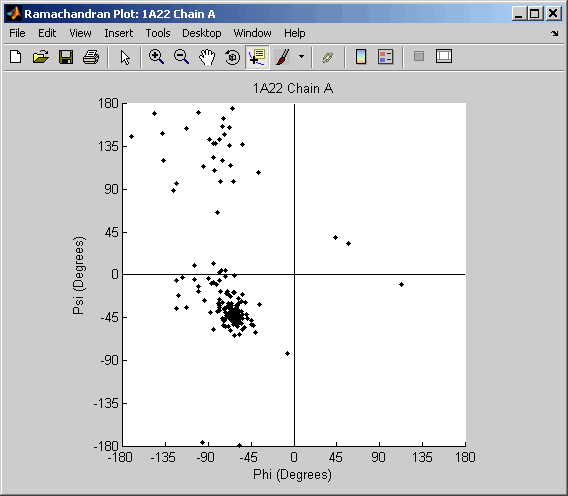
Используйте getpdb функция для извлечения данных о структуре белка для гормона роста человека из базы данных PDB и хранения информации в структуре.
Struct1a22 = getpdb('1a22');Нарисуйте комбинированный график Рамачандрана для всех цепей гормона роста человека, представленного в структуре PDB, 1a22Struct. Выделите глициновые остатки (с кругом) и нарисуйте ссылочные области Рамачандрана на графике.
ramachandran(Struct1a22,'glycine',true,'regions',true);
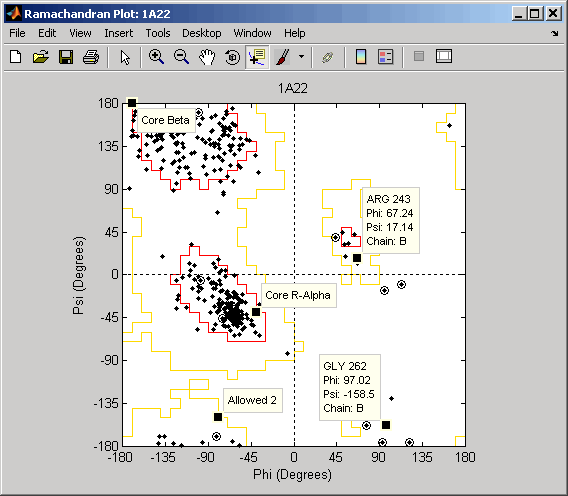
Совет
Щелкните точку данных, чтобы отобразить всплывающую подсказку с информацией об остатке. Щелкните область, чтобы отобразить всплывающую подсказку, определяющую область. Нажмите и удерживайте клавишу Alt, чтобы отобразить несколько всплывающих подсказок.
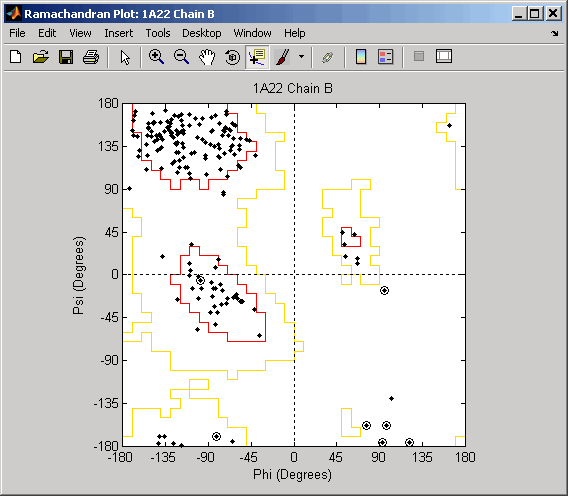
Нарисуйте отдельный график Рамачандрана для каждой цепи гормона роста человека, представленного в структуре PDB, 1a22Struct. Выделите глициновые остатки (с кругом) и нарисуйте ссылочные области Рамачандрана на графике.
ramachandran(Struct1a22,'plot','separate','chain','all',...
'glycine',true,'regions',true)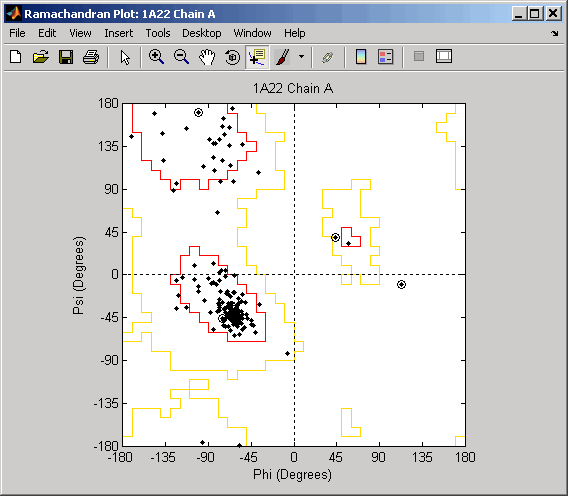
Создайте массив из двух структур, содержащих углы кручения для цепей A и D в кальций/кальмодулин-зависимой протеинкиназе, которая имеет идентификатор базы данных PDB 1hkx.
a = ramachandran('1hkx', 'chain', {'A', 'D'})
a =
1x2 struct array with fields:
Angles
ResidueNum
ResidueName
Chain
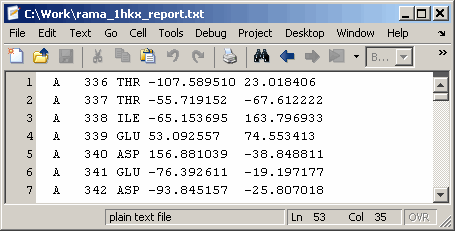
HPointsЗапишите файл отчета с разделителем табуляций, содержащий углы кручения phi (,) и psi (,) для цепей A и D в кальций/кальмодулин-зависимой протеинкиназе.
fid = fopen('rama_1hkx_report.txt', 'wt');
for c = 1:numel(a)
for i = 1:length(a(c).Angles)
if ~all(isnan(a(c).Angles(i,:)))
fprintf(fid,'%s\t%d\t%s\t%f\t%f\n', a(c).Chain, ...
a(c).ResidueNum(i), a(c).ResidueName{i}, ...
a(c).Angles(i,1:2));
end
end
end
fclose(fid);
Просмотрите файл, созданный в редакторе MATLAB.
edit rama_1hkx_report.txt
[1] Morris, A.L., MacArthur, M.W., Hutchinson, E. and Thornton, J.M. (1992). Стереохимическое качество координат структуры белка. БЕЛКИ: Структура, функция и генетика 12, 345-364.
getpdb | molviewer | pdbdistplot | pdbread | proteinpropplot