Этот пример показывает, как добавить ингибирование SGLT2 гипотетическим соединением к существующей модели глюкозо-инсулина с использованием SimBiology Model Builder.
Эта модель является другой реализацией SimBiology модели глюкозо-инсулина, упомянутой в примере Симуляции ответа глюкозо-инсулина. Модель основана на публикации Dalla Man, et al. В своей публикации 2007 года [1] авторы разработали модель человеческого глюкозо-инсулинового ответа после еды. Эта модель описывает динамику системы, используя обыкновенные дифференциальные уравнения. Авторы использовали свою модель для моделирования ответа глюкозо-инсулина после одного или нескольких приемов пищи, для нормальных людей и для людей с различного рода нарушениями инсулина.
Показано, что рецептор SGLT2 облегчает около 50% реабсорбции глюкозы в почках [3]. Этот пример принимает, что имеет гипотетическое соединение ингибитора SGLT2, которое ингибирует SGLT2 на 50%. Также приняты разумный режим дозирования и свойства PK. В этом примере вы включаете фармакокинетику/фармакодинамику (PK/PD) этого ингибиторного соединения в модель глюкозо-инсулина.
На следующих стадиях вы моделируете абсорбцию соединения и клиренс гипотетического соединения ингибитора SGLT2 с помощью двух реакций.
Откройте приложение SimBiology Model Builder, нажав SimBiology Model Builder на вкладке Apps или введя simBiologyModelBuilder в командной строке.
На вкладке Home приложения выберите Open.
Перейдите к папке matlabroot\examples\simbio\data\. matlabroot - папка, в которой установлен MATLAB. Вход matlabroot в командной строке указывается путь к корневой папке. Выберите файл проекта с именем SGLT2_model_incomplete.sbproj. Нажмите Open.
Примечание
В macOS вместо Ctrl используйте клавишу command.
Перетащите два блока видов с панели инструментов вкладки Diagram.
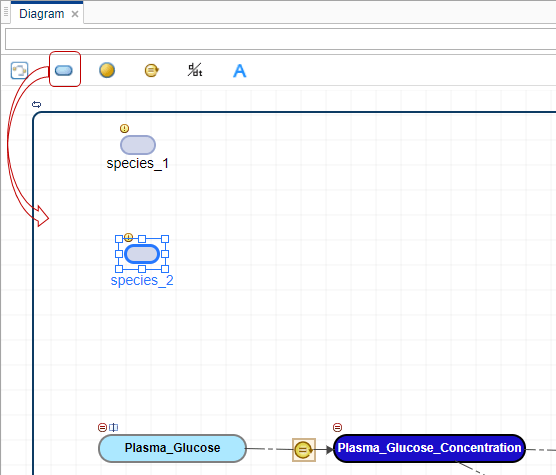
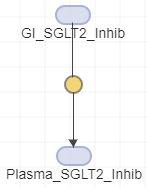
Нажмите Ctrl и перетащите линию от первого вида ко второму виду. Между ними появляется реакционный блок. Эта реакция представляет собой абсорбцию соединения.
Отредактируйте имя вида по умолчанию двойным щелчком мыши. Переименовать species_1 на GI_SGLT2_Inhib и species_2 на Plasma_SGLT2_Inhib.
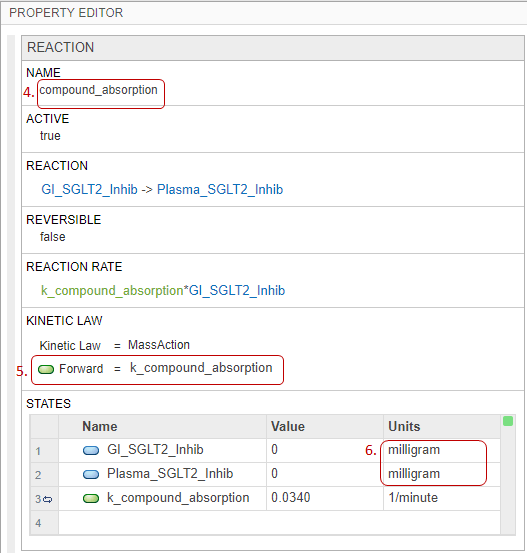
Щелкните блок реакции. На панели Property Editor справа измените Name реакции на compound_absorption.
В разделе Kinetic Law измените автоматическую обработку параметра скорости Forward kf на k_compound_absorption. Модель уже имеет параметр прямой скорости k_compound_absorption который был создан ранее. Приложение использует зеленый текст для имен параметров и синий текст для названий видов в выражении скорости реакции.
Совет
Чтобы изменить строения реакции по умолчанию, щелкните Preferences на вкладке Home. В диалоговом окне настроек нажмите Model Building. В Reaction Building разделе существуют три опции изменения кинетического закона по умолчанию, создания параметров кинетического закона и изменения возможностей созданных параметров.
В States таблице установите модули двух видов равными milligram.
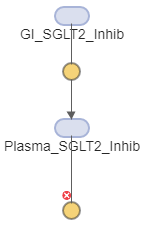
Перетащите другой блок реакции с панели инструментов схемы, чтобы смоделировать составной зазор.
Нажмите Ctrl и перетащите линию из Plasma_SGLT2_Inhib на реакцию.
Совет
Если блоки не выровнены, их можно выровнять с помощью инструментов выравнивания. На вкладке Home выберите Diagram Tools > Diagram Alignment Tools.
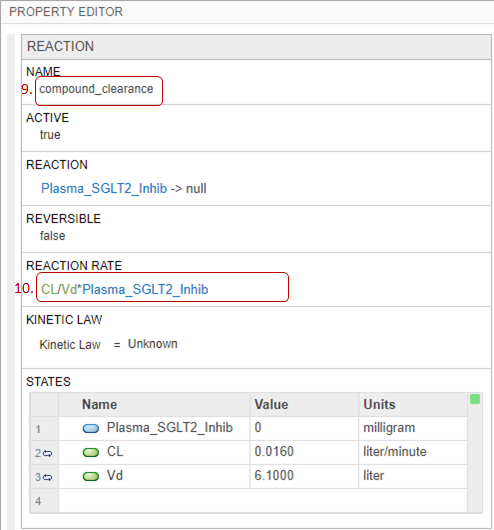
Щелкните блок реакции. На панели Property Editor измените Name реакции на compound_clearance.
Обновите Reaction Rate на CL/Vd*Plasma_SGLT2_Inhib. CL и Vd указывают параметры модели для зазора и объема распределения, соответственно.
Совет
Кинетический закон для вновь добавленной реакции сконфигурирован таким образом, чтобы он MassAction по умолчанию, и приложение SimBiology Model Builder автоматически создает и отображает виды и параметры, необходимые для скорости реакции. Для других кинетических законов создаются и отображаются только параметры. Вам нужно создать и сопоставить вид вручную. Используйте Unknown кинетический закон, чтобы задать пользовательскую скорость реакции с ее собственными параметрами. Вы должны задать и добавить виды и параметры, необходимые для пользовательской скорости.
MassAction и Unknown кинетические законы могут иметь различные результаты симуляции, даже когда скорость реакции одинаковая. Это может произойти, когда у вас есть обратимая реакция с видами в разных отсеках. Различие в результаты симуляции обусловлено масштабированием объема, выполненным SimBiology во время размерного анализа. Для получения дополнительной информации смотрите Вывод ОДУ из реакций. В частности, для MassAction SimBiology использует соответствующие объемы отсеков, чтобы умножить прямую и обратную скорости. Однако для Unknown и других встроенных кинетических законов SimBiology умножает всю скорость только на один отсек, который содержит реагенты. Чтобы увидеть, какие именно объемы отсеков используются для масштабирования, откройте вкладку Уравнения и проверьте раздел ODEs.
SimBiology позволяет вам задать математическое выражение, чтобы задать или обновить значение величины модели во время симуляции. Для получения дополнительной информации смотрите Определения и оценки правил в SimBiology моделях. На следующих стадиях вы добавляете повторное правило назначения для включения фармакодинамики ингибитора путем определения почечного порога, при котором глюкоза в плазме выделяется на основе эффективности соединения.
На панели Browser щелкните значок плюс на панели инструментов браузера и выберите Add Repeated Assignment. Приложение перемещает особое внимание в последнюю пустую строку в таблице Repeated Assignments.
Дважды кликните строку и введите следующее выражение, которое представляет ингибирование соединения на основе уравнения Хилла:
renal_threshold = basal_renal_threshold*(1-compound_Imax*Plasma_SGLT2_Inhib^2/(compound_IC50^2+Plasma_SGLT2_Inhib^2))
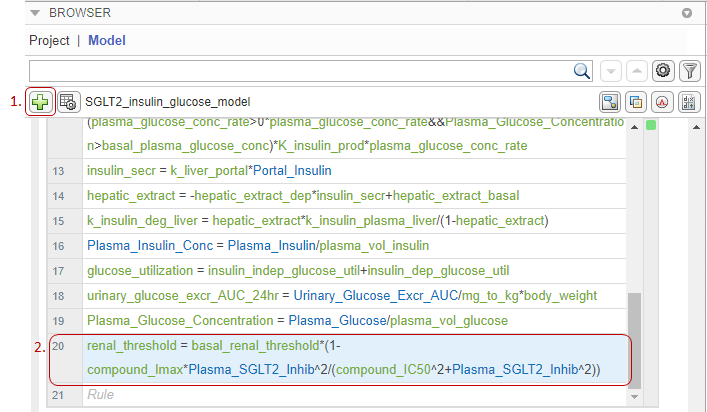
На вкладке Diagram теперь показан повторный блок правила назначения для renal_threshold параметр.
Совет
Чтобы просмотреть всю модель и панорамировать ее, разверните Model Assessment Tools на панели Browser и нажмите Overview.
Примечание
Приложение показывает только блок параметра для параметра, который находится на левой стороне (LHS) повторного правила назначения, правила скорости или функции события.
Приложение показывает только блоки правил для повторных назначений и правил скорости.
Приложение используют линии, чтобы соединить величины на правой стороне правила. По умолчанию эти линии не показаны. Чтобы отобразить линии, щелкните блок правил. На панели Property Editor в разделе Block установите для Expression Lines значение show.
Реакция почечной экскреции модели в настоящее время определяется как Plasma_Glucose -> Urinary_Glucose_Excr_AUC с параметром скорости реакции glucose_excretion. Параметр скорости определяется повторным правилом назначения следующим glucose_excretion = (Plasma_Glucose>renal_threshold)*GFR*(Plasma_Glucose-renal_threshold), где GFR является частотой клубочковой фильтрации, которая определяет поток реакции и оказывает влияние на эффект ингибирования SGLT2.
На следующих этапах вы обновляете реакцию почечной экскреции до Plasma_Glucose + Plasma_SGLT2_Inhib -> Plasma_SGLT2_Inhib + Urinary_Glucose_Excr_AUC, где соединение-ингибитор Plasma_SGLT2_Inhib является как реагентом, так и продуктом реакции.
На вкладке Diagram щелкните серый квадратный реакционный блок с именем Renal excretion.
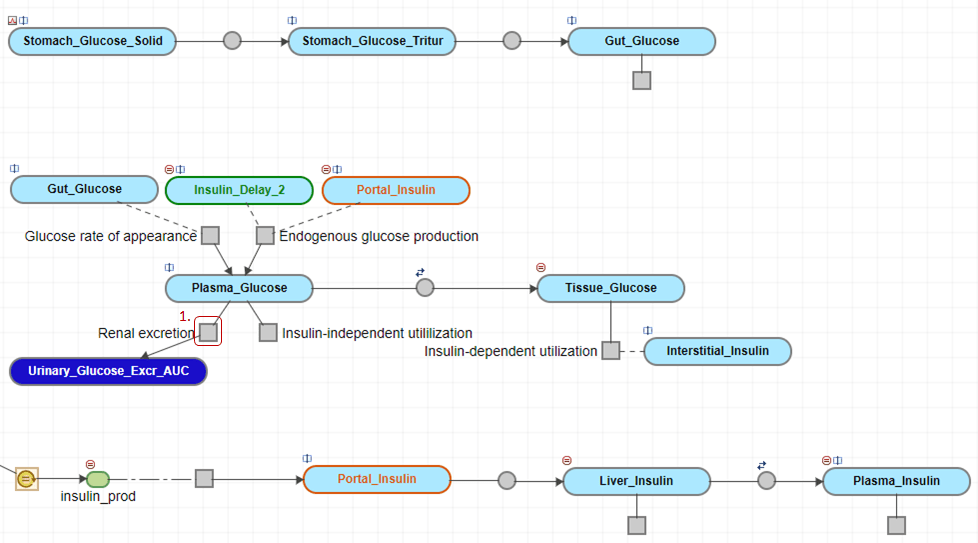
На панели Property Editor обновите строку Reaction следующим образом Plasma_Glucose + Plasma_SGLT2_Inhib -> Plasma_SGLT2_Inhib + Urinary_Glucose_Excr_AUC. Штриховая линия теперь соединяет Plasma_SGLT2_Inhib в блок реакции на вкладке Diagram.
Примечание
SimBiology использует штриховую линию, чтобы указать, что вид является как реагентом, так и продуктом реакции и не расходуется реакцией.
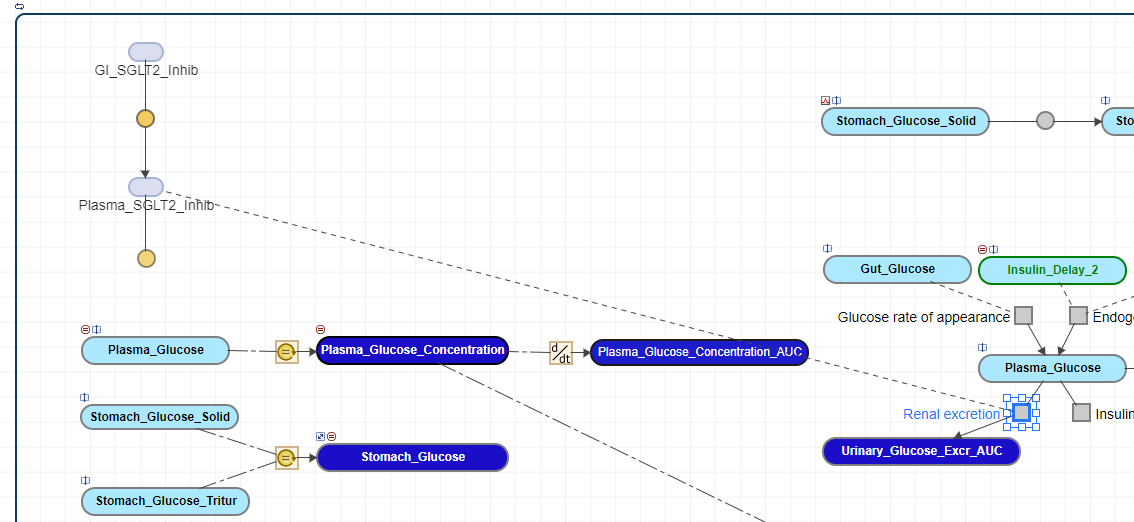
Когда существует несколько ссылок на одну и ту же величину, несколько линии соединяются с блоком. Чтобы сделать схему более ясной, можно разделить блок, то есть создать копии того же блока, чтобы каждая ссылка была соединена с другой копией блока. Можно также клонировать блок, чтобы добавить другое использование для него. Например, можно сначала клонировать блок species для ссылки в нескольких выражениях. Затем можно использовать каждый клон в каждом выражении при построении модели.
На следующих шагах вы клонируете Plasma_SGLT2_Inhib блок. Эти шаги являются необязательными и не влияют на поведение модели.
Нажмите на Plasma_SGLT2_Inhib блок в схеме.
На панели Property Editor перейдите к разделу Split.
Нажмите Clone.
На вкладке Diagram рядом с исходным блоком появляется клонированный блок. Каждый блок теперь имеет индикатор клона.
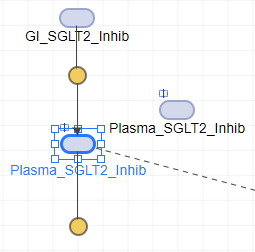
Теперь можно переместить штриховую линию в клонированный блок. Сначала щелкните штриховую линию. Нажмите Ctrl и перетащите штриховую линию в клонированный блок. Зеленый значок плюс появляется, когда линия находится рядом с клонированным блоком. Отпустите мышь, чтобы прикрепить линию к клонированному блоку.
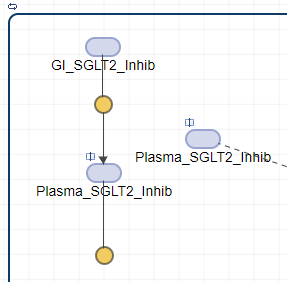
Теперь можно переместить клонированный блок ближе к Renal excretion реакционный блок, чтобы упростить чтение схемы.
Можно смоделировать внезапные изменения в поведении модели на основе заданного условия. Для примера можно сбросить количество вида в определенное время точки или когда определенный порог концентрации пересечен. SimBiology позволяет моделировать такие изменения с помощью компонента моделирования, называемого событием. Событие позволяет вам задать дискретные переходы в значениях величин, которые происходят, когда пользовательское условие становится истинным. Такое условие называется триггером события. Когда условие становится истинным, выполняется одна или несколько функций события. Для получения дополнительной информации смотрите События в Модели.
На следующих шагах вы сбрасываете общее количество глюкозы в моче, чтобы нулю каждые 24 часа, добавляя один триггер события и три функции события.
Щелкните значок плюс на панели инструментов браузера. Выберите Add Event. Приложение перемещает особое внимание в последнюю пустую строку в таблице Events.
Введите следующий триггер события: floor(time/timeDay)>num_day.
В следующей EventFcn строке введите Urinary_Glucose_Excr_AUC = 0.
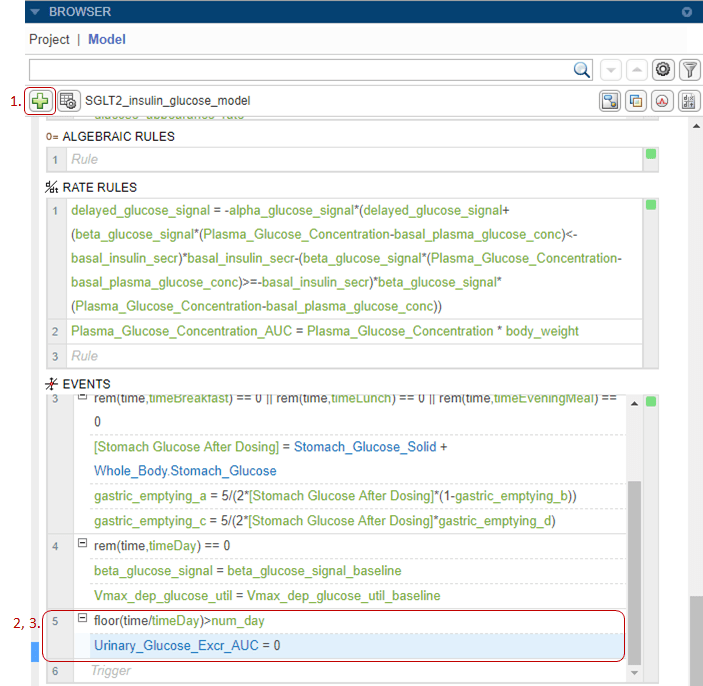
Чтобы добавить вторую функцию события к тому же событию, перейдите на Property Editor панель события. В таблице Event Fcns дважды щелкните пустую строку и введите следующее, чтобы сбросить количество дней: num_day = floor(time/1440).
Добавьте третью функцию события, чтобы сбросить общую концентрацию глюкозы в плазме путем ввода Plasma_Glucose_Concentration_AUC = 0.
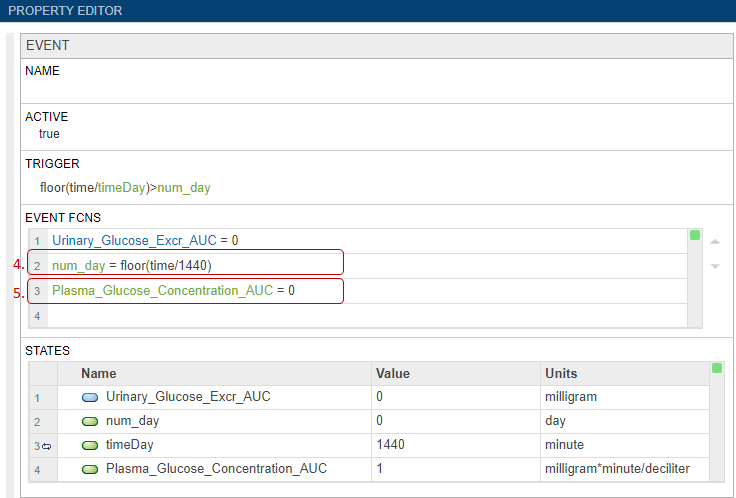
SimBiology позволяет моделировать увеличение количества вида из-за стимула, такого как пероральное или внутривенное введение препарата. Чтобы смоделировать такое увеличение количества вида, используйте компонент моделирования дозы. На следующих этапах вы моделируете потребление ингибиторного препарата, такого как один раз в день в течение x дней, дозируя GI_SGLT2_inhib вид.
На панели Browser щелкните значок Show doses на панели инструментов.
![]()
Появится вкладка Doses. В Doses разделе каждая строка представляет дозу. Столбец Type позволяет вам выбрать между Repeat Dose (по умолчанию) и Schedule Dose. Столбец Active позволяет вам выбрать, какие дозы применять при моделировании модели.
Дважды кликните Name столбец в последней пустой строке и введите SGLT2 Inhib QD.
В Properties разделе для Target Name введите GI_SGLT2_Inhib и выберите Whole_Body.GI_SGLT2_Inhib.
В разделе Dose введите следующее:
Amount = 300
Rate = 0
StartTime = 60
Interval = 1440
RepeatCount = 7
В разделе Units введите следующее:
AmountUnits = milligram
RateUnits =
TimeUnits = minute
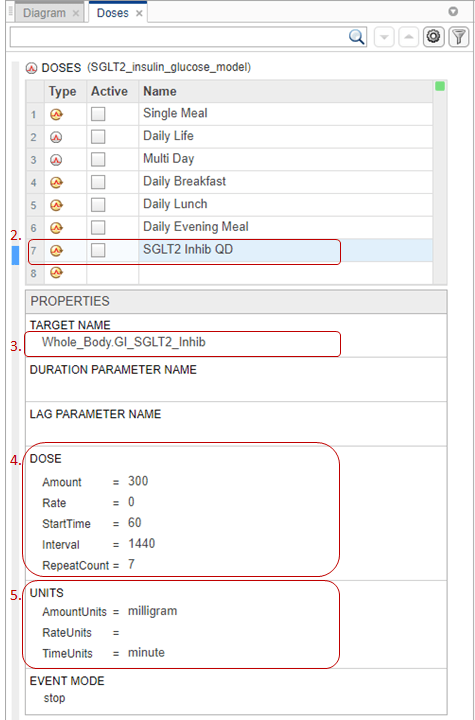
Можно смоделировать биологическую изменчивость с помощью компонента моделирования, называемого вариантом. Вариантом является набор величин с альтернативными значениями. Например, в этом примере вы можете иметь набор значений параметров для пациента с диабетом 2 типа и другое множество значений для пациента без диабета 2 типа.
В целях этого примера модель уже имеет два варианта. В следующих шагах вы открываете вкладку Variants, где можно отредактировать или добавить больше вариантов.
На панели Browser щелкните значок Show variants на панели инструментов.
![]()
Появится вкладка Variants. В Variants разделе каждая строка представляет вариант. Столбец Active позволяет выбрать варианты, которые будут применяться при моделировании модели. Можно выбрать несколько вариантов, и если существуют повторяющиеся спецификации для значения величины, последнее вхождение для значения в массиве вариантов используется во время симуляции. Приложение применяет варианты в том порядке, в котором они появляются в таблице сверху вниз. Переупорядочивание вариантов может изменить начальные условия, поскольку варианты применяются в новом порядке. Убедитесь, что вы предоставляете правильный порядок при моделировании модели в Model Analyzer приложения. В столбце Value в Content разделе показано окончательное значение величины после применения всех выбранных вариантов.
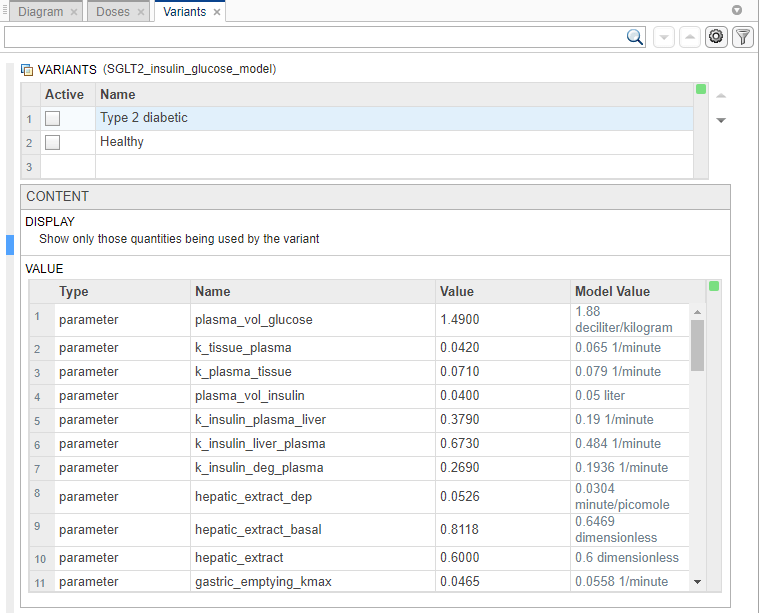
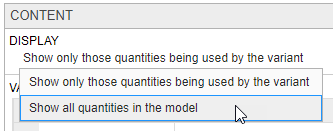
По умолчанию в Content разделе показаны только те величины, которые изменяются вариантом. Чтобы увидеть все величины модели, выберите Show all quantities in the model в разделе Display.
Можно просмотреть базовую систему уравнений, а именно обыкновенные дифференциальные уравнения (ОДУ) и правила, которые представляют модель. SimBiology получает ОДУ из реакций модели, и ОДУ определяют, какие величины интегрировать во время симуляции модели. Для получения дополнительной информации см. Симуляцию модели.
Можно использовать уравнения модели и начальные условия для отладки модели. Например, можно проверить начальные условия ОДУ, чтобы увидеть, инициализированы ли значения количества в соответствии с ожидаемыми. Можно также увидеть, как SimBiology корректирует размерности ОДУ путем деления правых сторон уравнений с объемами отсеков. Информация об исправлении объема может помочь вам отлажить неожиданные результаты симуляции, особенно когда у вас есть многокомпартовая модель с различными объемами отсеков.
Чтобы просмотреть уравнения модели, щелкните значок Show model equations на панели инструментов панели Browser.
![]()
Приложение открывает вкладку Equations.
По умолчанию приложение внедряет потоки реакции, когда она отображается в уравнениях модели. Снимите флажок Embed Fluxes, чтобы увидеть раздел Fluxes отдельно.
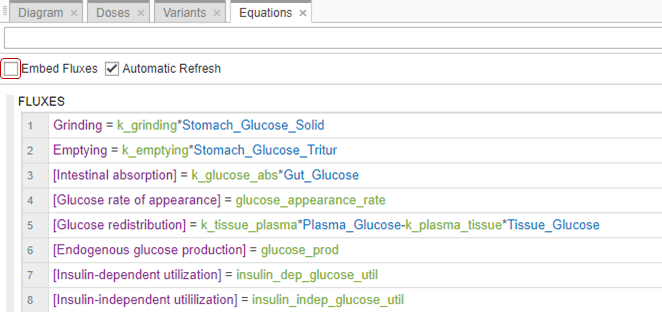
Обычно потоки реакции эквивалентны скорости реакции, за исключением того, что размерности потоков всегда amount/time. Размерности скоростей реакции могут быть в concentration/time или amount/time. Для получения дополнительной информации смотрите Вывод ОДУ из реакций.
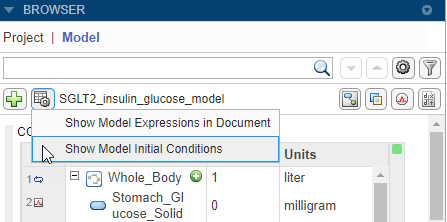
Можно просмотреть начальные условия величин модели, а именно отсеки, виды и параметры. Начальными условиями являются значения во время симуляции = 0. На панели инструментов панели Browser выберите View model documents options > Show Model Initial Conditions.
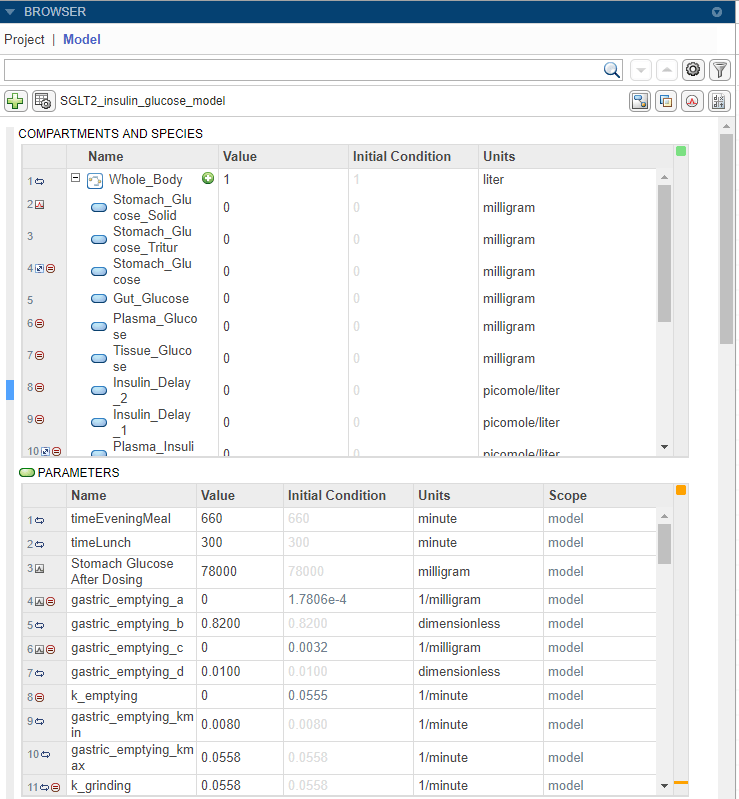
Приложение добавляет столбец с именем Initial Condition в таблицу Compartments and Species и Parameters таблицу.
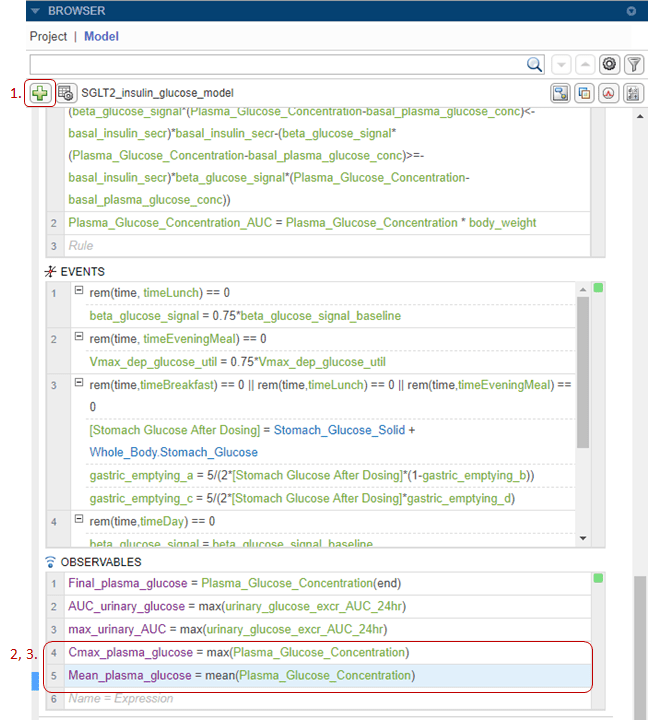
SimBiology позволяет вам выполнить вычисления после постсимуляции путем определения и оценки пользовательских выражений. Такое выражение называется Observable. На следующих этапах вы добавляете наблюдаемые выражения в модель, чтобы вычислить Cmax и средние значения профиля концентрации-времени глюкозы в плазме.
Щелкните значок плюс на панели инструментов браузера. Выберите Add Observable. Приложение перемещает особое внимание в последнюю пустую строку в таблице Observables.
Дважды кликните пустую строку и введите следующее выражение, чтобы получить значение Cmax: Cmax_plasma_glucose = max(Plasma_Glucose_Concentration).
Дважды кликните следующую пустую строку и введите следующее выражение, чтобы получить среднее значение: Mean_plasma_glucose = mean(Plasma_Glucose_Concentration).
Совет
Чтобы увидеть значения наблюдаемых выражений, симулируйте модель с помощью SimBiology Model Analyzer приложения. Запустите программу Simulate Model с Postprocessing: Calculate Observables шагом. В LastRun папке результатов симуляции программы разверните results. Векторные наблюдаемые перечислены под results. Скалярные наблюдаемые, такие как Cmax и mean в этом примере перечислены в подгруппе с именем scalars. Можно перетащить данные в таблицу данных или на график. Для получения дополнительных примеров анализа модели см. SimBiology Model Analyzer.
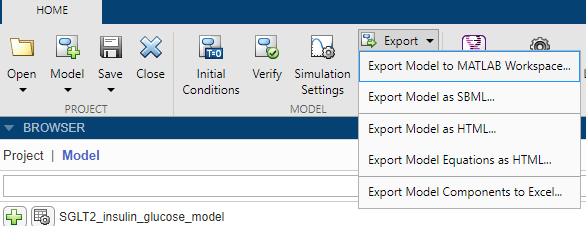
SimBiology Model Builder позволяет экспортировать модель в различные форматы файлов. Вы можете:
Экспортируйте модель в рабочее пространство MATLAB. Когда модель находится в рабочей области, можно работать над ней программно. Для получения дополнительных примеров командной строки см. Раздел «Создание моделей».
Экспорт модели в файл SBML.
Экспортируйте модель в HTML файла.
Экспортируйте уравнения модели в HTML файла.
Экспорт компонентов модели в Excel® файлы.
На вкладке Home, в разделе Model, выберите Export.
Совет
Завершенную модель для этого примера можно найти в matlabroot\examples\simbio\data\SGLT2_model.sbproj, где matlabroot - папка, в которой вы установили MATLAB.
[1] Далла Ман, Кьяра, Роберт А. Рицца и Клаудио Кобелли. Модель симуляции питания системы глюкозы-инсулина. Транзакции IEEE по биомедицинской инженерии 54, № 10 (октябрь 2007): 1740-49. https://ieeexplore.ieee.org/document/4303268.
[2] Далла Ман, Кьяра, М. Камиллери и К. Кобелли. «Системная модель поглощения глюкозы в полости рта: валидация на данных золотого стандарта». Транзакции IEEE по биомедицинской инженерии 53, № 12 (декабрь 2006): 2472-78. https://ieeexplore.ieee.org/document/4015600.
[3] Райт, Эрвест М., Дональд Д. Ф. Лоо и Брюс А. Хираяма. Биология переносчиков глюкозы натрия человека. Физиологические отзывы 91, № 2 (Апрель 2011): 733-94. https://journals.physiology.org/doi/full/10.1152/physrev.00055.2009.
SimBiology Model Analyzer | SimBiology Model Builder