Этот пример показывает, как идентифицировать важные параметры модели для модели роста опухоли [1]. В этом примере вы вычисляете локальные зависящие от времени чувствительности роста опухоли относительно параметров модели для противоопухолевого препарата.
Модель, используемая в этом примере, является SimBiology® реализация фармакокинетической/фармакодинамической (PK/PD) модели Simeoni et al. Он количественно определяет эффект противоопухолевых препаратов на кинетику роста опухоли из исследований на животных in vivo. Фармакокинетика препарата описывается двухкамерной моделью с IV болюсным дозированием болюсного внутривенного введения и линейным элиминацией (ke) из Central отсека. Рост опухоли является двухфазным процессом с начальным экспоненциальным ростом с последующим линейным ростом. Скорость роста пролиферирующих опухолевых камер описывается как
L0, L1, и Ψ являются параметрами роста опухоли, x1 - вес распространяющихся опухолевых клеток, и w общий вес опухоли. При отсутствии каких-либо лекарств опухоль состоит только из пролиферирующих камер, то есть w = x1. В присутствии противоопухолевого агента часть пролиферирующих камер превращается в непролиферирующие камеры. Скорость этого преобразования принимается в зависимости от концентрации лекарственного средства в плазме и k2 фактора эффективности. Непролиферирующие камеры x2 пройти ряд транзитных стадий (x3 и x4) и в конечном счете очищаются от системы. Прохождение транзитных отсеков моделируется как процесс первого порядка с константой скорости k1.
Модель SimBiology вносит эти коррективы в фармакодинамику роста опухоли:
Вместо того, чтобы определять вес опухоли как сумму x1, x2, x3 и x4, модель определяет вес опухоли по реакции, названной Increase, null → tumor_weight, со скоростью реакции .
tumor_weight - общий вес опухоли, x1 - вес пролиферирующих опухолевых камер, и L0, и L1 - параметры роста опухоли.
Точно так же модель определяет уменьшение массы опухоли реакцией под названием Decay, tumor_weight → nullсо скоростью реакции k1*x4. Постоянное k1 является параметром прямой скорости, и x4 является последним видом в серии переходных сокращений веса опухоли.
ke является функцией зазора и объема центрального отделения: ke = Cl_Central/Central.
Анализ чувствительности позволяет вам определить, какие константы скорости и концентрации в модели оказывают значительное влияние на общее поведение модели. Этот пример показывает, как исследовать, какие параметры модели чувствительны к росту опухоли, используя встроенную программу анализа чувствительности. В частности, этот пример показывает, как вычислить чувствительность tumor_weight видов относительно параметров модели следующим образом:
Предположим, что вы уже калибровали параметры модели роста опухоли к экспериментальным данным PK/PD и сохраняли оценки параметров и начальные условия как вариант, называемый parameterEstimates в вашей модели. В качестве последующего анализа вы хотите выяснить, какие параметры модели чувствительны к росту опухоли. Следующие шаги показывают, как это сделать с помощью встроенной программы анализа чувствительности.
В MATLAB® командная строка, загрузите модель (m1) путем ввода:
sbioloadproject tumor_growth_vpop_sa.sbprojОткройте модель в приложении SimBiology Model Analyzer путем ввода:
simBiologyModelAnalyzer(m1)
Также можно открыть приложение, нажав SimBiology Model Analyzer на вкладке Apps. Затем загрузите модель из приложения, выбрав Model > Import Model from MATLAB.
На вкладке Home выберите Program > Calculate Sensitivities. Откроется новая программа.
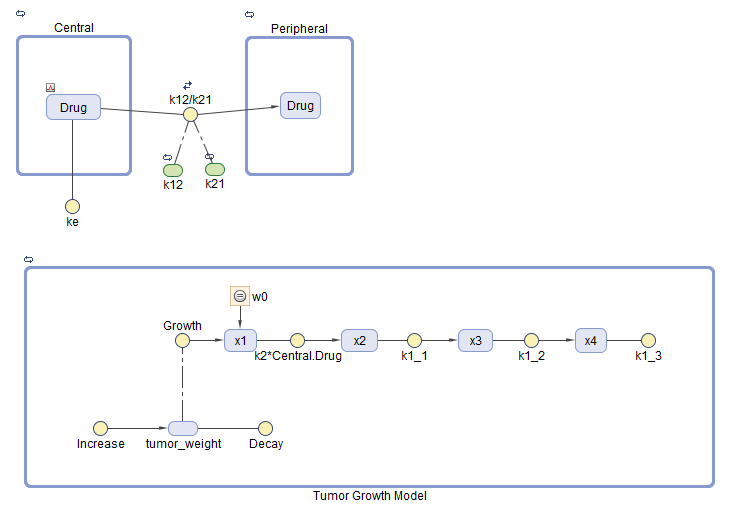
В Variants разделе Model шага (шага настройки) программы выберите parameterEstimates. Этот вариант содержит ранее оцененные значения параметров и начальные условия для препарата.
В Doses разделе выберите interval_dose. Эта повторная доза применяет 30 мг препарата каждые 4 дня, начиная с 7 дня, в общей сложности пять раз.
В States To Log разделе выберите только [Tumor Growth Model].tumor_weight.
Совет
Контекстные меню Variants, Doses и States To Log таблиц содержат опции, чтобы добавить соответствующие компоненты модели и показать детали этих компонентов в приложении Model Builder. Доступ к контекстному меню путем щелчка правой кнопкой мыши по таблице.
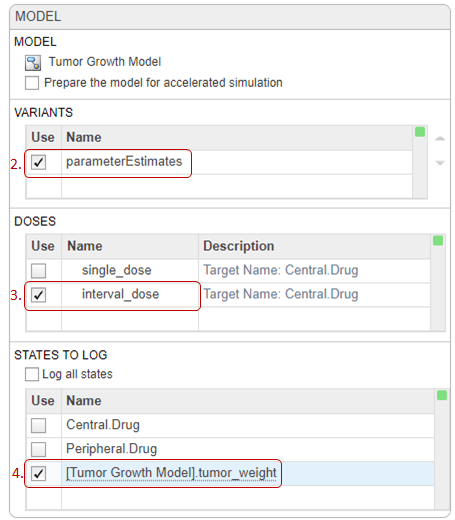
На Simulation этапе (шаге выполнения), по умолчанию, метод нормализации устанавливается на Full (full dedimensionalization), что означает, что приложение полностью нормализует чувствительности, так что они могут сравниваться друг с другом. Для получения дополнительной информации смотрите Normalization.
Совет
Каждый шаг выполнения программы можно запустить отдельно. Шаг выполнения включает кнопку Run рядом с именем шага. Выполнение отдельного шага особенно полезно, если программа содержит несколько шагов, и необходимо увидеть промежуточные результаты определенного шага. При этом можно внести необходимые корректировки перед запуском следующего шага или всей программы. Чтобы запустить программу целиком, нажмите кнопку Run на вкладке Home.
В Sensitivities to Compute разделе щелкните правой кнопкой мыши в любом месте таблицы и выберите Add All Constant Parameters. Очистить ke, потому что это определяется как функция Central том и Cl_Central, который уже является входом. Щелкните последнюю пустую строку и введите tumor. Затем выберите [Tumor Growth Model].tumor_weight из списка. Затем в качестве выходов добавляют tumor_weight вид.
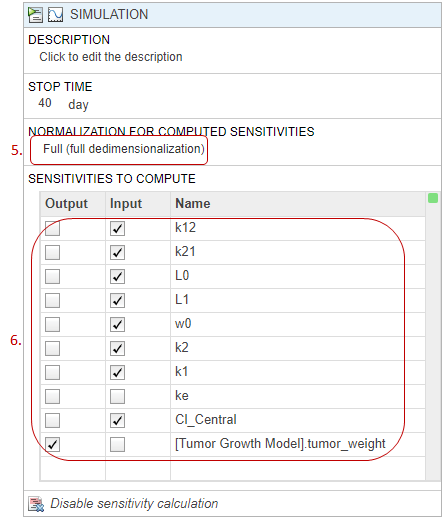
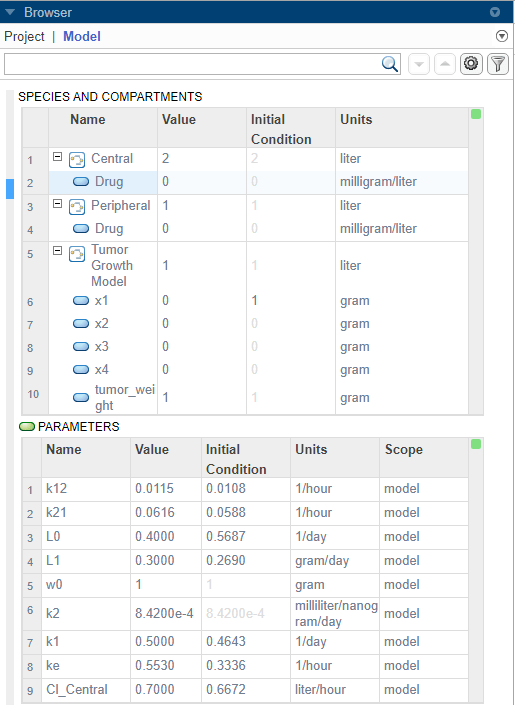
Можно просмотреть начальные условия величин модели, а именно отсеки, виды и параметры, перед симуляцией модели. Начальными условиями являются значения величины во время симуляции = 0 после применения правил присвоения, вариантов и доз (если таковые имеются во время = 0). Для получения дополнительной информации см. Симуляцию модели. Можно использовать эту информацию для отладки модели и проверки, инициализированы ли значения величины, как вы ожидаете, перед моделированием модели.
На панели Browser нажмите Model.
Щелкните правой кнопкой мыши в любом месте таблицы Species and Compartments или Parameters таблицы. Выберите Show Model Initial Conditions.
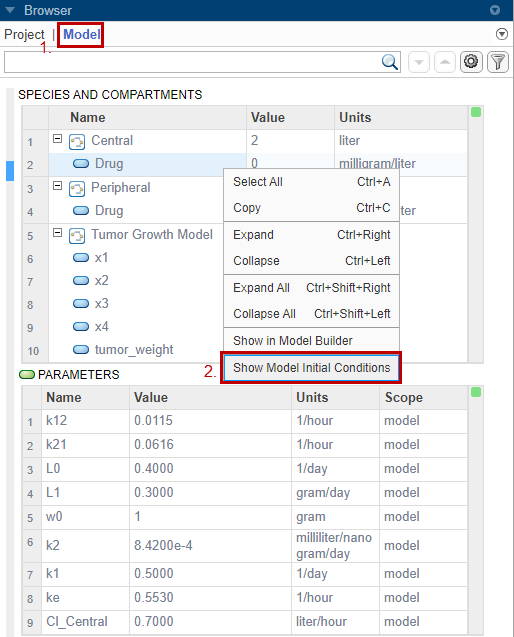
Приложение показывает столбец Initial Condition в обеих таблицах количества. Эти значения являются тем, что программа использует во время = 0 при моделировании.
Затем запустите программу, нажав Run на вкладке Home.
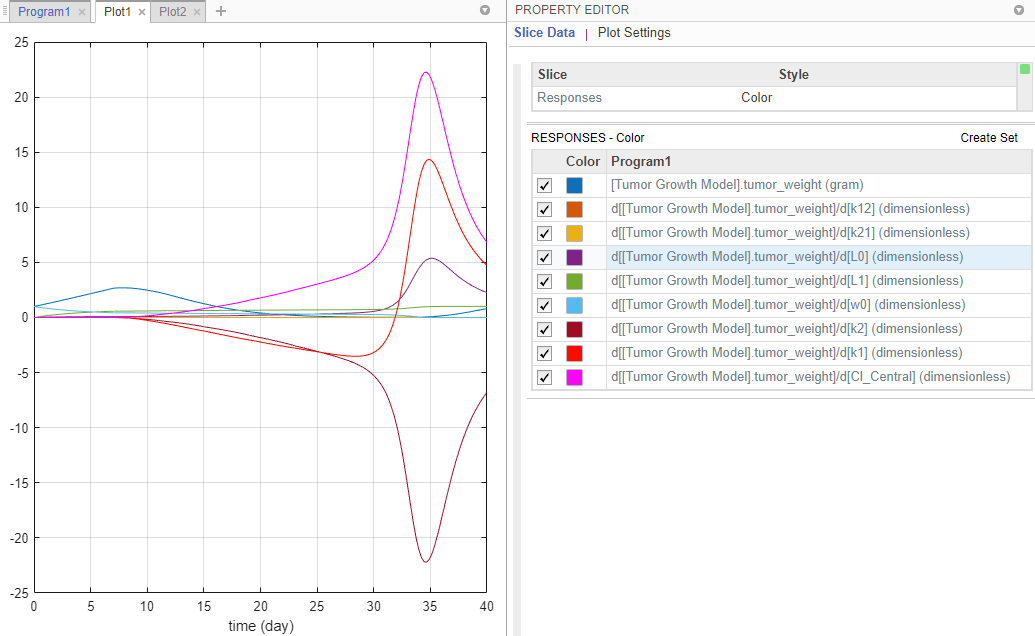
После анализа чувствительности программа автоматически генерирует два графика. Щелкните вкладку Plot1, чтобы просмотреть график времени течения веса опухоли и других значений чувствительности d[tumor_weight]/d[parameter]. Временные графики для d[tumor_weight]/d[k21] и d[tumor_weight]/d[k12] оставаться относительно плоским по сравнению с другими чувствительностью, что указывает на то, что рост опухоли не чувствителен к k21 и k12 параметрам. Этот Property Editor позволяет выбрать графики для отображения.
Совет
Графики поддерживаются данными, которые в настоящее время присутствуют в рабочей области приложения. Графики не являются снимками файловой системы. Когда данные (экспериментальные данные или результаты симуляции) удаляются или изменяются, графики также обновляются в соответствии с изменениями в базовых данных.
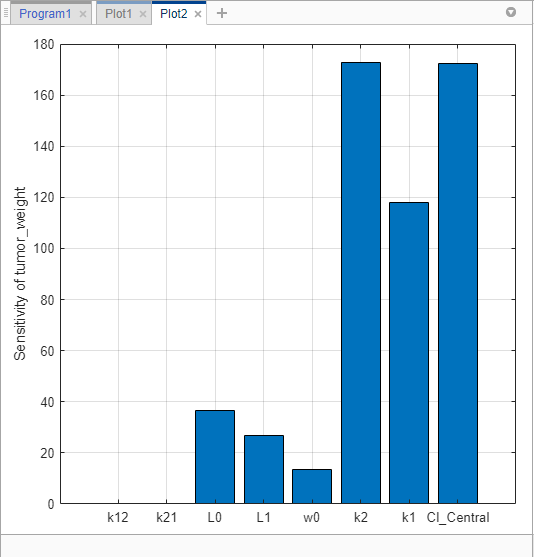
Можно также исследовать те же данные чувствительности, проверяя величину вычисленных чувствительности, интегрированных с течением времени. Второй сгенерированный график (Plot2) показывает штриховой график, содержащий такую информацию. График показывает, что вес опухоли чувствителен к L0, L1, w0, k2, k1, и Cl_Central, но не к k12 и k21.
Сохраните результаты анализа чувствительности в отдельной папке.
Щелкните Project на панели Browser. Разверните папку Program1.
Щелкните правой кнопкой мыши папку LastRun. Выберите Save Data.
В диалоговом окне Save Data введите sa_data как имя данных.
Результаты анализа чувствительности показывают, что вес опухоли не чувствителен к значениям модели k12 и k21. Исследуйте, приводит ли изменение этих значений параметров к различным результатам чувствительности.
Варьируйте k12. Во-первых, варьируйте значения для параметра k12.
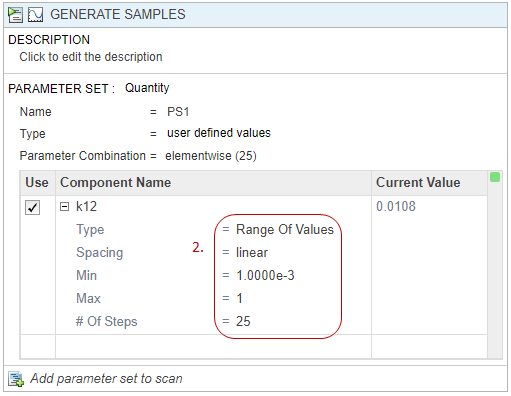
Перейдите на вкладку Program1. Нажмите (+) плюс в верхней части программы и выберите Generate Samples.
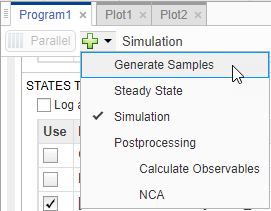
Появится Generate Samples шаг. В Parameter Set разделе шага дважды кликните пустую камеру в Component Name и введите k12. Установите следующие опции:
Type - Область значений значений
Spacing - линейный
Min - 1e-3
Max — 1
# Of Steps — 25
Отключите генерацию графика по умолчанию, нажав кнопку вывода в верхней части шага.
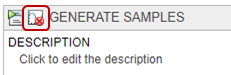
На Simulation этапе, под Sensitivities to Compute, очистите все входы, кроме k12. Сохраните tumor_weight как выход. Также отключите генерацию графика по умолчанию, нажав кнопку plot в верхней части шага.
На вкладке Home нажмите кнопку Run, чтобы запустить программу целиком.
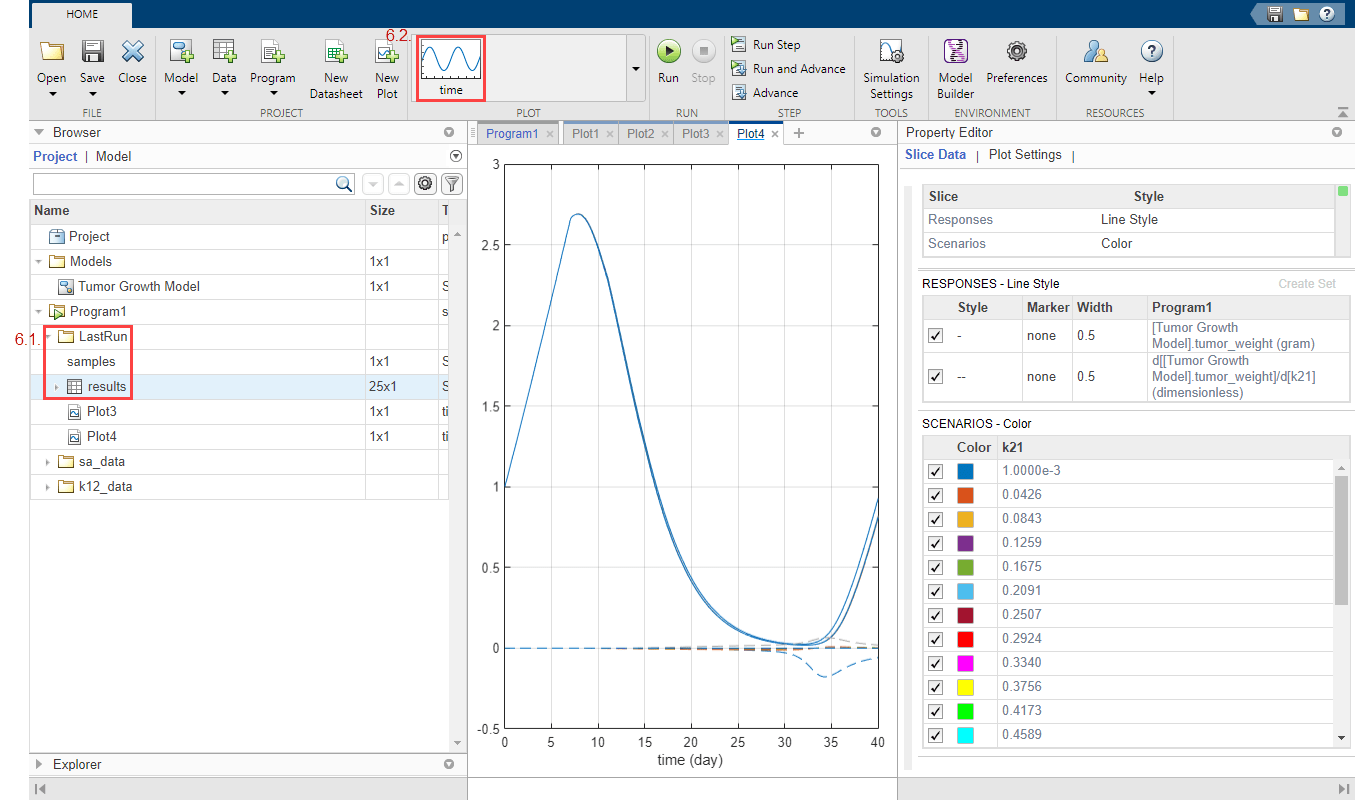
После завершения симуляции results чувствительности сохраняются в папке LastRun. Разверните папку LastRun. Нажмите results. Затем выберите time из Plot раздела на вкладке Home.
Responses на графике соответствуют массе опухоли и чувствительности массы опухоли к k12. Каждый ответ выводится на график с использованием другого стиля линии. Эти Scenarios соответствуют каждому скану параметра (сценарию симуляции). Каждый сценарий строится с использованием другого цвета.

Настройте график так, чтобы график вместо этого использовал другой цвет для каждого отклика. В Slice Data таблице очистите Style Scenarios, выбрав пустой. Установите Responses значение Color.
В Responses таблице очистите tumor_weight и сохраните только результаты чувствительности.
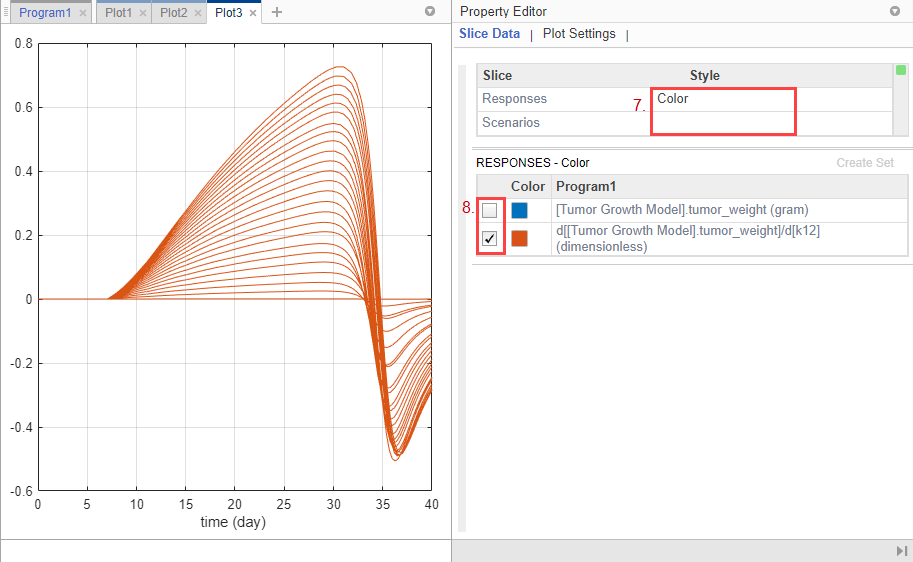
График предполагает, что вес опухоли чувствителен к некоторым k12 значениям параметров, когда исследуются варианты модели, отличные от предполагаемых значений параметров.
Постройте график чувствительности k12 вместе с предыдущими результатами чувствительности, чтобы увидеть, насколько чувствителен рост опухоли к k12 относительно других параметров модели. На панели Browser разверните ранее сохраненную sa_data папки. Затем разверните results.
Мультивыберите (Shift + Click) вводы данных чувствительности для L0, L1, w0, k2, k1, и Cl_Central. Затем перетащите их на график k12 чувствительности Plot3.
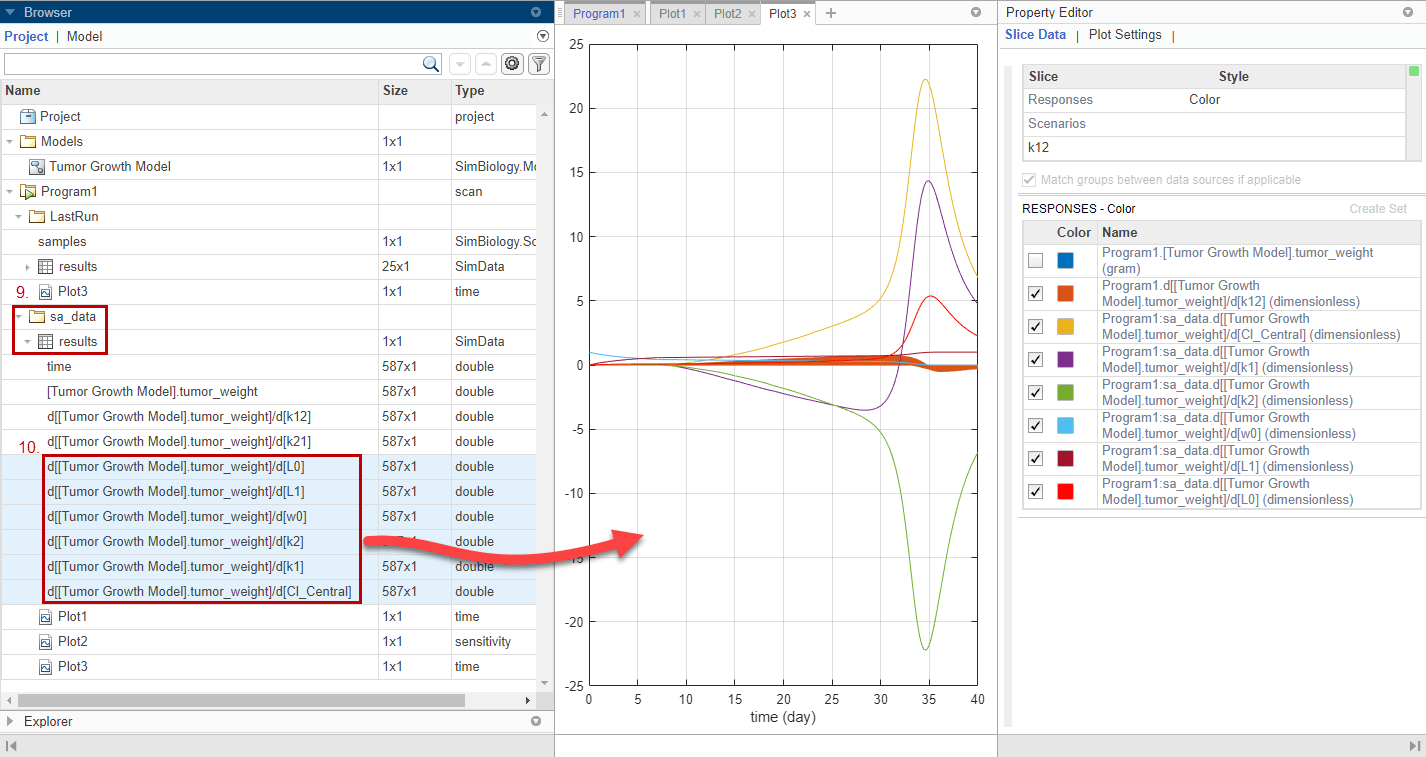
График предполагает, что рост опухоли все еще более чувствителен к большинству других параметров, чем k12.
Сохраните результаты в отдельной папке. Щелкните правой кнопкой мыши LastRun и выберите Save Data. Введите k12_data как имя данных.
Варьируйте k21. Можно выполнить подобный анализ, варьируя k21 и сравнивая его результаты с другими чувствительностью параметра.
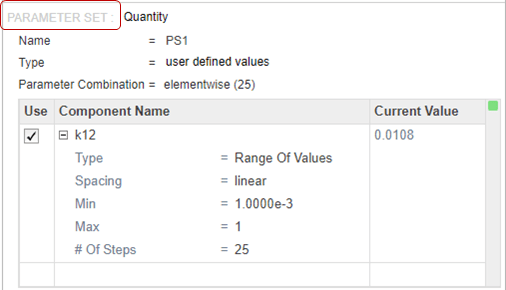
Вернитесь к Program1. На Generate Samples шаге нажмите Add parameter set to scan. Появится новый раздел Parameter Set.
Дважды кликните пустую камеру в Component Name и введите k21. Установите те же опции, что и k12:
Type - Область значений значений
Spacing - линейный
Min - 1e-3
Max — 1
# Of Steps — 25
Отключите первый набор параметров (PS1). Укажите верхний угол Parameter Set раздела PS1 и нажмите кнопку действия. Затем выберите Disable Parameter Set.

После этого Parameter Set становится серым, чтобы указать, что он был успешно отключен.
На Simulation шаге выберите k21 как единственный вход и tumor_weight как выход. Отключите генерацию графика по умолчанию, нажав кнопку вывода в верхней части шага.
Нажмите Run с вкладки Home, чтобы запустить программу снова.
После завершения симуляции results чувствительности сохраняются в папке LastRun. Разверните папку LastRun. Нажмите results. Затем выберите time из Plot раздела на вкладке Home.
В Slice Data таблице очистите Style Scenarios, выбрав пустой. Установите Responses значение Color.
В Responses таблице очистите tumor_weight и сохраните только результаты чувствительности.
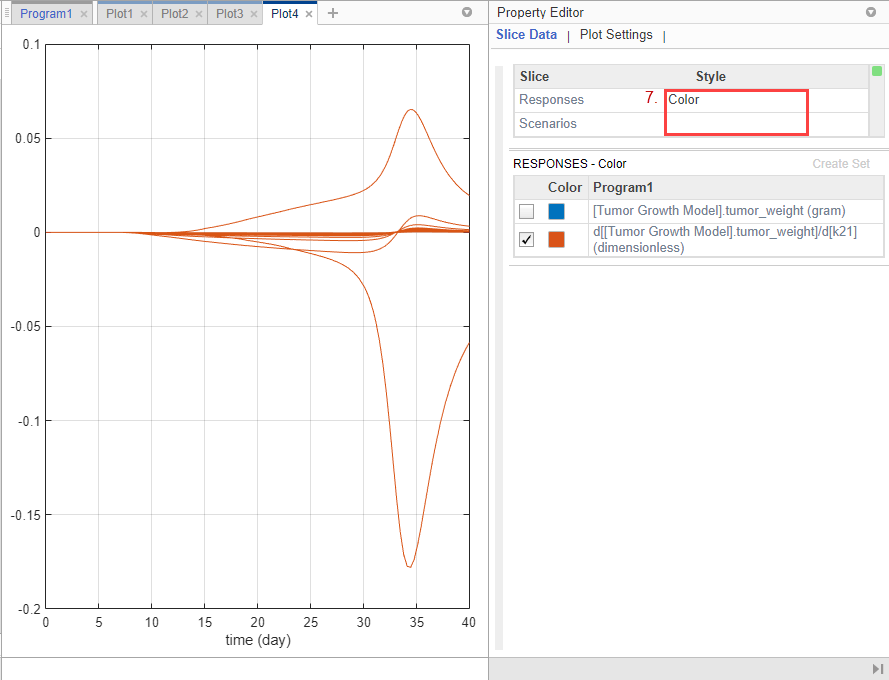
График предполагает, что вес опухоли чувствителен к некоторым k21 значениям параметров, когда исследуются варианты модели, отличные от предполагаемых значений параметров.
Постройте график чувствительности k21 вместе с предыдущими результатами чувствительности для сравнения. Нажмите Plot3. На панели Browser разверните папку LastRun. Затем разверните results.
Перетащите k21 данные чувствительности d[[Tumor Growth Model].tumor_weight]/d[k21] на к Plot3.
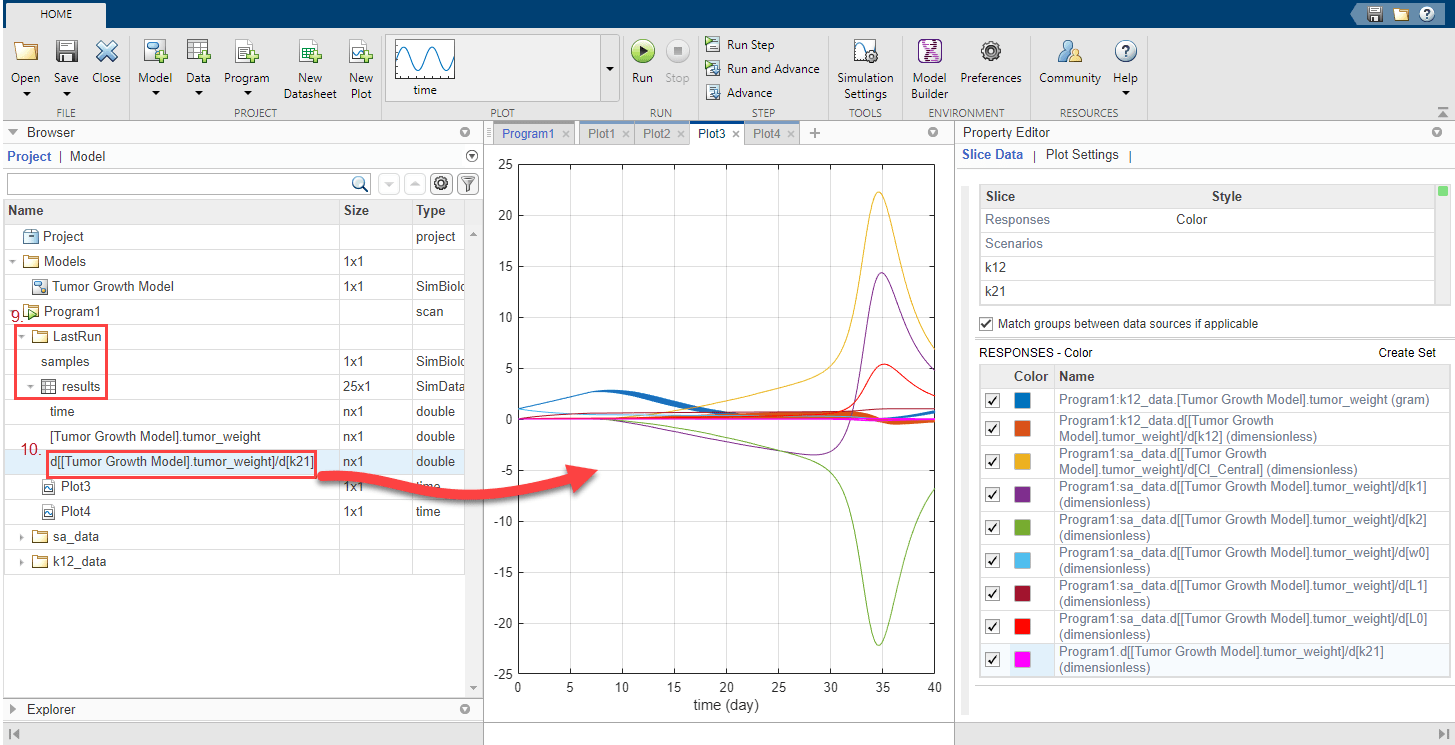
График предполагает, что рост опухоли относительно более чувствителен к большинству других параметров, чем k21 и k12.
Этот пример показывает, как выполнить анализ чувствительности, чтобы найти важные параметры модели, к которым чувствителен рост опухоли. Первоначальный анализ делает вывод, что вес опухоли не чувствителен к некоторым параметрам. Затем пример исследует пространство параметров этих менее чувствительных параметров и сравнивает результаты чувствительности с результатами других параметров модели. Можно использовать такую информацию о чувствительности для других анализов, таких как выполнение симуляций Монте-Карло путем варьирования чувствительных параметров, чтобы исследовать динамику модели и биологическую изменчивость.
[1] Simeoni, M., P. Magni, C. Cammia, G. De Nicolao, V. Croci, E. Pesenti, M. Germani, I. Poggesi, and M. Rocchetti. 2004. Прогнозирующее фармакокинетико-фармакодинамическое моделирование кинетики роста опухоли на ксенотрансплантатных моделях после введения противоопухолевых агентов. Исследования рака. 64:1094-1101.