Этот пример показывает, как выполнить некомпартментный анализ, чтобы вычислить параметры NCA и оценить параметры модели роста опухоли [1] из экспериментальных данных с использованием нелинейной регрессии в приложении SimBiology Model Analyzer.
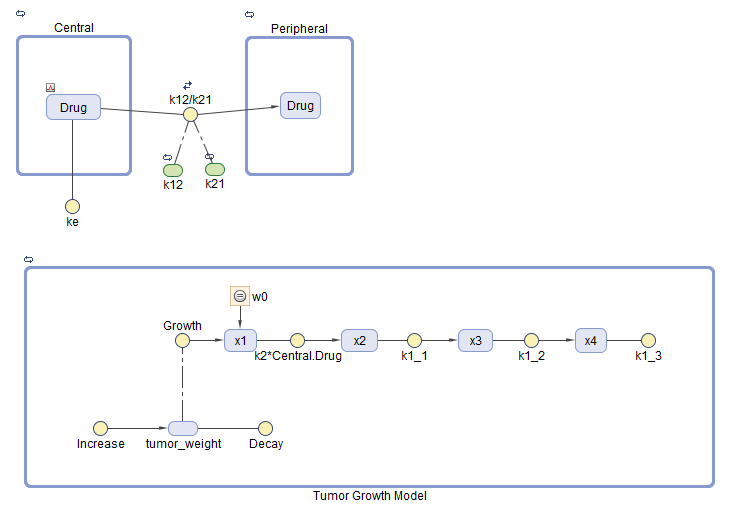
Модель, используемая в этом примере, является SimBiology® реализация фармакокинетической/фармакодинамической (PK/PD) модели Simeoni et al. Он количественно определяет эффект противоопухолевых препаратов на кинетику роста опухоли из исследований на животных in vivo. Фармакокинетика препарата описывается двухкамерной моделью с IV болюсным дозированием болюсного внутривенного введения и линейным элиминацией (ke) из Central отсека. Рост опухоли является двухфазным процессом с начальным экспоненциальным ростом с последующим линейным ростом. Скорость роста пролиферирующих опухолевых камер описывается как
L0, L1, и Ψ являются параметрами роста опухоли, x1 - вес распространяющихся опухолевых клеток, и w общий вес опухоли. При отсутствии каких-либо лекарств опухоль состоит только из пролиферирующих камер, то есть w = x1. В присутствии противоопухолевого агента часть пролиферирующих камер превращается в непролиферирующие камеры. Скорость этого преобразования принимается в зависимости от концентрации лекарственного средства в плазме и k2 фактора эффективности. Непролиферирующие камеры x2 пройти ряд транзитных стадий (x3 и x4) и в конечном счете очищаются от системы. Прохождение транзитных отсеков моделируется как процесс первого порядка с константой скорости k1.
Модель SimBiology вносит эти коррективы в фармакодинамику роста опухоли:
Вместо того, чтобы определять вес опухоли как сумму x1, x2, x3 и x4, модель определяет вес опухоли по реакции, названной Increase, null → tumor_weight, со скоростью реакции .
tumor_weight - общий вес опухоли, x1 - вес пролиферирующих опухолевых камер, и L0, и L1 - параметры роста опухоли.
Точно так же модель определяет уменьшение массы опухоли реакцией под названием Decay, tumor_weight → nullсо скоростью реакции k1*x4. Постоянное k1 является параметром прямой скорости, и x4 является последним видом в серии переходных сокращений веса опухоли.
ke является функцией зазора и объема центрального отделения: ke = Cl_Central/Central.
Экспериментальные (синтетические) данные содержат измерения от восьми пациентов для трех ответов: измеренные концентрации препарата в центральном отсеке, в периферическом отсеке и измеренный вес опухоли. Данные также содержат информацию о дозах, и каждый пациент получает внутривенную дозу в день 7.
Набор данных содержит следующие столбцы.
ID - Идентификаторы пациентов
Time - Время выполнения измерений
CentralConc - Концентрация наркотиков в центральном отделении
PeripheralConc - Концентрация наркотиков в периферийном отделении
Dose - Информация о дозах для каждого пациента
NaN значения используются всякий раз, когда нет измерения или не дается доза.
Откройте приложение SimBiology Model Analyzer, набрав simBiologyModelAnalyzer в командной строке или нажатием значка приложения на вкладке Apps.
На вкладке Home приложения выберите Open.
Перейдите к папке matlabroot\examples\simbio\data\. matlabroot папка, в которой установлен MATLAB. Выберите файл проекта с именем tumor_growth_fitPKPD.sbproj. На панели Browser папка Models содержит Tumor Growth Model и папка Data1 содержит экспериментальные данные.
Классифицируйте измеренные отклики данных как зависимые переменные. На панели Browser разверните папку Data1 и дважды щелкните Datasheet1.
В таблице Data1 дважды кликните мышью Classification под CentralConc. Выберите dependent. Повторите тот же процесс для PeripheralConc и TumorWeight. Теперь все столбцы данных имеют соответствующие классификации, и данные готовы к использованию.

После загрузки данных можно визуализировать измеренные отклики.
На панели «Браузер» нажмите кнопку Data1.
На вкладке Home, в разделе Plot, нажмите time график. Приложение генерирует график времени из всех трех ответов, а именно: CentralConc, PeripheralConc и TumorWeight.
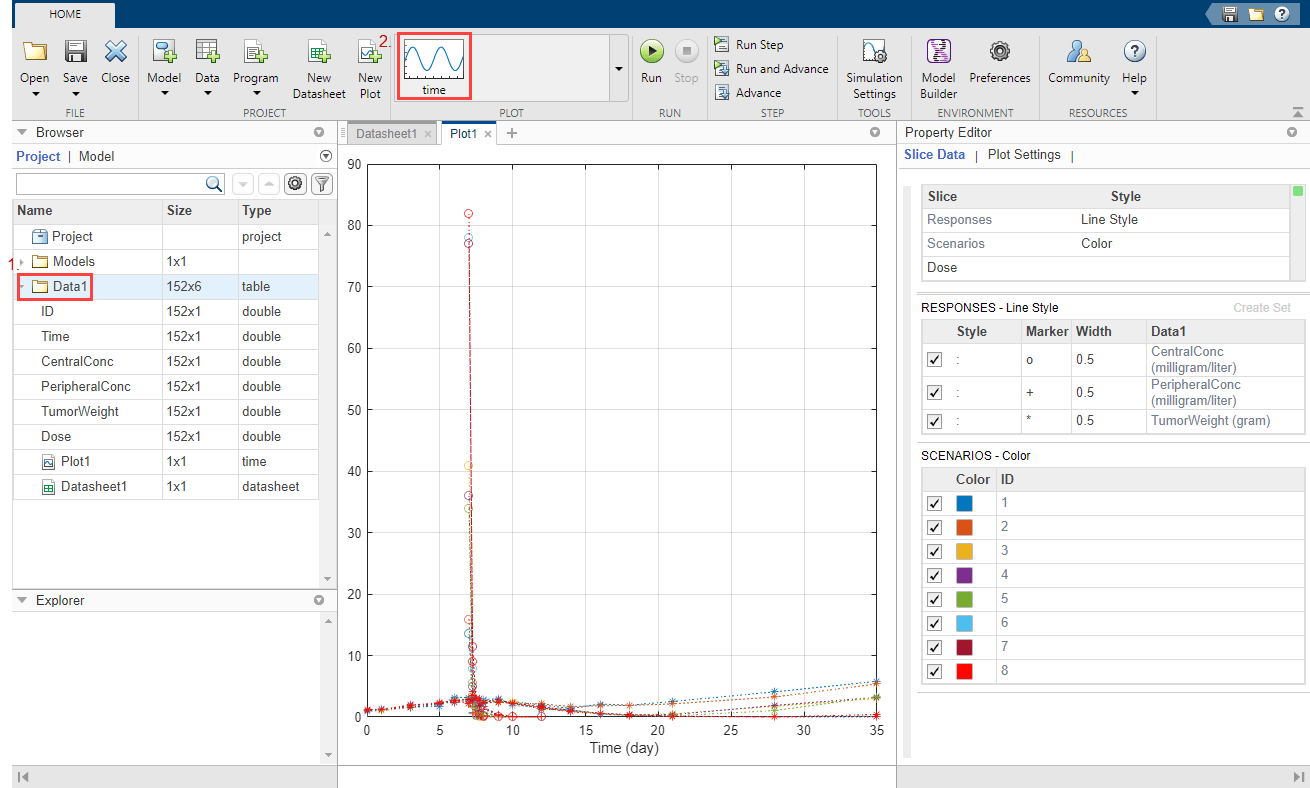
Во временном графике по умолчанию Responses соответствуют измеренным откликам и строятся с использованием различных стилей линии. Scenarios относится к различным группам (восемь пациентов) в данных и строится с использованием различных цветов.
Совет
Графики поддерживаются данными, которые в настоящее время присутствуют в рабочей области приложения. Графики не являются снимками файловой системы. Когда данные (экспериментальные данные или результаты симуляции) удаляются или изменяются, графики также обновляются в соответствии с изменениями в базовых данных.
Можно настроить график, чтобы сделать его более ясным. Для примера можно построить график данных PD (TumorWeight) на другой оси, чем данные PK (CentralConc и PeripheralConc). Для этого создайте две дифферентные группы (наборы) откликов, где первый набор содержит только TumorWeight а второй набор содержит CentralConc и PeripheralConc.
Щелкните правой кнопкой мыши TumorWeight (gram) в таблице Responses (средняя таблица в Property Editor) и выберите Create New Set. Приложение создает Set 1 и Set 2. Set 1 содержит только TumorWeight, которая теперь нанесена на другую ось, чем Set 2, которая содержит CentralConc и PeripheralConc.
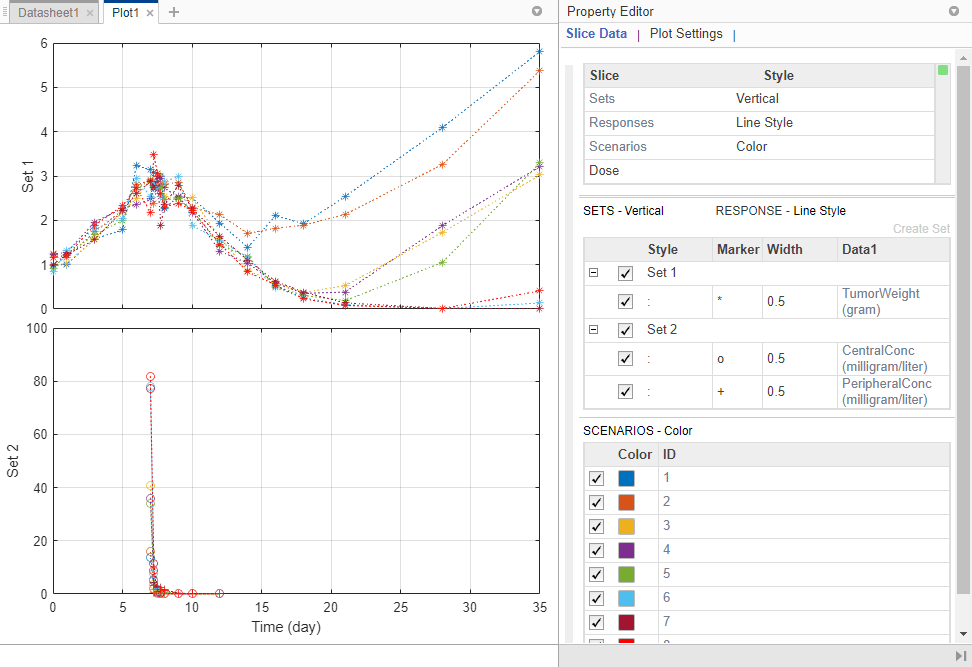
Теперь Slice таблица (верхняя таблица в Property Editor) содержит Sets. Эта таблица представляет собой сводную таблицу всех переменных среза, которые в данный момент присутствуют в графике, и соответствующих им стилей графика. На этом текущем графике переменные среза являются Sets, Responses, Scenarios и Dose.
Совет
Можно срезать данные с помощью различных переменных среза. Каждая переменная среза появляется на графике с другим визуальным стилем (или каналом), таким как цвет, стиль линии и положение осей. Переменные среза могут представлять атрибуты данных, такие как отклики или сценарии (то есть группы или выполняемые симуляции). Переменные среза также могут быть ковариатами или значениями параметров, связанными со сценарием или группой. По умолчанию приложение предоставляет переменные среза для различных переменных отклика и различных сценариев в нанесенных на график данных. Можно добавить другие визуальные стили (или каналы) для наборов откликов и связанных параметрических или ковариатных переменных.
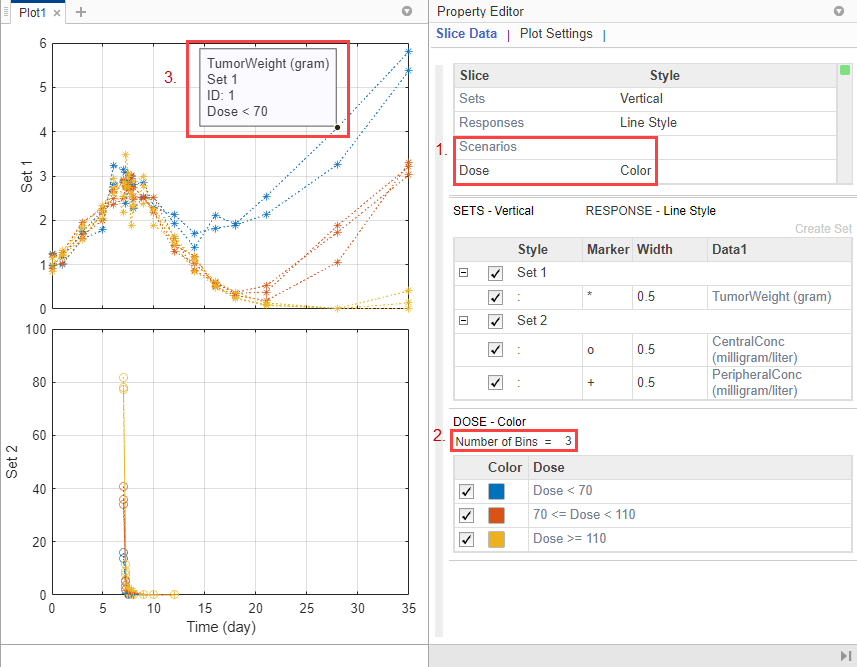
Можно также сгруппировать ответы на основе различных суммарных доз, которые получают пациенты. Существует три группы доз: 30, 75 и 150 мг.
В Slice таблице в Dose строке дважды кликните пустую камеру и выберите Color. Красный индикатор появляется, потому что другая переменная среза (Scenarios) имеет тот же стиль графика. Очистить стиль (визуальный канал) для Scenarios путем выбора пустого.
В Dose таблице (нижняя таблица) приложение автоматически связало суммарные дозы. Установите Number of Bins значение 3. Теперь можно увидеть, что суммарная доза влияет на размер опухоли. Чем выше доза, тем меньше становится опухоль.
Можно также запросить соответствующую группу доз из каждой линии, отобразив ее всплывающие подсказки. Нажмите Ctrl и щелкните синюю линию, чтобы отобразить всплывающие подсказки. Чтобы удалить его, Ctrl + Нажмите еще раз где угодно в той же линии.
Используя фармакокинетические данные о препарате, можно оценить параметры NCA. NCA является модельным агностиком и может дать представление о фармакокинетике препарата без каких-либо основополагающих предположений. Можно использовать некоторые результаты NCA в качестве начальных оценок при калибровке модели к данным, как обсуждается ниже в этом примере. Для получения дополнительной информации о списке доступных параметров NCA и их формулах, смотрите Некомпартментальный анализ.
На вкладке Home выберите Program > Non-Compartmental Analysis . Появится новая программа (Program1).
Шаг настройки Data программы определяет набор данных, который будет использоваться для анализа NCA. В этом примере программа автоматически выбирает Data1.
Шаг
выполнения NCA задает связи столбцов данных и детали алгоритма. В таблице Definition установите Concentration равным CentralConc. Оставьте другие настройки без изменений.
На вкладке Home нажмите Run.
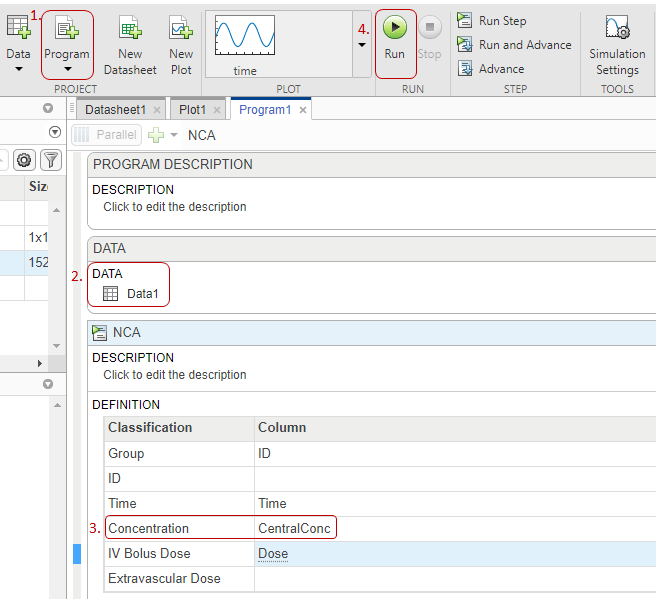
После завершения анализа NCA приложение открывает новая таблица данных, содержащий результаты.
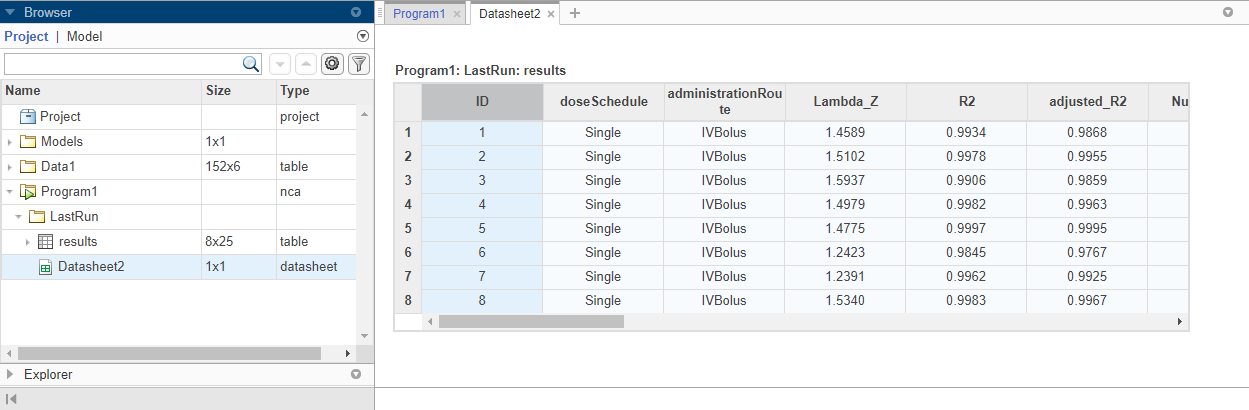
Программа также сохраняет результаты в LastRun папке программы по умолчанию. Чтобы получить доступ к результатам, на панели Browser разверните папку Program1. Затем разверните LastRun папку. Результаты NCA хранятся в таблице с именем results. Для получения дополнительной информации о вычисленных параметрах NCA, см. Некомпартментный анализ.
Можно экспортировать результаты NCA в MATLAB® Рабочая область и выполните дальнейший анализ данных в командной строке.
Щелкните правой кнопкой мыши results. Выберите Export Data to MATLAB Workspace.
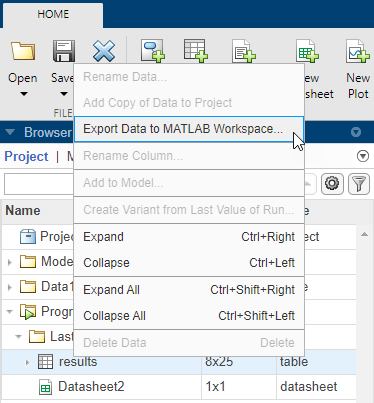
Откроется диалоговое окно SimBiology Data Export. Измените имя переменной на ncatable. Нажмите OK.
После экспорта данных в рабочее пространство MATLAB можно проанализировать данные в командной строке. Например, можно вычислить средний клиренс препарата из данных NCA и использовать его как значение параметра модели.
SimBiology предоставляет различные методы регрессии для оценки параметров модели на основе экспериментальных данных. В этом примере подробно описываются шаги для использования нелинейного метода регрессии lsqnonlin (требует Optimization Toolbox™), чтобы соответствовать модели данным. Если у вас нет Optimization Toolbox, приложение использует fminsearch вместо этого. В целях примера оцениваются только некоторые параметры модели PK/PD, а именно: k1, L0, L1, Cl_Central, k12 и k21.
На вкладке Home выберите Program > Fit Data . Новая программа (Program2) появится на новой вкладке. Data и Model стадии были предварительно заполнены Data1 и Tumor Growth Model, соответственно.
По умолчанию Fit шаг автогенерирует графики после завершения подбора кривой. Отключите генерацию графика, пока щелкнув значок plot в верхней части Fit шага программы. Графики будут исследованы позже в примере.
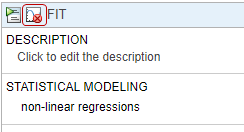
Определите отображение между компонентами модели и столбцами данных из экспериментальных данных. В частности, сопоставьте CentralConc с видами моделей, Drug в центральном отсеке (Central.Drug). Точно так же сопоставьте PeripheralConc с Peripheral.Drug.
Сопоставьте TumorWeight с [Tumor Growth Model].tumor_weight.
Отобразите Dose столбец на Central.Drug, чтобы указать, что виды Drug в Central отсеке дозируются.
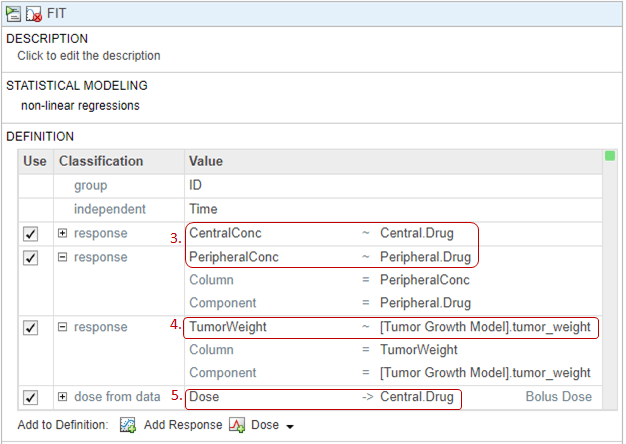
Определите параметры модели для оценки в таблице Estimated Parameters. Дважды кликните пустую камеру в Estimated Parameters столбце и введите k1. Приложение показывает компоненты модели с совпадающими именами. Выберите k1 из списка.
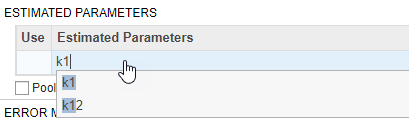
По умолчанию параметр логарифмически преобразуется, как обозначено log преобразования. Можно изменить преобразование на отсутствие преобразования none, probit, или logit преобразование. В данном примере сохраните преобразование журнала по умолчанию, потому что это часто улучшает сходимость. Значение Initial Untransformed Value автоматически устанавливается на значение модели 0,5.
Заставьте биологические параметры оставаться положительными путем определения Untransformed Lower Bound и Untransformed Upper Bound как 1e-5 и 10, соответственно.
Точно так же добавьте следующие параметры: Cl_Central, L0, L1, k12 и k21.
Выберите Pooled fit, чтобы оценить один набор параметров для всех пациентов (население подгонки). Если вы не выбираете Pooled fit, приложение оценивает один набор параметров для каждого пациента (индивидуальная подгонка).
Модель ошибки по умолчанию является моделью постоянной ошибки. SimBiology поддерживает постоянные, пропорциональные, экспоненциальные и комбинированные модели ошибок. Для получения дополнительной информации см. «Модели ошибок». На данный момент используйте модель постоянной ошибки.
Оставьте оставшуюся часть параметров модели без изменений. Эти настройки
Метод оценки - метод по умолчанию является lsqnonlin если у вас есть Optimization Toolbox. Если вы этого не делаете, приложение использует fminsearch.
Для получения дополнительной информации смотрите Поддерживаемые методы оценки параметра в SimBiology.
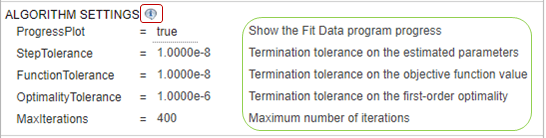
Настройки алгоритма - наиболее распространенные опции для метода оценки. Щелкните, чтобы развернуть раздел и увидеть опции. Чтобы просмотреть описание каждой опции, щелкните значок информации справа от заголовка.
Расширенные настройки алгоритма - Расширенные настройки для метода оценки. По умолчанию таблица пуста.
После установки опций модели можно запустить Fit шаг.
В верхней части Fit шага нажмите кнопку Run this program step.
По умолчанию Fit шаг показывает прогресс оценки параметра на отдельном рисунке. График прогресса показывает текущий статус оценки параметра и подгонки показателей качества, таких как журнал вероятность. Для получения дополнительной информации смотрите График прогресса.
График прогресса показывает, что подгонка сходилась. График прогресса можно закрыть.
Если вы используете fminsearch, подгонка может не сходиться из-за достижения максимального количества итераций. Можно увеличить MaxIter в Algorithm Settings, но в целях этого примера можно продолжить выполнение шагов, не делая этого.
Когда оценка параметра завершена, результаты подгонки показаны в новом листе данных. Таблица данных содержит оценки параметров и другую информацию, связанную с показателями качества соответствия, такими как AIC и BIC, которые могут быть полезны для сравнения эффективности различных моделей ошибок.
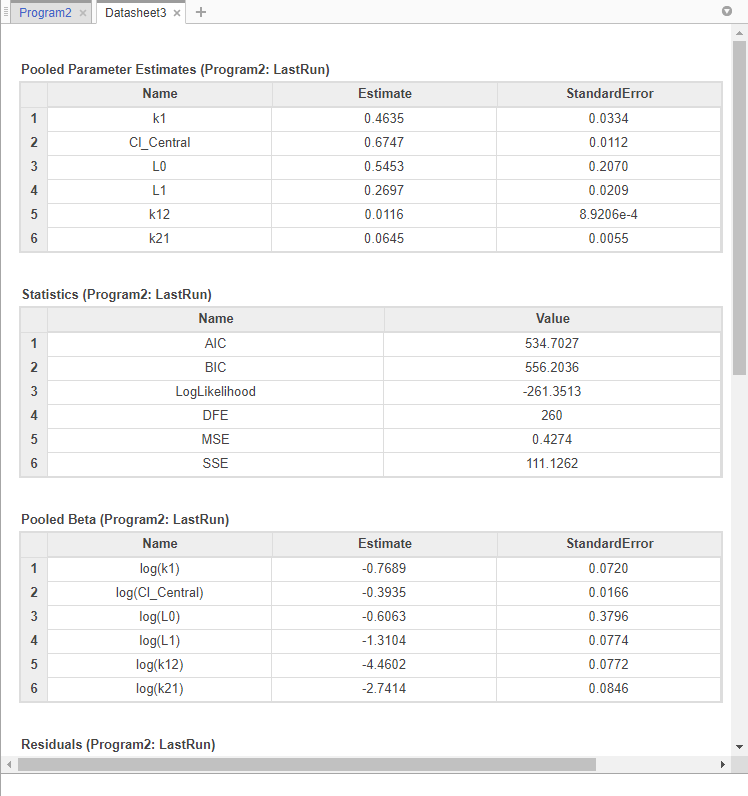
В дополнение к статистике качества можно также просмотреть различные графики подгонки, такие как фактические и предсказанные графики и графики остаточного распределения.
На панели Browser разверните раздел Program2 > LastRun, который содержит results и simdataI. results содержит предполагаемые значения параметров и статистику соответствия. simdataI содержит моделируемые ответы модели для каждого индивидуума (пациента или группы), используя оцененные значения параметров.
Нажмите results. Доступные графики подгонки автоматически перечисляются в разделе Plot на вкладке Home. Затем выберите Act vs Pred из списка.
Фактический график от предсказанного появляется на отдельной вкладке. Предсказанные отклики строятся на оси x, а наблюдаемые (экспериментальные) отклики - на оси y.
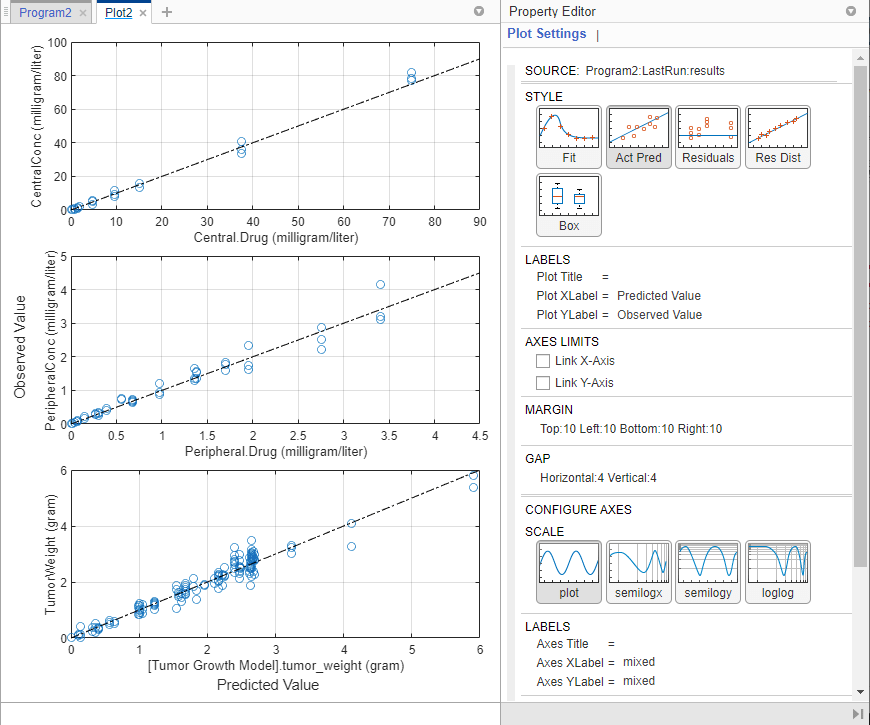
Можно изменить график на другие поддерживаемые графики, выбрав один из графиков из Style раздела Property Editor. Если требуется создать график на отдельной вкладке и не требуется повторно использовать существующую вкладку графика, выберите график из раздела Plot на вкладке Home.
Измените график на график остаточного распределения путем выбора Res Dist в Style разделе.
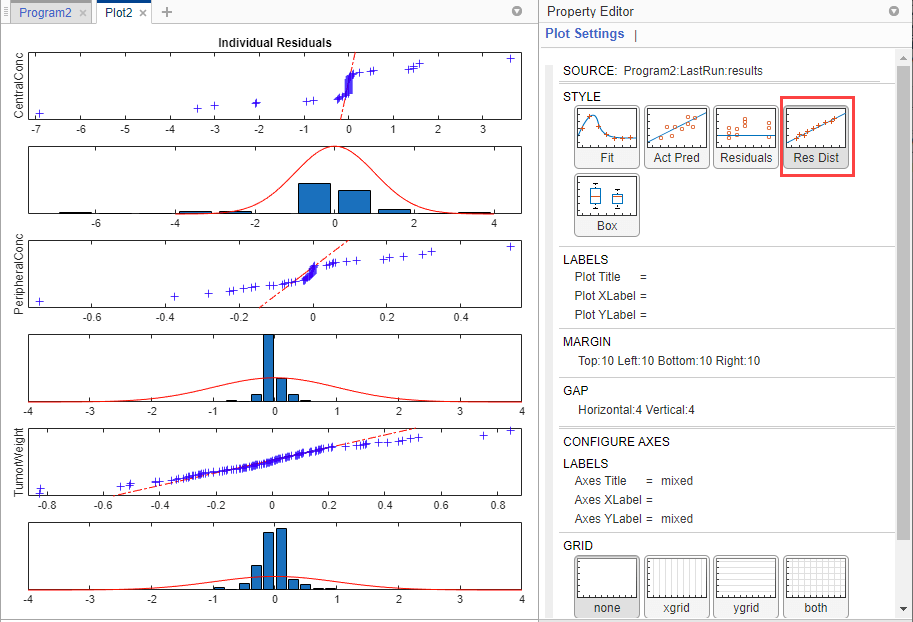
График показывает, распределены ли обычно остатки для каждого отклика. В идеальном графике нормальной вероятности невязок остатки выстраиваются вдоль диагональной линии через график, а гистограммы указывают на нормальную подгонку. Однако из графика невязки для всех трех откликов, особенно CentralConc и PeripheralConc, кажется, не распределены нормально. Это может быть показанием того, что предположение модели постоянной ошибки неправильно.
Следующие шаги показывают, как изменить модель ошибки на экспоненциальную модель ошибки, чтобы снова подогнать данные и сравнить статистику соответствия для двух различных моделей ошибки.
Сохранение результатов подгонки. Прежде чем подгонка данных снова с помощью экспоненциальной модели ошибки, сохраните результат модели постоянной ошибки в отдельной папке. В противном случае программа по умолчанию перезаписывает результаты из папки LastRun каждый раз, когда вы запускаете подгонка.
Щелкните правой кнопкой мыши LastRun папку программы подгонки на панели Browser.
Выберите Save Data.
В диалоговом окне Save Data введите fit_constant как имя данных.
Перезапустите подгонку с экспоненциальной моделью ошибок. После сохранения данных можно перезапустить программу подгонки с другой моделью ошибки.
Вернитесь к программе подгонки, щелкнув вкладку Program2. В Error Model разделе выберите exponential.

В верхней части Fit шага нажмите кнопку Run this program step.

Закройте график прогресса после завершения подгонки.
Если вы закрыли предыдущую таблицу данных (Datasheet3), содержащий статистику подгонки из предыдущего подгонки, снова откройте таблицу данных. На панели Browser разверните раздел Program2 > fit_constant. Затем дважды кликните Datasheet3.
Из папки LastRun перетащите results в Datasheet3. Новые столбцы (Program2_LastRun), содержащие последние результаты подгонки, добавляются рядом с предыдущими результатами подгонки (Program2_fit_constant).
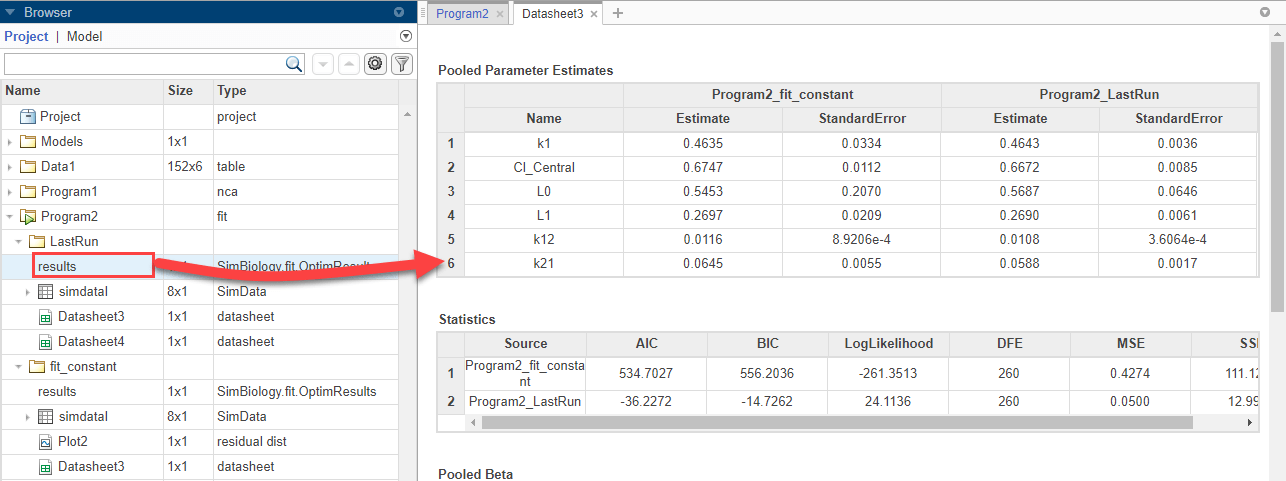
В Statistics таблице сравниваются измерения качества аппроксимации. Из сравнения, и AIC, и BIC подгонки, используя экспоненциальную модель ошибки, меньше, чем у предыдущей подгонки. Это указывает, что экспоненциальная модель ошибки подходит к данным лучше, чем постоянная модель ошибки. Большой журнал вероятность экспоненциальной модели ошибки также указывает, что это лучшая подгонка.
Далее рассмотрим график остаточного распределения. Щелкните results из папки LastRun. Затем щелкните Residual Dist из раздела Plot на вкладке Home.
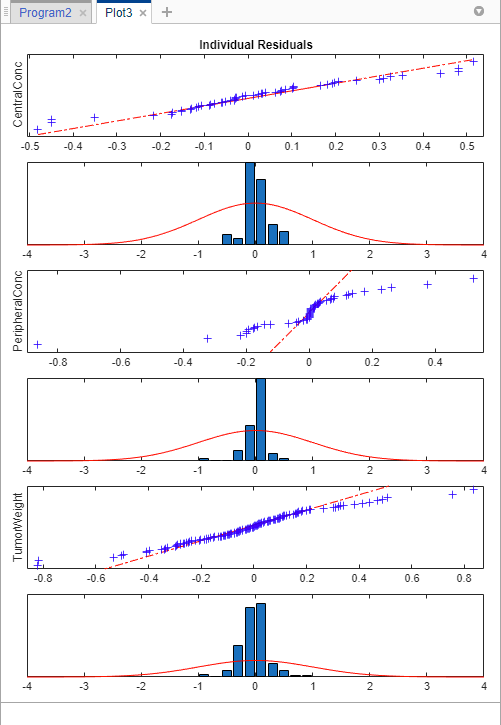
По сравнению с остаточными распределениями модели постоянной ошибки, остаточные распределения из экспоненциальной модели ошибки выглядят более нормально, что указывает на то, что экспоненциальная модель ошибки лучше подходит для данных.
Другой способ оценки качества результатов подгонки - вычислить 95% доверительные интервалы для предполагаемых параметров и предсказаний модели - то есть симуляции модели с использованием предполагаемых параметров. Этот шаг требует Statistics and Machine Learning Toolbox™.
Вернитесь к программе подгонки. Щелкните значок (+) в верхнем левом углу и выберите Confidence Interval. После Confidence Interval шага появляется Fit шаг.
В верхней части Confidence Interval шага отключите автогенерацию графиков, щелкнув значок plot. Для Parameter Confidence Intervals и Prediction Confidence Intervals используйте метод по умолчанию gaussian и 95% доверительный уровень. Нажмите кнопку Run this program step, чтобы вычислить доверительные интервалы.

Для доверительных интервалов параметров поддерживаемые методы являются гауссовыми, profileLikelihood и bootstrap.
Для доверительных интервалов предсказания поддерживаемые методы являются гауссовыми и загрузочными.
После завершения результаты сохраняются как parameterCI и predictionCI в LastRun папке программы. parameterCI содержит 95% доверительных интервалов для предполагаемых параметров. predictionCI содержит 95% доверительных интервалов для предсказаний модели.
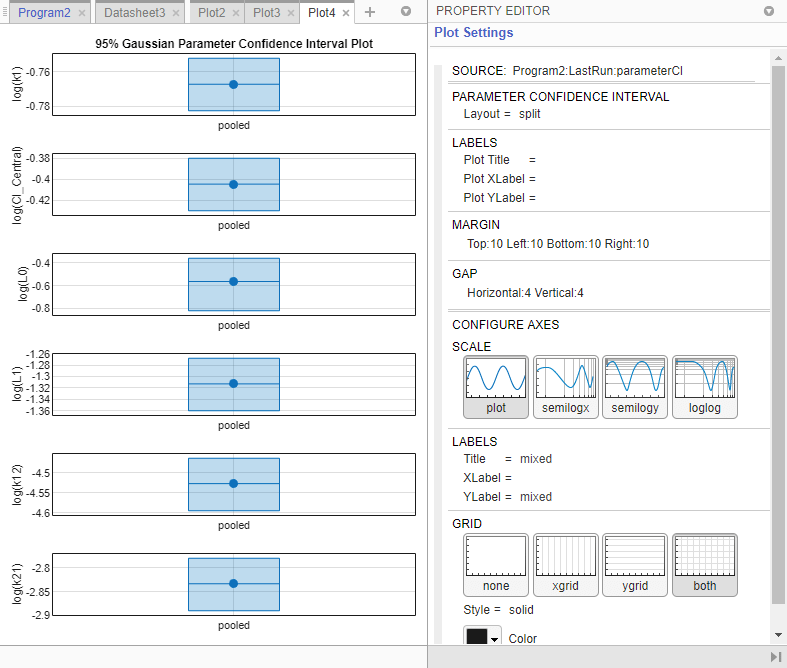
Постройте график 95% доверительных интервалов для предполагаемых параметров. Щелкните parameterCI на панели Browser и выберите Confidence в Plot разделе.
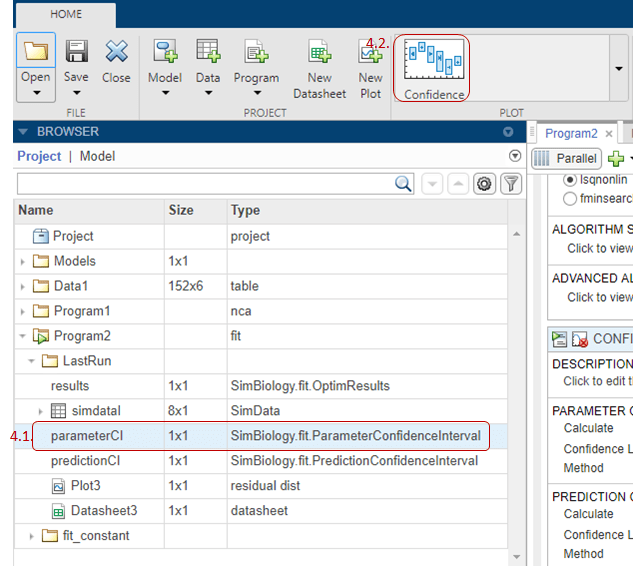
Интервал доверия для каждого оцененного параметра показан на новом графике. График указывает на успешный расчет доверительных интервалов для всех предполагаемых параметров.
В зависимости от результата (статуса) оценки доверительного интервала, приложение строит графики результатов по-разному.
Если состояние оценки доверительного интервала является успешным (как на вышеописанном графике), приложение использует первый цвет по умолчанию (синий), чтобы построить график линии и центрированную точку для каждой оценки параметра. Приложение также строит график для указания доверительных интервалов.
Если состояние ограничено или оценивается, приложение использует второй цвет по умолчанию (красный) и строит график линии, центрированной точки и рамки, чтобы указать доверительные интервалы.
Если состояние не оценивается, приложение строит графики только линии и центрированного креста красного цвета.
Если существуют какие-либо преобразованные параметры с оценочными значениями, которые равны 0 (для log преобразование) и 0 или 1 (для probit или logit transform), никакое доверие интервалы не строятся для этих оценок параметра.
Для получения дополнительной информации об определениях различных статусов, смотрите Статус оценки доверительного интервала параметра.
Можно также изменить Layout графика в Plot Settings.
The 'split' размещение отображает доверительный интервал для каждой оценки параметра на отдельной оси.
The 'grouped' В размещении отображаются все доверительные интервалы на одной оси, сгруппированные по оценкам параметров. Каждый оценочный параметр разделяется вертикальной черной линией.
В обоих случаях границы параметров, заданные в исходной подгонке, отмечены квадратными скобками. Приложение использует вертикальные пунктирные линии, чтобы сгруппировать доверительные интервалы оценок параметров, которые были вычислены в общей подгонке.
Точно так же постройте график 95% доверительных интервалов для предсказаний модели. Щелкните predictionCI на панели Browser и выберите Confidence в Plot разделе.
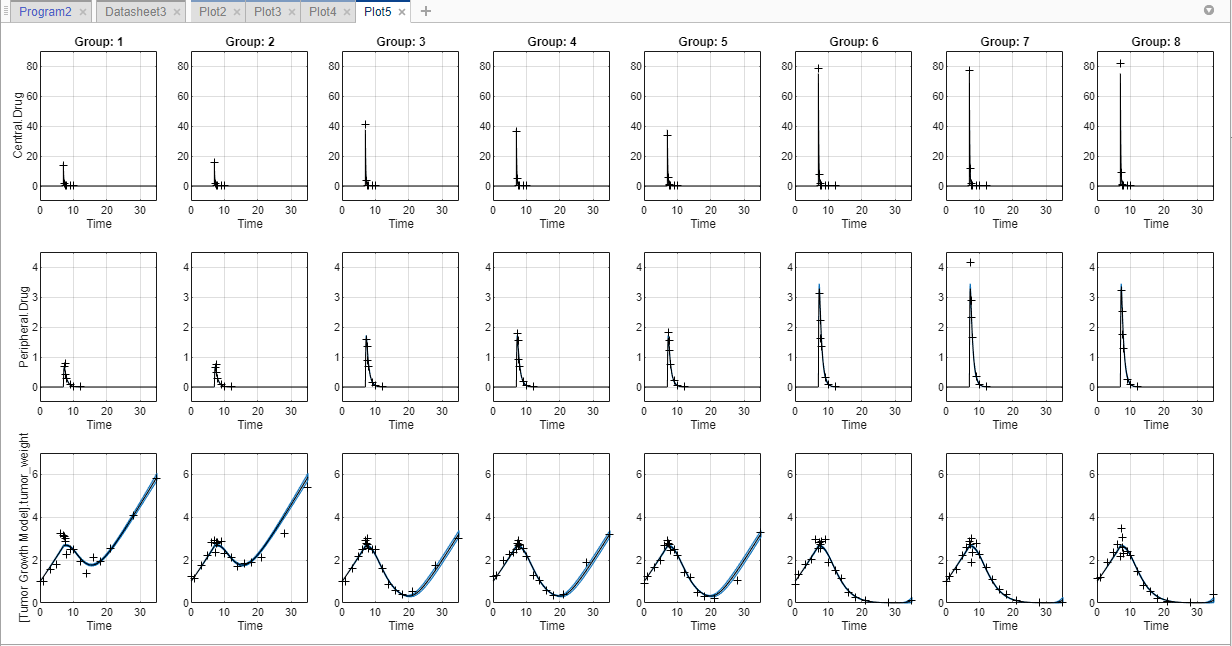
На этом графике доверительный интервал для каждой группы строится в отдельном столбце, и каждый ответ строкой. График указывает на успешный расчет доверительных интервалов.
Поведение графического изображения отличается в зависимости от результата (статуса) вычисления доверительного интервала.
Если состояние ограничено или не оценивается, приложение использует второй цвет по умолчанию (красный), чтобы построить график доверительных интервалов.
В противном случае приложение использует первый цвет по умолчанию (синий) и строит графики доверительных интервалов как затененные области (как на вышеописанном графике).
Для получения дополнительной информации смотрите Расчет Доверительного Интервала Гауссова для Предсказаний Модели и Расчет Доверительного Интервала Bootstrap.
Когда оценка параметра завершена, можно построить график результатов симуляции поверх экспериментальных данных. Есть, как правило, два способа получить график.
Первый подход состоит в том, чтобы использовать график Fit по умолчанию, который показывает подгоняемые результаты по экспериментальным данным для каждой группы.
Щелкните results из LastRun папки программы подгонки.
Выберите Fit в Plot разделе на вкладке Home.
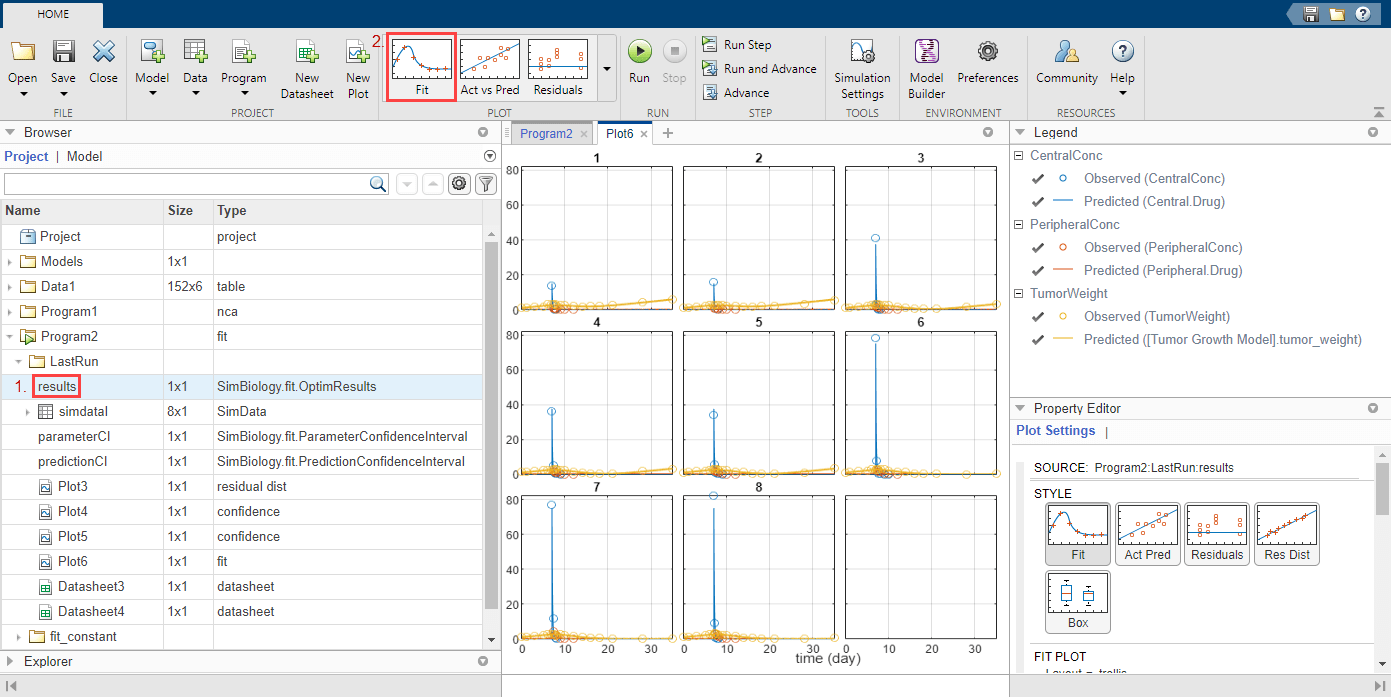
Второй подход более настроен и задействован, но у вас есть больше контроля над тем, как выглядит график. Следующие шаги показывают, как построить графики роста опухоли всех групп на одних и тех же осях, но использовать различные стили линии для экспериментальных данных и данных моделирования.
Нажмите simdataI в LastRun папке программы подгонки.
Щелкните time из Plot раздела на вкладке Home. Вы получаете следующий график.
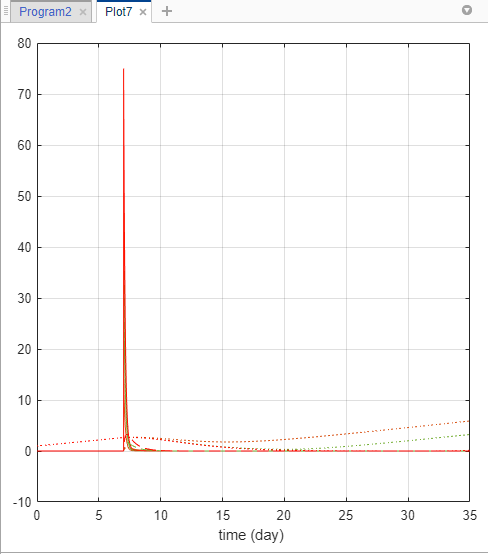
Перетащите Data1 (экспериментальные данные) к графику.
В Property Editor графика выберите несколько элементов (Ctrl
+ нажатие кнопки)
и удалите следующие ответы: Central.Drug, Peripheral.Drug от Program2 и CentralConc и PeripheralConc от Data1.
Нажмите на tumor_weight ответ от Program2 и нажмите Create Set.
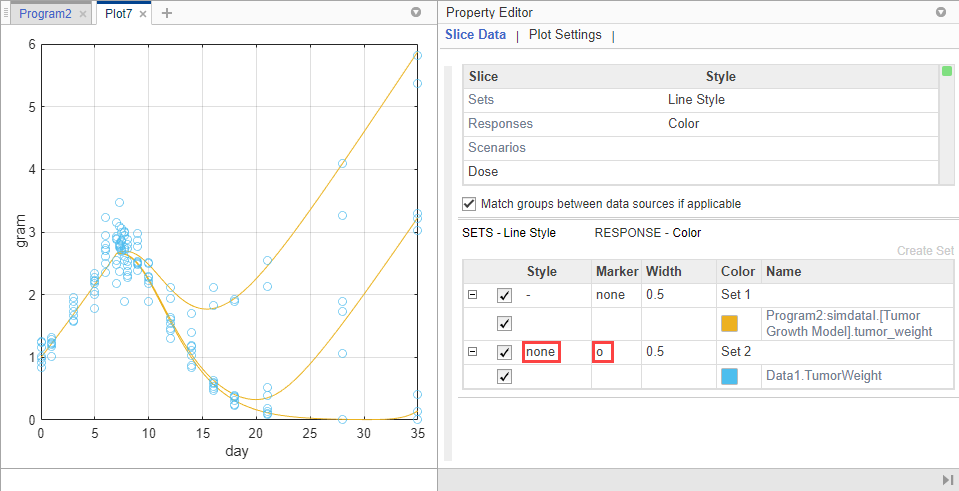
В таблице Slice измените Style Sets, Responses и Scenarios следующим образом. Чтобы не использовать стиль для Scenarios, выберите пустой.

Установите Style экспериментальных данных (Data1.TumorWeight) к none. Установите Marker равным o.
После подбора кривой к данным можно установить значения модели на оценки параметров и выполнить другие анализы. Например, можно узнать важные параметры модели с помощью анализа чувствительности и изменить чувствительные параметры, чтобы исследовать изменчивость модели при помощи виртуальных пациентов.
[1] Simeoni, M., P. Magni, C. Cammia, G. De Nicolao, V. Croci, E. Pesenti, M. Germani, I. Poggesi, and M. Rocchetti. 2004. Прогнозирующее фармакокинетико-фармакодинамическое моделирование кинетики роста опухоли на ксенотрансплантатных моделях после введения противоопухолевых агентов. Исследования рака. 64:1094-1101.