Постройте свойства последовательности аминокислот
proteinpropplot (SeqAA)
proteinpropplot(SeqAA,
...'PropertyTitle', PropertyTitleValue, ...)
proteinpropplot(SeqAA,
...'Startat', StartatValue, ...)
proteinpropplot(SeqAA,
...'Endat', EndatValue, ...)
proteinpropplot(SeqAA,
...'Smoothing', SmoothingValue, ...)
proteinpropplot(SeqAA,
...'EdgeWeight', EdgeWeightValue, ...)
proteinpropplot(SeqAA,
...'WindowLength', WindowLengthValue, ...)
SeqAA | Последовательность аминокислот. Введите любое следующее:
|
PropertyTitleValue | Вектор символов или строка, которая задает свойство построить. Значением по умолчанию является Hydrophobicity (Kyte & Doolittle). Чтобы отобразить список свойств построить, введите пустой символьный вектор или пустую строку для PropertyTitleValue. Например, тип:proteinpropplot(sequence, 'propertytitle', '') Совет К ссылкам доступа для свойств просмотрите |
StartatValue | Целое число, которое задает начальную точку для графика от конца N-терминала последовательности аминокислот SeqAA. Значением по умолчанию является 1. |
EndatValue | Целое число, которое задает конечную точку для графика от конца N-терминала последовательности аминокислот SeqAA. Значением по умолчанию является длина (. |
SmoothingValue | Вектор символов или строка задавание метода сглаживания. Выбор:
|
EdgeWeightValue | Значение, которое задает вес ребра, используемый для методов линейного и экспоненциального сглаживания. Уменьшение этого значения подчеркивает peaks в графике. Выбором является любое значение ≥0 и ≤1. Значением по умолчанию является 1. |
WindowLengthValue | Целое число, которое задает длину окна для метода сглаживания. Увеличение этого значения дает более сглаженный график, который показывает меньше детали. Значением по умолчанию является 11. |
proteinpropplot ( отображает график гидрофобности (Kyte и Doolittle, 1982) остатков в последовательности SeqAA)SeqAA.
proteinpropplot ( вызовы SeqAAPropertyName ', PropertyValue, ...)proteinpropplot с дополнительными свойствами, которые используют имя свойства / пары значения свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и нечувствительный к регистру. Это имя свойства / пары значения свойства следующие:
proteinpropplot( задает свойство построить для последовательности аминокислот SeqAA,
...'PropertyTitle', PropertyTitleValue, ...)SeqAA. Значением по умолчанию является Hydrophobicity (Kyte & Doolittle). Чтобы отобразить список возможных свойств построить, введите пустой символьный вектор или пустую строку для PropertyTitleValue. Например, тип:
proteinpropplot(sequence, 'propertytitle', '')
Совет
К ссылкам доступа для свойств просмотрите proteinpropplot файл.
proteinpropplot( задает начальную точку для графика от конца N-терминала последовательности аминокислот SeqAA,
...'Startat', StartatValue, ...)SeqAA. Значением по умолчанию является 1.
proteinpropplot( задает конечную точку для графика от конца N-терминала последовательности аминокислот SeqAA,
...'Endat', EndatValue, ...)SeqAA. Значением по умолчанию является длина (.SeqAA)
proteinpropplot( задает метод сглаживания. Выбор: SeqAA,
...'Smoothing', SmoothingValue, ...)
linear (значение по умолчанию)
exponential
lowess
proteinpropplot( задает вес ребра, используемый для методов линейного и экспоненциального сглаживания. Уменьшение этого значения подчеркивает peaks в графике. Выбором является любое значение SeqAA,
...'EdgeWeight', EdgeWeightValue, ...)≥0 и ≤1. Значением по умолчанию является 1.
proteinpropplot( задает длину окна для метода сглаживания. Увеличение этого значения дает более сглаженный график, который показывает меньше детали. Значением по умолчанию является SeqAA,
...'WindowLength', WindowLengthValue, ...)11.
Используйте getpdb функция, чтобы получить последовательность белка.
prion = getpdb('1HJM', 'SEQUENCEONLY', true);Постройте гидрофобность (Kyte и Doolittle, 1982) остатков в последовательности.
proteinpropplot(prion)
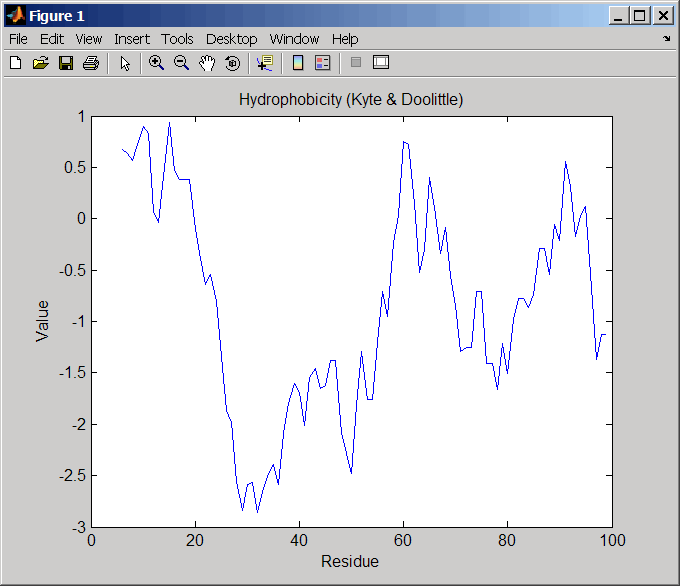
Используйте getgenpept функция, чтобы получить последовательность белка.
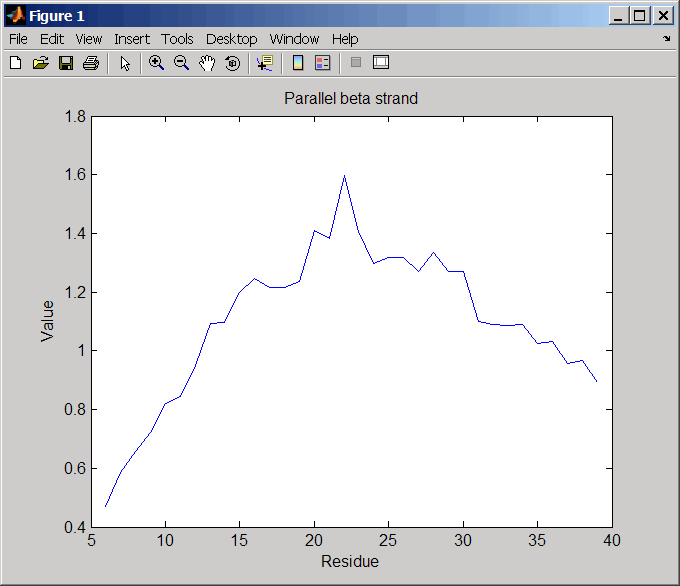
s = getgenpept('aad50640');Постройте конформационную настройку параллельной бета скрутке для остатков в последовательности.
proteinpropplot(s,'propertytitle','Parallel beta strand')
[1] Живот, J., и Doolittle, R.F. (1982). Простой метод для отображения символа водолечебницы белка. J молекулярная масса Biol 157 (1), 105–132.
aacount | atomiccomp | molviewer | molweight | pdbdistplot | plotyy | proteinplot | ramachandran | seqviewer