Выполните корректировку фона GC Rustive Multi-array Average (GCRMA) для данных на уровне зондов микрочипов Affymetrix с использованием информации о последовательности
PMMatrix_Adj = gcrmabackadj(PMMatrix, MMMatrix, AffinPM, AffinMM)
[PMMatrix_Adj, nsbStruct] = gcrmabackadj(PMMatrix, MMMatrix, AffinPM, AffinMM)
... = gcrmabackadj( ...'OpticalCorr', OpticalCorrValue, ...)
... = gcrmabackadj( ...'CorrConst', CorrConstValue, ...)
... = gcrmabackadj( ...'Method', MethodValue, ...)
... = gcrmabackadj( ...'TuningParam', TuningParamValue, ...)
... = gcrmabackadj( ...'AddVariance', AddVarianceValue, ...)
... = gcrmabackadj( ...'GSBCorr', GSBCorrValue, ...)
... = gcrmabackadj( ...'Showplot', ShowplotValue, ...)
... = gcrmabackadj( ...'Verbose', VerboseValue, ...)
PMMatrix | Матрица значений интенсивности, где каждая строка соответствует зонду совершенного соответствия (PM), а каждый столбец соответствует файлу Affymetrix ® CEL. (Каждый файл CEL генерируется из отдельной микросхемы. Все микросхемы должны быть одного типа .) Совет Вы можете использовать
|
MMMatrix | Матрица значений интенсивности, где каждая строка соответствует зонду несоответствия (MM), а каждый столбец соответствует файлу Affymetrix CEL. (Каждый файл CEL генерируется из отдельной микросхемы. Все микросхемы должны быть одного типа.) Совет Вы можете использовать
|
AffinPM | Вектор столбца сродства зонда PM, например, возвращенный affyprobeaffinities функция. Каждая строка соответствует зонду. |
AffinMM | Вектор столбца сродства зонда MM, например, возвращенный affyprobeaffinities функция. Каждая строка соответствует зонду. |
OpticalCorrValue | Управляет использованием оптической фоновой коррекции значений интенсивности зондов PM и MM в PMMatrix и MMMatrix. Варианты: true (по умолчанию) или false. |
CorrConstValue | Значение, определяющее корреляционную константу rho для логарифмической фоновой интенсивности для каждой пары зондов PM/MM. Варианты - это любое значение ≥ 0 и ≤ 1. По умолчанию: 0.7. |
MethodValue | Символьный вектор или строка, определяющая метод оценки сигнала. Варианты: 'MLE', более быстрый, специальный метод оценки максимального правдоподобия или 'EB', более медленный, более формальный, эмпирический метод Байеса. По умолчанию: 'MLE'. |
TuningParamValue | Значение, указывающее параметр настройки, используемый методом оценки. Этот параметр настройки устанавливает нижнюю границу значений сигнала с положительной вероятностью. Выбор - это положительное значение. По умолчанию: 5 (MLE) или 0.5 (EB).Совет Информацию об определении параметров для этого параметра см. в Wu et al., 2004. |
AddVarianceValue | Управляет добавлением дисперсии сигнала к весовой функции для сглаживания нижнего фронта сигнала. Варианты: true или false (по умолчанию). |
GSBCorrValue | Указывает, следует ли выполнять коррекцию геноспецифического связывания (GSB) с использованием данных о сродстве зондов. Варианты: true (по умолчанию) или false. Если информация о сходстве зонда отсутствует, это свойство игнорируется. |
ShowplotValue | Управляет отображением графика, показывающего log2 значений интенсивности зонда из указанного столбца (микросхемы) в MMMatrix, в сравнении со сродством зонда в AffinMM. Варианты: true, false, или I, целое число, указывающее столбец в MMMatrix. Если установлено значение true, первый столбец в MMMatrix выводится на печать. Значение по умолчанию:
|
VerboseValue | Управляет отображением отчета о ходе выполнения, показывающего количество каждой микросхемы по мере ее завершения. Варианты: true (по умолчанию) или false. |
PMMatrix_Adj | Матрица скорректированных по фону значений интенсивности ТЧ (идеального соответствия). |
nsbStruct | Структура, содержащая неспецифические параметры фона связывания, оцененные по интенсивности и аффинности зондов в массиве Affymetrix GeneChip ® .
|
PMMatrix_Adj = gcrmabackadj(PMMatrix, MMMatrix, AffinPM, AffinMM)PMMatrix_Adjматрица скорректированных по фону значений интенсивности ТЧ (идеального соответствия).
Примечание
Если AffinPM и AffinMM данные недоступны, вы по-прежнему можете использовать gcrmabackadj путем ввода пустых векторов столбцов для обоих этих входов в синтаксисе.
[ прибыль PMMatrix_Adj, nsbStruct] = gcrmabackadj(PMMatrix, MMMatrix, AffinPM, AffinMM)nsbStructструктура, содержащая неспецифические параметры фона связывания, оцененные по интенсивности и аффинности зондов в массиве Affymetrix GeneChip. nsbStruct включает следующие поля:
sigma
mu_pm
mu_mm
... = gcrmabackadj( ...' требования PropertyName', PropertyValue, ...)gcrmabackadj с необязательными свойствами, использующими пары имя/значение свойства. Можно указать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и не учитывать регистр. Эти пары имя/значение свойства следующие:
... = gcrmabackadj( ...'OpticalCorr', управляет использованием оптической фоновой коррекции на значениях интенсивности зондов PM и MM в OpticalCorrValue, ...)PMMatrix и MMMatrix. Варианты: true (по умолчанию) или false.
... = gcrmabackadj( ...'CorrConst', задает константу корреляции rho для интенсивности log-фона для каждой пары зондов PM/MM. Варианты - это любое значение CorrConstValue, ...)≥ 0 и ≤ 1. По умолчанию: 0.7.
... = gcrmabackadj( ...'Method', определяет метод оценки сигнала. Варианты: MethodValue, ...)MLE, более быстрый, специальный метод оценки максимального правдоподобия или EB, более медленный, более формальный, эмпирический метод Байеса. По умолчанию: MLE.
... = gcrmabackadj( ...'TuningParam', задает параметр настройки, используемый методом оценки. Этот параметр настройки устанавливает нижнюю границу значений сигнала с положительной вероятностью. Выбор - это положительное значение. По умолчанию: TuningParamValue, ...)5 (MLE) или 0.5 (EB).
Совет
Информацию об определении параметров для этого параметра см. в Wu et al., 2004.
... = gcrmabackadj( ...'AddVariance', управляет добавлением дисперсии сигнала к весовой функции для сглаживания нижнего фронта сигнала. Варианты: AddVarianceValue, ...)true или false (по умолчанию).
... = gcrmabackadj( ...'GSBCorr', указывает, следует ли выполнять коррекцию специфического связывания генов (GSB) с использованием данных о сродстве зондов. Варианты: GSBCorrValue, ...)true (по умолчанию) или false. Если информация о сходстве зонда отсутствует, это свойство игнорируется.
... = gcrmabackadj( ...'Showplot', управляет отображением графика, показывающего log2 значений интенсивности зонда из указанного столбца (микросхемы) в ShowplotValue, ...)MMMatrix, в сравнении со сродством зонда в AffinMM. Варианты: true, false, или I, целое число, указывающее столбец в MMMatrix. Если установлено значение true, первый столбец в MMMatrix выводится на печать. Значение по умолчанию:
false - Когда указаны возвращаемые значения.
true - Когда возвращаемые значения не указаны.
... = gcrmabackadj( ...'Verbose', управляет отображением отчета о ходе выполнения, показывающего количество каждой микросхемы по мере ее завершения. Варианты: VerboseValue, ...)true (по умолчанию) или false.
Загрузите MAT-файл, включенный в программное обеспечение Bioinformatics Toolbox™, который содержит данные Affymetrix из исследования рака предстательной железы. Переменные в MAT-файле включают seqMatrixматрица, содержащая информацию о последовательности для PM-зондов, pmMatrix и mmMatrix, матрицы, содержащие значения интенсивности зонда PM и MM, и probeIndicesвектор столбца, содержащий информацию индексации зонда.
load prostatecancerrawdataВычислите сродства зондов Affymetrix PM и MM по их последовательностям и интенсивностям зондов MM.
[apm, amm] = affyprobeaffinities(seqMatrix, mmMatrix(:,1),... 'ProbeIndices', probeIndices);
Выполните корректировку фона GCRMA для данных уровня зонда микрочипа Affymrix, создав матрицу скорректированных в фоновом режиме значений интенсивности ТЧ. Также отобразите график, показывающий log2 значений интенсивности зонда из столбца 3 (микросхема 3) в mmMatrix, в сравнении со сродством зонда в amm.
pms_adj = gcrmabackadj(pmMatrix, mmMatrix, apm, amm, 'showplot', 3);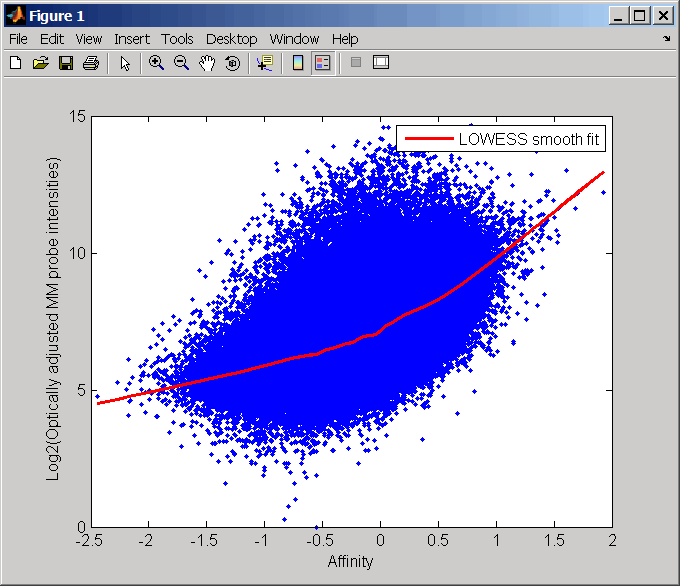
Снова выполните корректировку фона GCRMA, используя более медленный, более формальный, эмпирический метод Байеса.
pms_adj2 = gcrmabackadj(pmMatrix, mmMatrix, apm, amm, 'method', 'EB');
prostatecancerrawdata.mat файл, используемый в этом примере, содержит данные Best et al., 2005.
[1] Ву, З., Иризарри, Р.А., Джентльмен, Р., Мурильо, Ф.М., и Спенсер, Ф. (2004). Основанная на модели корректировка фона для массивов олигонуклеотидной экспрессии. Журнал Американской статистической ассоциации 99 (468), 909-917.
[2] Ву, З. и Иризарри, Р.А. (2005). Стохастические модели, вдохновленные теорией гибридизации для коротких олигонуклеотидных массивов. Работа РЕКОМБ в 2004 году. J Comput Biol. 12 (6), 882-93.
[3] Ву, З. и Иризарри, Р.А. (2005). Статистическая основа для анализа данных на уровне зондов микрочипов. Университет Джона Хопкинса, рабочие документы по биостатистике 73.
[4] Ву, З. и Иризарри, Р.А. (2003). Основанная на модели корректировка фона для массивов олигонуклеотидной экспрессии. Семинар RSS по экспрессии генов, Уай, Англия, http://biosun01.biostat.jhsph.edu/%7Eririzarr/Talks/gctalk.pdf.
[5] Абд Раббо, Н. А., и Баракат, Х. М. (1979). Проблемы оценки в двумерном логнормальном распределении. Индиец Дж. Pure Appl. Math 10 (7), 815-825.
[6] Best, C.J.M., Gillespie, J.W., Yi, Y., Chandramouli, G.V.R., Perlmutter, M.A., Собирается, Я., Эриксон, Х. С., Георгевич, Л., Тангреа, М. А., Дюрей, П.Х., Гонсалес, С., Веласко, А., Линехан, В.М., Матусик, Р.Дж., Прайс, Д.К., Фигг, В.Д., Эммерт-Бак, М.Р., и Чуакки, Р.Ф. (2005). Молекулярные изменения при первичном раке предстательной железы после терапии андрогенной абляцией. Клинические исследования рака 11, 6823-6834.
affygcrma | affyprobeseqread | affyread | celintensityread | probelibraryinfo