Создание графика зависимости интенсивности от коэффициента рассеяния данных микрочипов
mairplot(DataX, DataY)
[Intensity, Ratio] = mairplot(DataX, DataY)
[Intensity, Ratio, H] = mairplot(DataX, DataY)
... = mairplot(..., 'Type', TypeValue, ...)
... = mairplot(..., 'LogTrans', LogTransValue, ...)
... = mairplot(..., 'FactorLines', FactorLinesValue, ...)
... = mairplot(..., 'Title', TitleValue, ...)
... = mairplot(..., 'Labels', LabelsValue, ...)
... = mairplot(..., 'Normalize', NormalizeValue, ...)
... = mairplot(..., 'LowessOptions', LowessOptionsValue, ...)
... = mairplot(..., 'Showplot', ShowplotValue, ...)
... = mairplot(..., 'PlotOnly', PlotOnlyValue, ...)
DataX, DataY | Объект DataMatrix или вектор значений экспрессии генов, где каждая строка соответствует гену. Например, в двухцветном эксперименте с микрочипами DataX могут быть значениями интенсивности cy3 и DataY могут быть значениями интенсивности cy5. |
TypeValue | Символьный вектор или строка, указывающая тип графика. Варианты: 'IR' (график log10 произведения DataX и DataY интенсивности в сравнении с log2 соотношений интенсивности) или 'MA' (графики (1/2) log2 произведения DataX и DataY интенсивности в сравнении с log2 соотношений интенсивности). По умолчанию: 'IR'. |
LogTransValue | Управляет преобразованием данных в X и Y от естественной шкалы к шкале log2. Набор LogTransValue кому false, если данные уже имеют масштаб log2. По умолчанию: true, что предполагает естественный масштаб данных. |
FactorLinesValue | Добавление линий на график с коэффициентом N изменение. Значение по умолчанию - 2, что соответствует уровню 1 и -1 в масштабе log2.Совет После создания графика можно также изменить факторные линии в интерактивном режиме. |
TitleValue | Символьный вектор или строка, указывающая заголовок для графика. |
LabelsValue | Массив ячеек символьных векторов или строковых векторов, содержащих метки для данных. Если метки определены, то щелчок мыши на точке на графике показывает метку, соответствующую этой точке. |
NormalizeValue | Управляет отображением значений нормализованного отношения с наименьшим числом. Войти true для отображения значений нормализованного отношения с понижением. По умолчанию: false. Совет После создания графика можно также нормализовать данные в окне Печать MAIR (MAIR Plot). |
LowessOptionsValue | Массив ячеек, состоящий из одной, двух или трех пар «имя/значение свойства» в любом порядке, влияющем на нормализацию с наименьшим значением. Варианты пар имя/значение свойства:
Дополнительные сведения о предыдущих парах имя/значение свойства см. в разделе |
ShowplotValue | Управление отображением графика рассеяния. Варианты: true (по умолчанию) или false. |
PlotOnlyValue | Управление отображением графика рассеяния без компонентов интерфейса пользователя. Варианты: Примечание Если установить |
Intensity | Объект или вектор DataMatrix, содержащий значения интенсивности для данных экспрессии гена микрочипа, рассчитанные как:
Примечание Если |
Ratio | Объект или вектор DataMatrix, содержащий отношения данных экспрессии генов микрочипов, рассчитанные как log2(.Примечание Если |
H | Ручка сюжета. |
mairplot( создает график рассеяния, который отображает log10 произведения DataX, DataY)DataX и DataY интенсивности в сравнении с log2 соотношений интенсивности.
[ возвращает значения интенсивности и отношения. Если установить Intensity, Ratio] = mairplot(DataX, DataY)'Normalize' кому trueвозвращаемые значения отношения нормализуются.
[ возвращает дескриптор графика.Intensity, Ratio, H] = mairplot(DataX, DataY)
... = mairplot(..., 'PropertyName', PropertyValue, ...)mairplot с необязательными свойствами, использующими пары имя/значение свойства. Можно указать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и не учитывать регистр. Эти пары имя/значение свойства следующие:
определяет тип печати. Варианты: ... = mairplot(..., 'Type', TypeValue, ...)'IR' (график log10 произведения DataX и DataY интенсивности в сравнении с log2 соотношений интенсивности) или 'MA' (графики (1/2) log2 произведения DataX и DataY интенсивности в сравнении с log2 соотношений интенсивности). По умолчанию: 'IR'.
... = mairplot(..., 'LogTrans', LogTransValue, ...)X и Y от естественной до логарифмической 2 шкалы. Набор LogTransValue кому false, если данные уже имеют масштаб log 2. По умолчанию: true, что предполагает естественный масштаб данных.
... = mairplot(..., 'FactorLines', FactorLinesValue, ...)N
Совет
После создания графика можно также изменить факторные линии в интерактивном режиме.
... = mairplot(..., 'Title', TitleValue, ...)
... = mairplot(..., 'Labels', LabelsValue, ...)
... = mairplot(..., 'Normalize', NormalizeValue, ...)true для отображения значений нормализованного отношения с понижением. По умолчанию: false.
Совет
После создания графика можно также нормализовать данные в окне Печать MAIR (MAIR Plot).
... = mairplot(..., 'LowessOptions', LowessOptionsValue, ...)
'Order', OrderValue
'Robust', RobustValue
'Span', SpanValue
Дополнительные сведения о предыдущих трех парах имя/значение свойства см. в разделе malowess функция.
... = mairplot(..., 'Showplot', ShowplotValue, ...)true (по умолчанию) или false.
... = mairplot(..., 'PlotOnly', PlotOnlyValue, ...)true или false (по умолчанию).
Примечание
Если установить 'PlotOnly' свойство для trueможно по-прежнему отображать метки для точек данных щелчком мыши на точке данных, а также корректировать горизонтальные линии изменения сгиба, перетаскивая линии щелчком мыши.
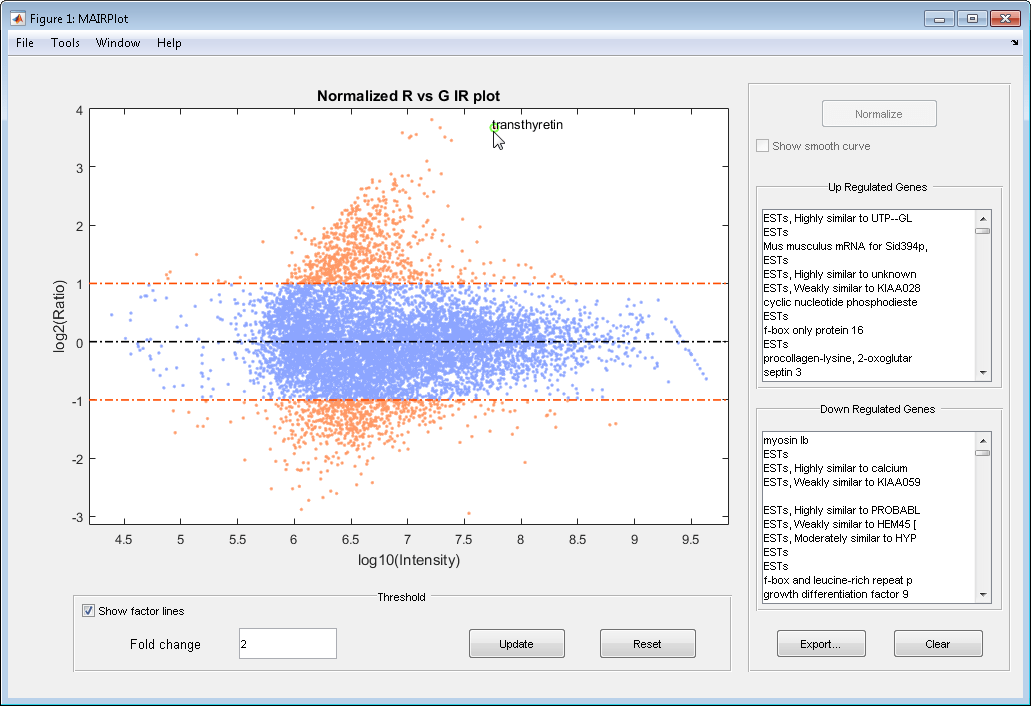
Ниже приведен ИК-график нормированных данных.
Ниже приведен график MA ненормализованных данных.
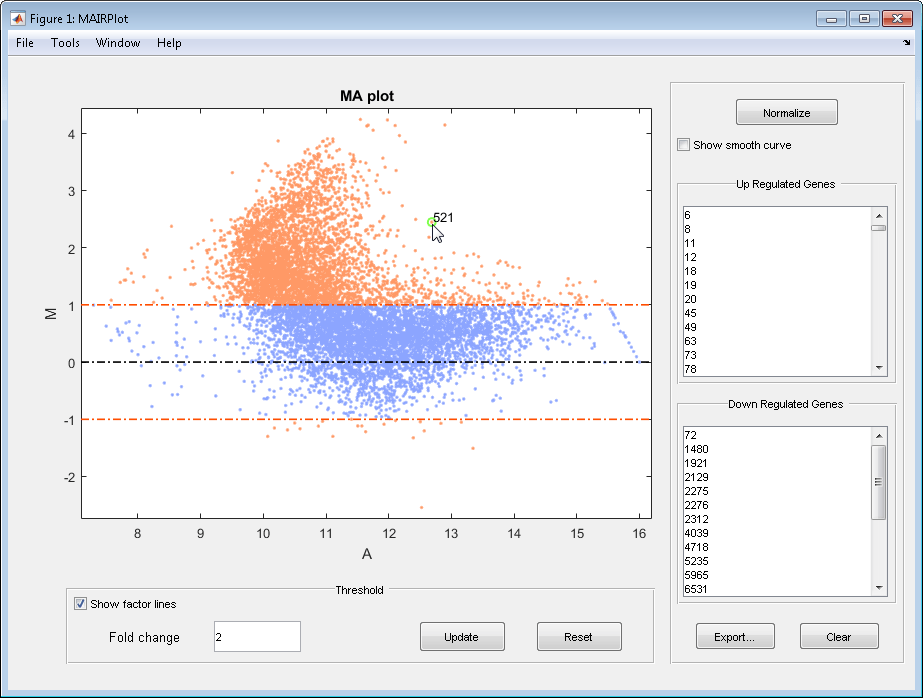
График зависимости интенсивности от отношения рассеяния отображает следующее:
log10 (Интенсивность) против log2 (Отношение) график рассеяния генов.
Две горизонтальные линии сгиба изменяются на уровне сгиба 2, что соответствует соотношению 1 и -1 по шкале log 2 (Ratio). (Линии будут находиться на разных уровнях изменения сгиба, если вы использовали 'FactorLines' собственность.)
Точки данных для генов, которые считаются дифференциально экспрессированными (вне линий изменения сгиба), появляются оранжевым цветом.
После отображения графика зависимости интенсивности от отношения рассеяния можно в интерактивном режиме выполнить следующие действия.
Отрегулируйте горизонтальные линии изменения сгиба, перетащив одну линию щелчком мыши или введя значение в текстовое поле Изменение сгиба (Fold Change), а затем нажав кнопку Обновить (Update).
Отображение меток для точек данных щелчком мыши на точке данных.
Выберите ген из списка Регулируемый вверх или Регулируемый вниз, чтобы выделить соответствующую точку данных на графике. Для выбора нескольких генов нажмите и удерживайте клавишу Ctrl или Shift.
Увеличьте масштаб графика, выбрав меню «Сервис» > «Увеличить» или «Инструменты» > «Уменьшить».
Просмотрите списки значительно повышенных и пониженных генов и при необходимости экспортируйте метки и индексы генов в структуру в рабочей области MATLAB ®, нажав кнопку Экспорт.
Нормализуйте данные, нажав кнопку Нормализовать (Normalize) и выбрав, следует ли отображать нормализованный график в отдельном окне. Если нормализованный график отображается в отдельном окне, флажок Показать гладкую кривую (Show smooth curve) становится доступным на исходном (ненормализованном) графике.
Совет
Чтобы перед нормализацией выбрать другие параметры нормализации нижнего предела, выберите «Сервис» > «Задать параметры нормализации нижнего предела», затем введите параметры в диалоговом окне «Параметры для нижнего предела».
Используйте gprread создание структуры, содержащей данные микрочипов.
maStruct = gprread('mouse_a1wt.gpr');Используйте magetfield функция для извлечения зеленого (cy3) и красного (cy5) сигналов из структуры.
cy5data = magetfield(maStruct,'F635 Median'); cy3data = magetfield(maStruct,'F532 Median');
Создайте график зависимости интенсивности от отношения рассеяния данных cy3 и cy5. Нормализуйте данные и добавьте заголовок и метки:
mairplot(cy5data, cy3data, 'Normalize', true, ... 'Title','Normalized R vs G IR plot', ... 'Labels', maStruct.Names)
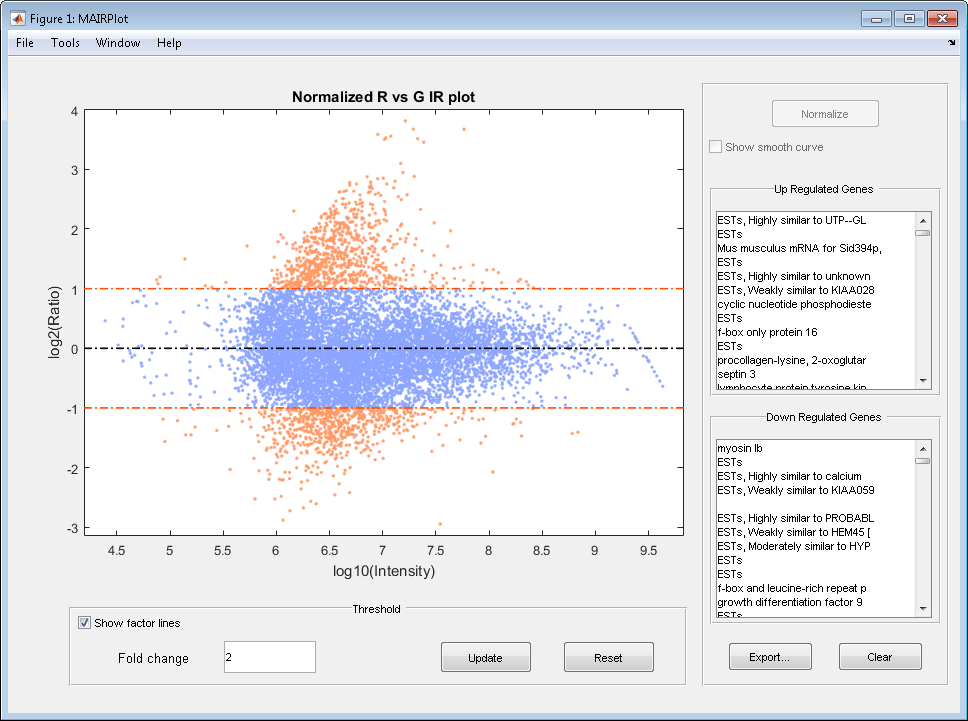
Возвращает значения интенсивности и отношения без отображения графика.
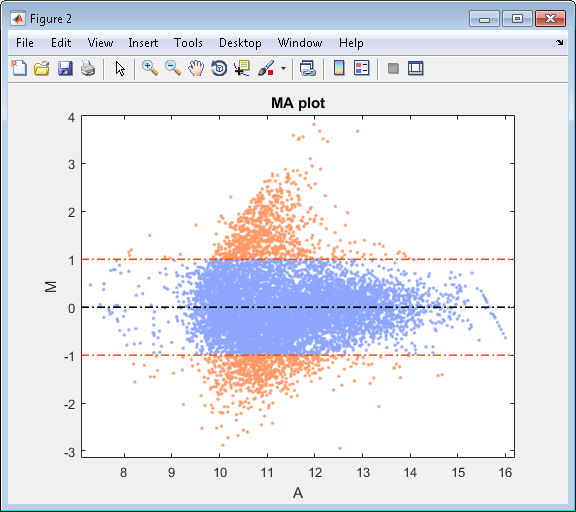
[intensities, ratios] = mairplot(cy5data, cy3data, 'Showplot', false);Создайте нормированный график MA данных cy3 и cy5 без компонентов пользовательского интерфейса.
mairplot(cy5data, cy3data, 'Normalize', true, ... 'Type','MA','PlotOnly',true)
[1] Квакенбуш, J. (2002). Нормализация и преобразование данных микрочипов. Nature Genetics Suppl. 32, 496-501.
[2] Dudoit, S., Yang, Y.H., Callow, M.J., and Speed, T.P. (2002). Статистические методы идентификации дифференциально экспрессируемых генов в экспериментах с реплицированными микрочипами кДНК. Statistica Sinica 12, 111-139.
maboxplot | magetfield | maimage | mainvarsetnorm | maloglog | malowess | manorm | mattest | mavolcanoplot