Повторная выборка сигнала с пиками при сохранении пиков
[X, Intensities] = msppresample(Peaklist, N)
msppresample(Peaklist, N, ...'Range', RangeValue, ...)
msppresample(Peaklist, N, ...'FWHH', FWHHValue, ...)
msppresample(Peaklist, N, ...'ShowPlot', ShowPlotValue, ...)
Peaklist | Одно из следующих действий:
Совет Вы можете использовать |
N | Целое число, указывающее количество равноотстоящих точек (значения единиц разделения) в повторно дискретизированном сигнале. |
RangeValue | Вектор 1 на 2, задающий минимальное и максимальное значения единиц разделения для выходной матрицы Intensities. RangeValue должно быть в пределах [min(, где inputSU - конкатенированные значения единиц разделения на входе Peaklist. По умолчанию - полный диапазон [min(. |
FWHHValue | Значение, указывающее полную ширину на половине высоты (FWHH) в единицах разделения. FWHH используется для преобразования каждого пика в кривую гауссовой формы. По умолчанию: median(diff(, где inputSU - конкатенированные значения единиц разделения на входе Peaklist. Значение по умолчанию - грубое приближение разрешения, наблюдаемого во входных данных, Peaklist.Совет Для обеспечения сохранения разрешающей способности пиков установите |
ShowPlotValue | Управляет отображением графика исходного и повторно дискретизированного сигнала. Варианты: true, false, или I, целое число, указывающее индекс сигнала в Intensities. Если задано значение true, первый сигнал в Intensities выводится на печать. Значение по умолчанию:
|
X | Вектор равноотстоящих, единиц разделения для набора сигналов с пиками. Число элементов в векторе равно Nили количество строк в матрице Intensities. |
Intensities | Матрица восстановленных значений интенсивности для набора пиков, которые имеют одинаковый диапазон единиц разделения. Каждая строка соответствует значению единицы разделения, и каждый столбец соответствует либо набору сигналов с пиками, либо времени удержания. Количество строк равно Nили количество элементов в векторе X. |
Совет
Используйте следующие синтаксисы с данными любого метода разделения, который производит данные сигнала, такие как спектроскопия, ЯМР, электрофорез, хроматография или масс-спектрометрия.
[ передискретизирует X, Intensities] = msppresample(Peaklist, N)Peaklistсписок пиков путем преобразования центроидальных пиков в полунепрерывный необработанный сигнал, который сохраняет пиковую информацию. Повторно дискретизированный сигнал имеет N равноудаленные точки. Продукция X является вектором N элементы, определяющие равноотстоящие, общие значения единиц разделения для набора сигналов с пиками. Продукция Intensities - матрица восстановленных значений интенсивности для набора пиков, которые имеют одинаковый диапазон единиц разделения. Каждая строка соответствует значению единицы разделения, и каждый столбец соответствует либо набору сигналов с пиками, либо времени удержания. Количество строк равно N.
msppresample использует гауссово ядро для восстановления сигнала. Интенсивность при любом заданном значении единицы разделения берется из максимальной интенсивности любых составляющих (перекрывающих) пиков.
Совет
msppresample полезен для подготовки набора сигналов для функций визуализации, таких как msheatmap и функции предварительной обработки, такие как msbackadj и msnorm.
msppresample( требования Peaklist, N, ... 'PropertyName', PropertyValue, ...)msppresample с необязательными свойствами, использующими пары имя/значение свойства. Можно указать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и не учитывать регистр. Эти пары имя/значение свойства следующие:
msppresample( задает диапазон единиц разделения для выходной матрицы Peaklist, N, ...'Range', RangeValue, ...)Intensities используя минимальное и максимальное значения разделения, указанные в векторе 1 на 2 RangeValue. RangeValue должно быть в пределах [min(, где inputSU) max(inputSU)]inputSU - конкатенированные значения единиц разделения на входе Peaklist. По умолчанию - полный диапазон [min(inputSU) max(inputSU)]
msppresample( устанавливает полную ширину на половине высоты (FWHH) в единицах разделения. FWHH используется для преобразования каждого пика в кривую гауссовой формы. По умолчанию: Peaklist, N, ...'FWHH', FWHHValue, ...)median(diff(, где inputSU))/2inputSU - конкатенированные значения единиц разделения на входе Peaklist. Значение по умолчанию - грубое приближение разрешения, наблюдаемого во входных данных, Peaklist.
Совет
Для обеспечения сохранения разрешающей способности пиков установите FWHHValue до половины расстояния между двумя интересующими пиками, ближайшими друг к другу.
msppresample( управляет отображением графика исходного и повторно дискретизированного сигнала. Варианты: Peaklist, N, ...'ShowPlot', ShowPlotValue, ...)true, false, или I, целое число, указывающее индекс сигнала в Intensities. Если задано значение true, первый сигнал в Intensities выводится на печать. Значение по умолчанию:
false - Когда указаны возвращаемые значения.
true - Когда возвращаемые значения не указаны.
Загрузите MAT-файл, включенный в программное обеспечение Bioinformatics Toolbox™, который содержит переменные данных жидкостной хроматографии/масс-спектрометрии (LC/MS). Он включает в себя peaksмассив ячеек списков пиков, где каждый элемент является матрицей из двух столбцов значений m/z и значений интенсивности ионов, и каждый элемент соответствует спектру или времени удержания.
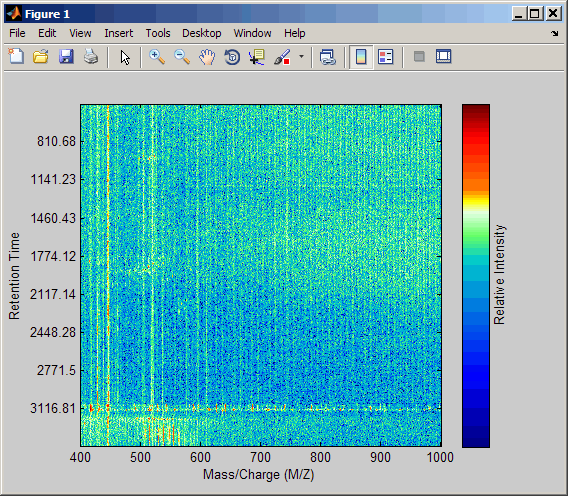
load lcmsdataВыполните повторную выборку данных, указав значения 5000 м/з в сигнале повторной выборки. Затем создайте тепловую карту данных LC/MS.
[MZ,Y] = msppresample(ms_peaks,5000); msheatmap(MZ,ret_time,log(Y))
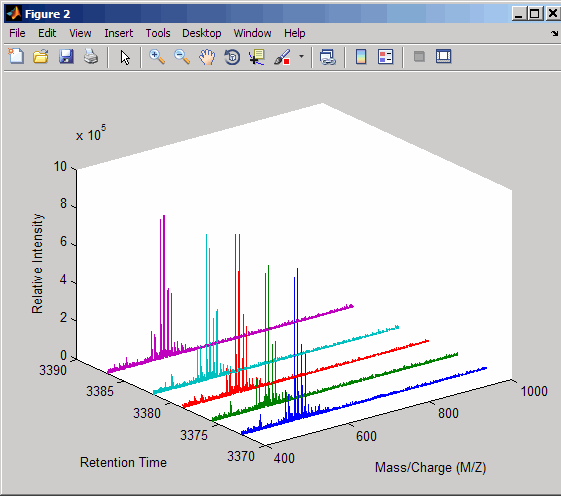
Постройте график восстановленных спектров профиля между двумя временами удержания.
figure t1 = 3370; t2 = 3390; h = find(ret_time>t1 & ret_time<t2); [MZ,Y] = msppresample(ms_peaks(h),10000); plot3(repmat(MZ,1,numel(h)),repmat(ret_time(h)',10000,1),Y) xlabel('Mass/Charge (M/Z)') ylabel('Retention Time') zlabel('Relative Intensity')
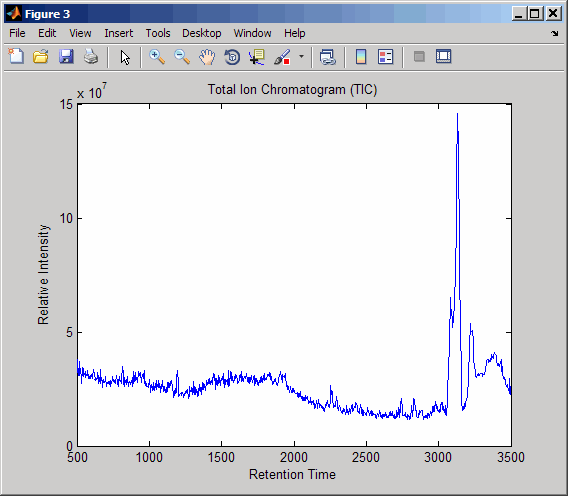
Выполните повторную выборку данных для построения общей ионной хроматограммы (TIC).
figure [MZ,Y] = msppresample(ms_peaks,5000); plot(ret_time,sum(Y)) title('Total Ion Chromatogram (TIC)') xlabel('Retention Time') ylabel('Relative Intensity')
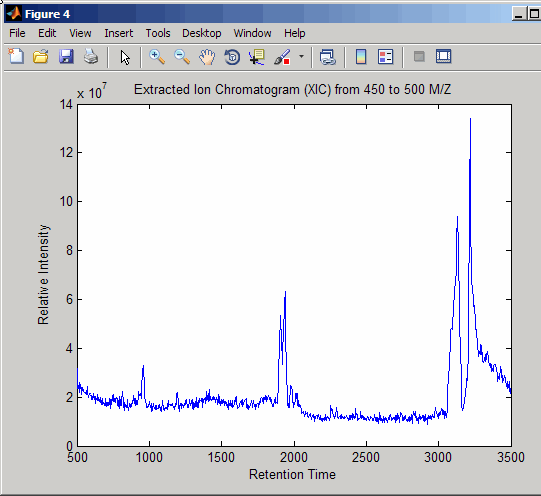
Выполните повторную выборку данных для построения графика извлеченной ионной хроматограммы (XIC) в диапазоне от 450 до 500 м/z.
figure [MZ,Y] = msppresample(ms_peaks,5000,'Range',[450 500]); plot(ret_time,sum(Y)) title('Extracted Ion Chromatogram (XIC) from 450 to 500 M/Z') xlabel('Retention Time') ylabel('Relative Intensity')
msalign | msbackadj | msdotplot | msheatmap | mslowess | msnorm | mspalign | mspeaks | msppresample | msresample | mssgolay | msviewer