График плотности нуклеотидов по последовательности
ntdensity(SeqNT)
Density = ntdensity(SeqNT)
... = ntdensity(..., 'Window', WindowValue, ...)
[Density, HighCG] = ntdensity(..., 'CGThreshold', CGThresholdValue, ...)
SeqNT | Одно из следующих:
Примечание Хотя вы можете представить последовательность с нуклеотидами, отличными от |
WindowValue | Значение, указывающее длину окна для расчета плотности. По умолчанию: |
CGThresholdValue | Управляет возвратом индексов для регионов, где |
ntdensity( строит график плотности нуклеотидов SeqNT)A, C, G, и T в последовательности SeqNT.
Density = ntdensity(SeqNT)A, C, G, и T.
... = ntdensity( требования SeqNT, ...'PropertyName', PropertyValue, ...)ntdensity с необязательными свойствами, использующими пары имя/значение свойства. Можно указать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и не учитывать регистр. Эти пары имя/значение свойства следующие:
... = ntdensity(..., 'Window', использует окно длины WindowValue, ...)WindowValue для расчета плотности. Дефолт WindowValue является length(.SeqNT)/20
[ возвращает индексы для регионов, где Density, HighCG] = ntdensity(..., 'CGThreshold', CGThresholdValue, ...)CG содержание SeqNT больше, чем CGThresholdValue. Дефолт CGThresholdValue является 5.
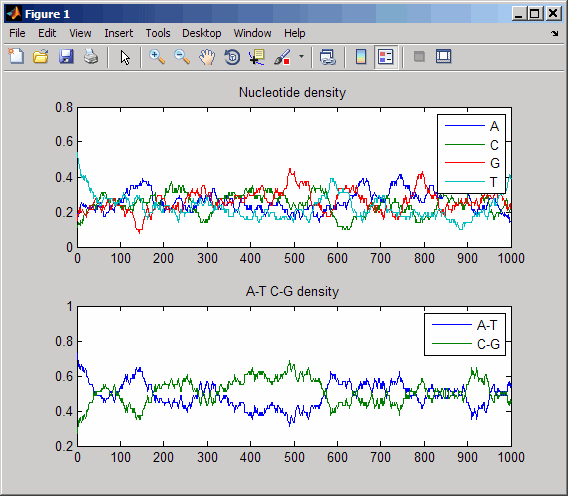
Создайте вектор случайных символов для представления нуклеотидной последовательности.
s = randseq(1000, 'alphabet', 'dna');
Постройте график плотности нуклеотидов вдоль последовательности.
ntdensity(s)
basecount | codoncount | cpgisland | dimercount | filter