Найти острова CpG в последовательности ДНК
cpgStruct = cpgisland(SeqDNA)
cpgStruct = cpgisland(SeqDNA, ...'Window', WindowValue, ...)
cpgStruct = cpgisland(SeqDNA, ...'MinIsland', MinIslandValue, ...)
cpgStruct = cpgisland(SeqDNA, ...'GCmin', GCminValue, ...)
cpgStruct = cpgisland(SeqDNA, ...'CpGoe', CpGoeValue, ...)
cpgStruct = cpgisland(SeqDNA, ...'Plot', PlotValue, ...)
SeqDNA | Одно из следующих:
Допустимые символы:
|
WindowValue | Целое число, указывающее размер окна для вычисления содержимого GC и отношения CpGobserved/CpGexpected. По умолчанию: 100 базы. Меньший размер окна увеличивает шум на графике. |
MinIslandValue | Целое число, указывающее минимальное количество последовательных помеченных баз для отчета как CpG-остров. По умолчанию: 200 базы. |
GCminValue | Значение, определяющее минимальный процент GC в окне, необходимый для пометки основания. Варианты - это значение между 0 и 1. По умолчанию: 0.5. |
CpGoeValue | Значение, указывающее минимальное отношение CpGobserved/CpGexpected в каждом окне, необходимое для пометки базы. Варианты - это значение между CPGobs/CpGexp = (NumCpGs*Length)/(NumGs*NumCs) |
PlotValue | Управляет выводом на печать содержимого GC, содержимого CpGoe, CpG-островов, размер которых превышает минимальный размер острова, и всех потенциальных CpG-островов в соответствии с указанными критериями. Варианты: true или false (по умолчанию). |
cpgStruct | Структура MATLAB, содержащая начальное и конечное основания островов CpG, превышает минимальный размер острова. |
cpgStruct = cpgisland(SeqDNA)SeqDNA, ДНК нуклеотидная последовательность для CpG островов с содержанием GC больше, чем 50% и отношение CpGobserved/CpGexpected больше, чем 60%. Он помечает базы, удовлетворяющие этому критерию, в пределах движущегося окна 100 ДНК-основания, а затем возвращает результаты в cpgStruct, структура MATLAB, содержащая начальное и конечное основания островов CpG, превышающие минимальный размер острова 200 базы.
cpgStruct = cpgisland(SeqDNA, ...'PropertyName', PropertyValue, ...)cpgisland с необязательными свойствами, использующими пары имя/значение свойства. Можно указать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и не учитывать регистр. Эти пары имя/значение свойства следующие:
задает размер окна для вычисления содержимого GC и отношения CpGobserved/CpGexpected. По умолчанию: cpgStruct = cpgisland(SeqDNA, ...'Window', WindowValue, ...)100 базы. Меньший размер окна увеличивает шум на графике.
cpgStruct = cpgisland(SeqDNA, ...'MinIsland', MinIslandValue, ...)200 базы.
cpgStruct = cpgisland(SeqDNA, ...'GCmin', GCminValue, ...)0 и 1. По умолчанию: 0.5.
cpgStruct = cpgisland(SeqDNA, ...'CpGoe', CpGoeValue, ...)0 и 1. По умолчанию: 0.6. Это соотношение определяется как:
CPGobs/CpGexp = (NumCpGs*Length)/(NumGs*NumCs)
cpgStruct = cpgisland(SeqDNA, ...'Plot', PlotValue, ...)true или false (по умолчанию).
Импортируйте нуклеотидную последовательность из базы данных GenBank ®. Например, извлекают последовательность из хромосомы 12 Homo sapiens.
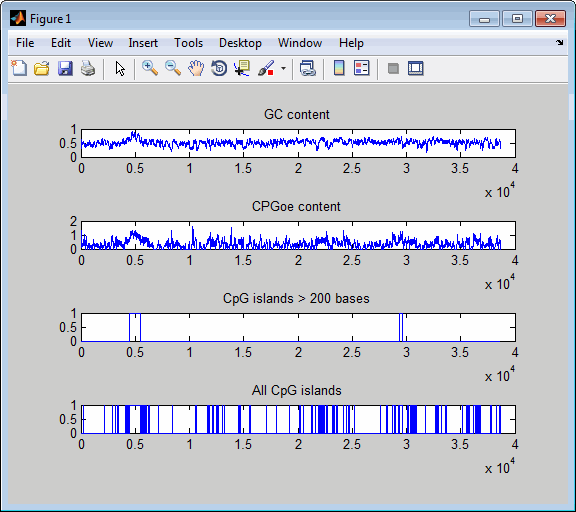
S = getgenbank('AC156455');Вычислите островки CpG в последовательности и постройте график результатов.
cpgisland(S.Sequence,'PLOT',true)
ans =
Starts: [4510 29359]
Stops: [5468 29604]
Перечислены острова CpG длиной более 200 оснований и отображается график.