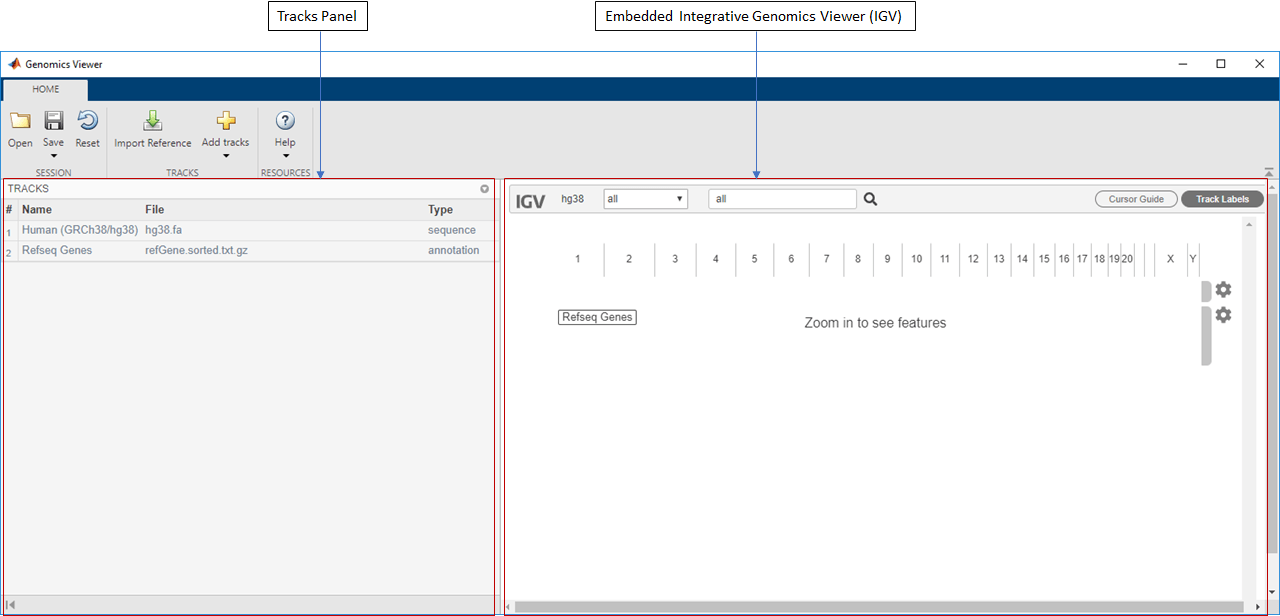
Приложение Genomics Viewer позволяет просматривать и исследовать интегрированные геномные данные с помощью встроенной версии [1][2] Integrative Genomics Viewer (IGV). Геномные данные включают выравнивания чтения NGS, варианты генома и сегментированные данные номера копии.
В первой части этого примера представлен краткий обзор приложения и поддерживаемых форматов файлов. Вторая часть примера исследует однонуклеотидную вариацию гена цитохрома р450 (CYP2C19).
В командной строке введите genomicsViewer. Либо щелкните значок приложения на вкладке Приложения. Приложению требуется подключение к Интернету.
По умолчанию приложение загружает Human (GRCh38/hg38) в качестве ссылочной последовательности и Refseq Genes в качестве файла аннотации. В приложении имеются две основные панели. Левая панель является панелью «Дорожки», а правая панель является встроенным веб-приложением ВНА. Панель «Дорожки» - это область, доступная только для чтения, в которой отображаются имена дорожек, имена исходных файлов и типы дорожек. Панель «Дорожки» обновляется соответствующим образом при настройке дорожек во встроенном приложении ВНА.
Кнопка Сброс восстанавливает приложение в представление по умолчанию с двумя треками (HG38 с Refseq Genes) и удаляет любые другие существующие треки. Перед сбросом можно сохранить текущее представление как сеанс (.json) и восстановите его позже.
Можно импортировать одну ссылочную последовательность. Ссылочная последовательность должна находиться в файле FASTA. Выберите Импортировать ссылку (Import Reference) на вкладке Главная страница (Home). Также можно импортировать соответствующий файл цитобенда, содержащий данные цитогенетического G-banding. Можно добавить локальные файлы или указать внешние URL-адреса. URL-адрес должен начинаться с https или gs. Другие протоколы передачи файлов, например ftp, не поддерживаются.
Можно импортировать несколько наборов данных последовательности считывания данных трассы. Данные выравнивания должны быть файлом BAM или CRAM. Не обязательно иметь соответствующий индексный файл (.BAI или .CRAI) в том же месте, что и файл BAM или CRAM. Однако отсутствие индексного файла замедлит работу приложения.
Файлы трассы для чтения можно добавлять с помощью опций «Добавить дорожки из файла» и «Добавить дорожки из URL-адреса» с помощью кнопки «Добавить дорожки». Если указан URL-адрес, он должен начинаться с https или gs. Другие протоколы передачи файлов, например ftp, не поддерживаются.
Можно импортировать несколько наборов аннотаций элементов из нескольких файлов, содержащих данные для одной последовательности привязок. Поддерживаются следующие файлы аннотаций: .BED, .GFF, .GFF3, и .GTF.
Можно также импортировать структурные варианты (.VCF) и визуализировать генетические изменения, такие как вставки и удаления.
Можно просмотреть сегментированные данные номера копии (.SEG) и количественные геномные данные (.WIG, .BIGWIG, и .BEDGRAPH), такие как пики ChIP и покрытие выравнивания.
Можно добавить файлы аннотаций и геномных данных с помощью опций Добавить дорожки из файла и Добавить дорожки из URL-адреса с помощью кнопки Добавить дорожки. Если указан URL-адрес, он должен начинаться с https или gs. Другие протоколы передачи файлов, например FTP, не поддерживаются.
Ген CYP2C19 входит в семейство генов цитохрома P450. Ферменты, полученные из генов цитохрома P450, участвуют в метаболизме различных молекул и химических веществ в клетках. Фермент CYP2C19 играет роль в метаболизме по меньшей мере 10 процентов обычно назначаемых лекарств [3]. Полиморфизмы в семействе цитохрома р450 могут вызывать неблагоприятные реакции лекарств у людей. Одним из примеров однонуклеотидной вариации является rs4986893 в положении chr10: 94 780 653, гдеG заменяется на A. Этот аллельный вариант также известен как CYP2C19 * 3. Следующие шаги показывают, как визуализировать такое изменение в приложении, используя данные как с низким покрытием, так и с высоким покрытием.
Для целей этого примера начните с файла сеанса, который имеет некоторые предварительно загруженные дорожки. Чтобы загрузить файл, нажмите кнопку «Открыть». Перейти к matlabroot\examples\bioinfo\, где matlabroot - папка, в которой установлен MATLAB ®. Выбратьrs4986893.json.
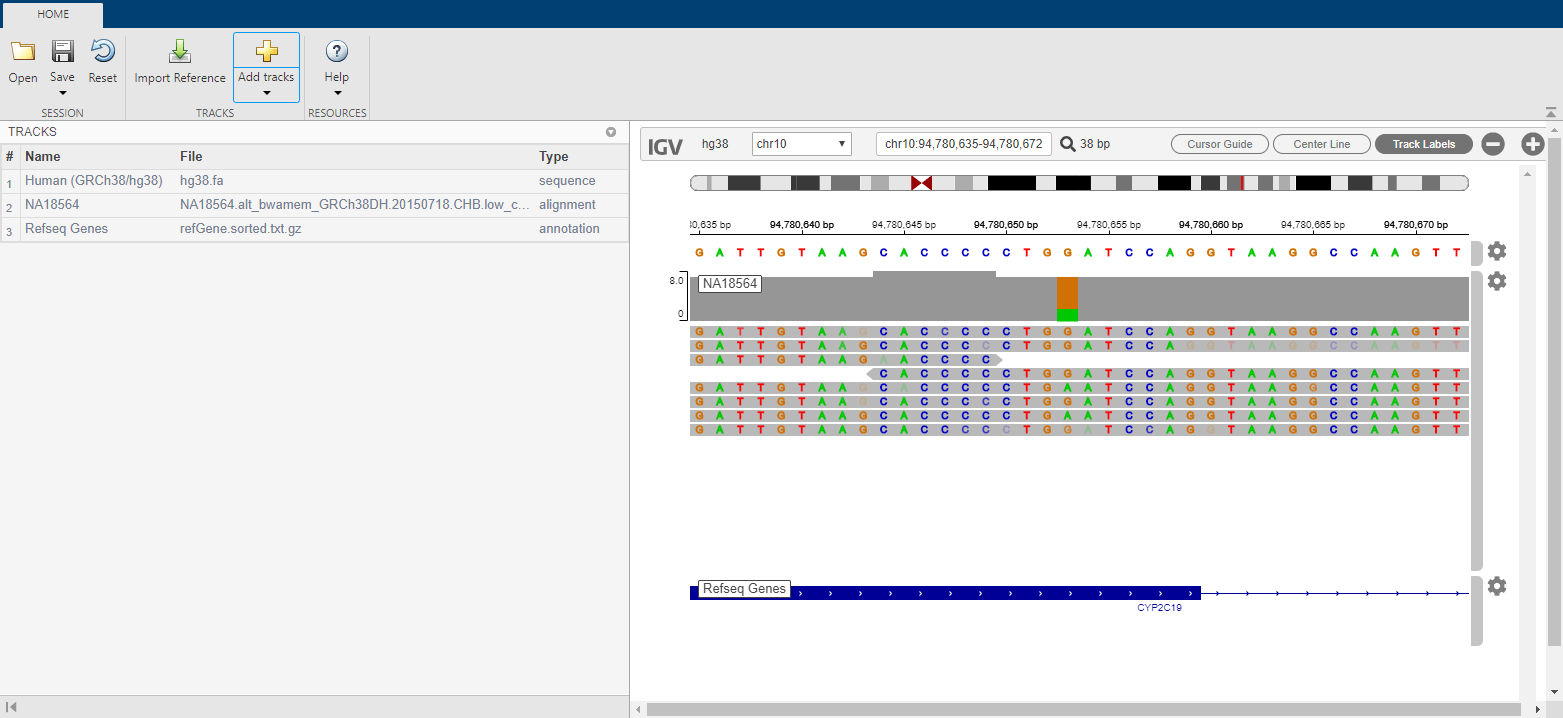
Сессия содержит три направления:
Человек (GRCh38/hg38) в качестве эталона
NA18564 как данные выравнивания с низким уровнем покрытия
Гены Refseq
Данные о выравнивании с низким охватом получены от китаянки Хань из Пекина, Китай. Идентификатор образца является NA18564, и образец был идентифицирован мутацией CYP2C19 * 3 [4].
В этом файле сеанса данные выравнивания были центрированы вокруг местоположения мутации на гене CYP2C19.
Щелкните оранжевую строку в зоне покрытия для просмотра информации о положении и распределении аллелей.
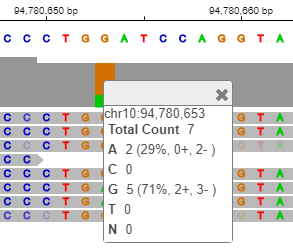
Это показывает, что 71% чтения имеют G, в то время как 29% имеют A в месте chr10: 94 780 653. Эти данные являются данными с низким охватом и могут не показывать все проявления этой мутации. Данные с высоким охватом будут изучены позже в примере.
Закройте окно подсказки по данным.
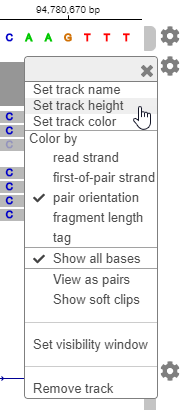
Можно настроить различные аспекты отображения данных в приложении. Например, можно изменить высоту дорожки, чтобы освободить место для последующих дорожек. Щелкните значок второго зубчатого колеса. Выбрать Set track height. Введите 200.
Подробную информацию о встроенном приложении для ВНА и его доступных вариантах можно найти здесь.
Вы можете посмотреть на данные о высоком покрытии из того же образца, чтобы увидеть проявления этой мутации.
Перейдите на веб-сайт Международного ресурса образцов генома.
Выполните поиск образца NA18564.
Загрузите файл выравнивания Exome, который находится в .CRAM формат.
Также загрузите соответствующий индексный файл, который находится в .CRAI формат. Сохранить файл в том же расположении, что и источник .CRAM файл.
Щелкните значок (+) на вкладке Главная страница. Выберите загруженное .CRAM и нажмите кнопку «Открыть».
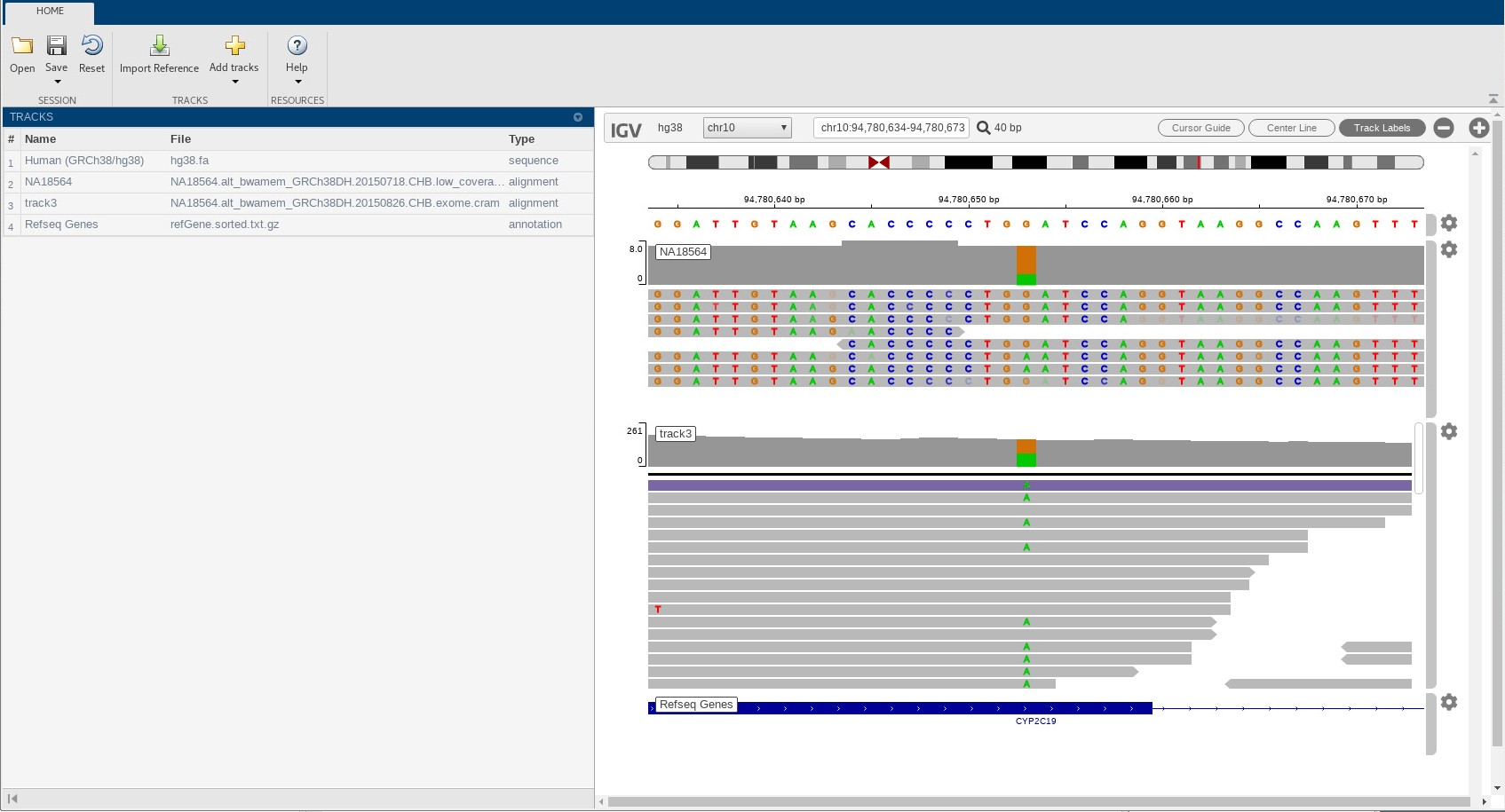
Данные с высоким уровнем покрытия отображаются как track3. Теперь вы можете увидеть много случаев мутации в нескольких чтениях.
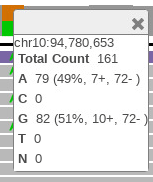
Щелкните оранжевую полосу в зоне покрытия, чтобы увидеть распределение аллелей. Это показывает, что G заменяется на A почти за 50% времени.
[1] Робинсон, Дж., Х. Торвальдсдоттир, У. Винклер, М. Гуттман, Э. Ландер, Г. Гетц, Ж. Месиров. 2011. Средство просмотра интегративной геномики. Биотехнология природы. 29:24–26.
[2] Торвальдсдоттир, Х., Дж. Робинсон, Дж. Месиров. 2013. Integrative Genomics Viewer (IGV): высокопроизводительная визуализация и исследование данных геномики. Брифинги по биоинформатике. 14:178–192.
Средство просмотра геномики | Выравнивание последовательности | Просмотр последовательности