Выполните 2D демонстрационный t-тест, чтобы оценить дифференциальную экспрессию генов от двух экспериментальных условий или фенотипов
PValues = mattest(DataX,
DataY)
[PValues, TScores] = mattest(DataX, DataY)
[PValues, TScores, DFs]
= mattest(DataX, DataY)
... = mattest(..., 'VarType', VarTypeValue, ...)
... = mattest(..., 'Permute', PermuteValue, ...)
... = mattest(..., 'Bootstrap', BootstrapValue, ...)
... = mattest(..., 'Showhist', ShowhistValue, ...)
... = mattest(..., 'Showplot', ShowplotValue, ...)
... = mattest(..., 'Labels', LabelsValue, ...)
DataX, DataY | Объект DataMatrix или матрица значений экспрессии гена, где каждая строка соответствует гену и каждому столбцу, соответствуют реплицированию.
|
VarTypeValue | Вектор символов, который задает тип отклонения теста. VarTypeValue может быть 'equal' или 'unequal' (значение по умолчанию). Если установлено в 'equal', mattest выполняет тест, принимающий, что эти две выборки имеют равные отклонения. Если установлено в 'unequal', mattest выполняет тест, принимающий, что эти две выборки имеют неизвестные и неравные отклонения. |
PermuteValue | Средства управления, запущены ли тесты перестановки, и если так, сколько. Выбором является true, false (значение по умолчанию) или любое целое число, больше, чем 2. Если установлено в true, количеством перестановок является 1000. |
BootstrapValue | Средства управления, запущены ли тесты начальной загрузки, и если так, сколько. Выбором является true, false (значение по умолчанию) или любое целое число, больше, чем 2. Если установлено в true, количеством тестов начальной загрузки является 1000. |
ShowhistValue | Управляет отображением гистограмм дистрибутивов t-счета и p-распределений-значений. Выбором является |
ShowplotValue | Управляет отображением нормального графика квантиля t-счета. Выбором является |
LabelsValue | Массив ячеек из символьных векторов или вектор строки, содержащий метки (обычно названия генов или тестовые идентификаторы набора) для каждой строки в DataX и DataY. Метки отображаются, если вы кликаете по точке данных в графике квантиля t-счета. |
PValues | Одно из следующего:
|
TScores | Вектор-столбец t-музыки к каждому гену в DataX и DataY. |
DFs | Вектор-столбец, содержащий степень свободы для каждого гена в DataX и DataY. |
PValues = mattest(DataX,
DataY)DataX и DataY и возвращает p-значение для каждого гена. DataX и DataY являются или объектом DataMatrix или матрицей значений экспрессии гена, в которых каждая строка соответствует гену, и каждый столбец соответствует реплицированию. DataX содержит данные из одного экспериментального условия, и DataY содержит данные из другого экспериментального условия. DataX и DataY должны иметь одинаковое число строк и приняты, чтобы быть нормально распределенными в каждом классе. PValues является вектор-столбцом p-значений для каждого гена, или, если по крайней мере одни из входных параметров являются объектом DataMatrix, объект DataMatrix со строкой называет то же самое как первый объект входа DataMatrix и имя столбца p-values.
[ также возвращает t-счет к каждому гену в PValues, TScores] = mattest(DataX, DataY)DataX и DataY. TScores является вектор-столбцом t-музыки к каждому гену.
[ также возвращает PValues, TScores, DFs]
= mattest(DataX, DataY)DFs, вектор-столбец, содержащий степень свободы для каждого гена через оба набора данных, DataX и DataY.
... = mattest(..., 'PropertyName', PropertyValue, ...) mattest с дополнительными свойствами, которые используют имя свойства / пары значения свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и нечувствительный к регистру. Это имя свойства / пары значения свойства следующие:
... = mattest(..., 'VarType', задает тип отклонения теста. VarTypeValue, ...)VarTypeValue может быть 'equal' или 'unequal' (значение по умолчанию). Если установлено в 'equal', mattest выполняет тест, принимающий, что эти две выборки имеют равные отклонения. Если установлено в 'unequal', mattest выполняет тест, принимающий, что эти две выборки имеют неизвестные и неравные отклонения.
... = mattest(..., 'Permute', средства управления, запущены ли тесты перестановки, и если так, сколько. PermuteValue, ...)PermuteValue может быть true, false (значение по умолчанию) или любое целое число, больше, чем 2. Если установлено в true, количеством перестановок является 1000.
... = mattest(..., 'Bootstrap', средства управления, запущены ли тесты начальной загрузки, и если так, сколько. BootstrapValue, ...)BootstrapValue может быть true, false (значение по умолчанию) или любое целое число, больше, чем 2. Если установлено в true, количеством тестов начальной загрузки является 1000.
... = mattest(..., 'Showhist', управляет отображением гистограмм дистрибутивов t-счета и p-распределений-значений. Когда ShowhistValue, ...)ShowhistValue является true, гистограммы отображений mattest. Значением по умолчанию является false.
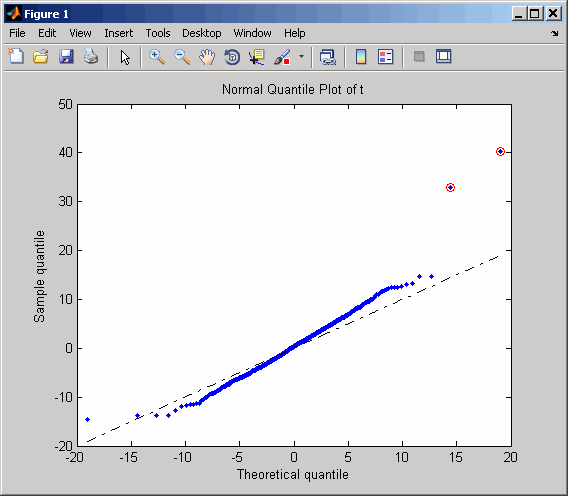
... = mattest(..., 'Showplot', управляет отображением нормального графика квантиля t-счета. Когда ShowplotValue, ...)ShowplotValue является true, mattest отображает график квантиля квантиля. Значением по умолчанию является false. В графике квантиля t-счета строка черной диагонали представляет демонстрационный квантиль, являющийся равным теоретическому квантилю. Точки данных генов, считавшихся дифференцированно выраженными, лежат дальше от этой строки. А именно, точки данных с t-очками> (1 - 1/(2N)) или <отображение 1/(2N) с красными кругами. N является общим количеством генов.
... = mattest(..., 'Labels', управляет отображением меток, когда вы кликаете по точке данных в графике квантиля t-счета. LabelsValue, ...)LabelsValue является массивом ячеек из символьных векторов или вектором строки, содержащим метки (обычно названия генов или тестовые идентификаторы набора) для каждой строки в DataX и DataY.
Загрузите MAT-файл, включенный с программным обеспечением Bioinformatics Toolbox™, которое содержит данные Affymetrix® из исследования рака простаты, в частности тестовые данные об интенсивности из массивов Affymetrix HG-U133A GeneChip®. Эти две переменные в MAT-файле, dependentData и independentData, являются двумя матрицами значений экспрессии гена от двух экспериментальных условий.
load prostatecancerexpdataВычислите p-значения и t-музыку к значениям экспрессии гена в этих двух матрицах и отобразите нормальный график квантиля t-счета.
[pvalues,tscores] = mattest(dependentData, independentData,... 'showplot',true);
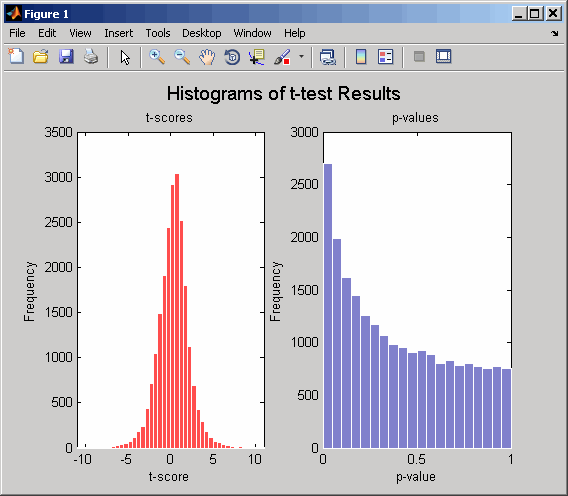
Вычислите p-значения и t-очки снова с помощью тестов перестановки (1 000 перестановок) и отобразив гистограммы дистрибутивов t-счета и p-распределений-значений.
[pvalues,tscores] = mattest(dependentData,independentData,... 'permute',true,'showhist',true,... 'showplot',true);
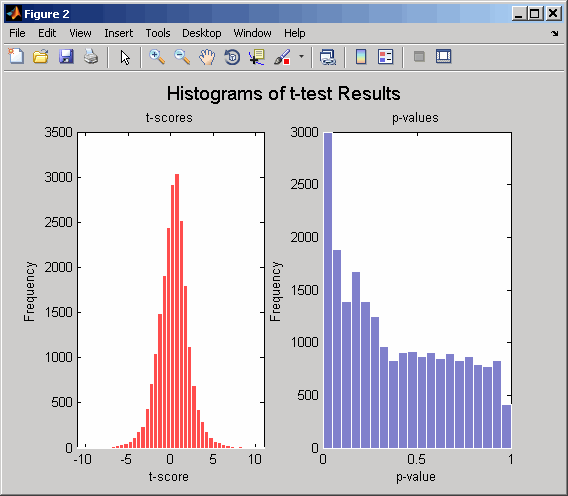
Вычислите p-значения и t-очки снова с помощью тестов начальной загрузки (2 000 тестов) и отобразив гистограммы дистрибутивов t-счета и p-распределений-значений.
[pvalues,tscores] = mattest(dependentData,independentData,... 'bootstrap',2000,'showhist',true,... 'showplot',true);
Файл prostatecancerexpdata.mat, используемый в этом примере, содержит данные из Лучшего и др., 2005.
[1] Литература анализа: Хубер, W., фон Хайдебрек, A., Зюлтманн, H., Poustka, A. и Vingron, M. (2002). Стабилизация отклонения применилась к калибровке микроданных массива и квантификации дифференциального выражения. Биоинформатика 18 (Suppl. 1), S96–S104.
[2] Лучше всего, C.J.M., Гиллеспи, J.W., И, Y., Chandramouli, G.V.R., Perlmutter, M.A., Gathright, Y., Эриксон, H.S., Георгевич, L., Tangrea, M.A., Duray, P.H., Гонсалес, S., Веласко, A., Linehan, W.M., Matusik, R.J., Цена, D.K., Figg, W.D., Emmert-маркер, M.R., и Chuaqui, R.F. (2005). Молекулярные изменения при первичном раке простаты после терапии абляции андрогена. Клинические Исследования рака 11, 6823–6834.
affygcrma | affyrma | maboxplot | mafdr | mainvarsetnorm | mairplot | maloglog | malowess | manorm | mavolcanoplot | rmasummary