Постройте плотность нуклеотидов вдоль последовательности
ntdensity(SeqNT)
Density = ntdensity(SeqNT)
... = ntdensity(..., 'Window', WindowValue,
...)
[Density, HighCG]
= ntdensity(..., 'CGThreshold', CGThresholdValue,
...)
SeqNT | Одно из следующего:
ПримечаниеНесмотря на то, что можно представить последовательность с нуклеотидами кроме |
WindowValue | Значение, которое задает длину окна для вычисления плотности. Значением по умолчанию является |
CGThresholdValue | Управляет возвратом индексов для областей, где содержимое |
ntdensity( строит плотность нуклеотидов SeqNT)A, C, G и T в последовательности SeqNT.
Density = ntdensity(SeqNT)A, C, G и T.
... = ntdensity(SeqNT, ...'PropertyName', PropertyValue, ...) ntdensity с дополнительными свойствами, которые используют имя свойства / пары значения свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и нечувствительный к регистру. Это имя свойства / пары значения свойства следующие:
... = ntdensity(..., 'Window', использует окно длины WindowValue,
...)WindowValue для вычисления плотности. WindowValue по умолчанию является length(SeqNT)/20
[ возвращает индексы для областей, где содержимое Density, HighCG]
= ntdensity(..., 'CGThreshold', CGThresholdValue,
...)CG SeqNT больше, чем CGThresholdValue. CGThresholdValue по умолчанию является 5.
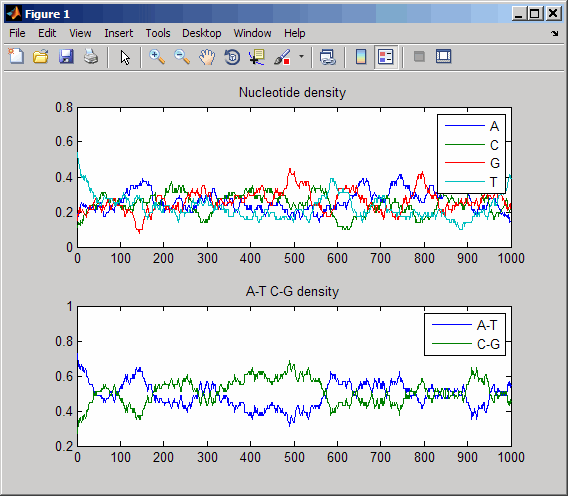
Создайте случайный вектор символов, чтобы представлять последовательность нуклеотида.
s = randseq(1000, 'alphabet', 'dna');
Постройте плотность нуклеотидов вдоль последовательности.
ntdensity(s)
basecount | codoncount | cpgisland | dimercount | filter