Свойства графика аминокислотной последовательности
proteinpropplot (SeqAA)
proteinpropplot(SeqAA, ...'PropertyTitle', PropertyTitleValue, ...)
proteinpropplot(SeqAA, ...'Startat', StartatValue, ...)
proteinpropplot(SeqAA, ...'Endat', EndatValue, ...)
proteinpropplot(SeqAA, ...'Smoothing', SmoothingValue, ...)
proteinpropplot(SeqAA, ...'EdgeWeight', EdgeWeightValue, ...)
proteinpropplot(SeqAA, ...'WindowLength', WindowLengthValue, ...)
SeqAA | Аминокислотная последовательность. Введите любое из следующих значений:
|
PropertyTitleValue | Символьный вектор или строка, задающая свойство для печати. По умолчанию: Hydrophobicity (Kyte & Doolittle). Чтобы отобразить список свойств для печати, введите пустой символьный вектор или пустую строку для PropertyTitleValue. Например, введите:proteinpropplot(sequence, 'propertytitle', '') Совет Чтобы получить доступ к ссылкам для свойств, просмотрите |
StartatValue | Целое число, указывающее начальную точку графика от N-концевого конца аминокислотной последовательности SeqAA. По умолчанию: 1. |
EndatValue | Целое число, указывающее конечную точку графика от N-концевого конца аминокислотной последовательности SeqAA. По умолчанию: length(. |
SmoothingValue | Символьный вектор или строка, определяющая метод сглаживания. Возможны следующие варианты:
|
EdgeWeightValue | Значение, указывающее вес кромки, используемый для методов линейного и экспоненциального сглаживания. Уменьшение этого значения подчеркивает пики на графике. Варианты - это любое значение ≥0 и ≤1. По умолчанию: 1. |
WindowLengthValue | Целое число, указывающее длину окна для метода сглаживания. Увеличение этого значения дает более плавный график, который показывает меньшую детализацию. По умолчанию: 11. |
proteinpropplot ( отображает график гидрофобности (Kyte and Doolittle, 1982) остатков в последовательности SeqAA)SeqAA.
proteinpropplot( требования SeqAA, ...'PropertyName', PropertyValue, ...)proteinpropplot с необязательными свойствами, использующими пары имя/значение свойства. Можно указать одно или несколько свойств в любом порядке. Каждый PropertyName должен быть заключен в одинарные кавычки и не учитывать регистр. Эти пары имя/значение свойства следующие:
proteinpropplot( задает свойство для построения графика аминокислотной последовательности SeqAA, ...'PropertyTitle', PropertyTitleValue, ...)SeqAA. По умолчанию: Hydrophobicity (Kyte & Doolittle). Чтобы отобразить список возможных свойств для печати, введите пустой символьный вектор или пустую строку для PropertyTitleValue. Например, введите:
proteinpropplot(sequence, 'propertytitle', '')
Совет
Чтобы получить доступ к ссылкам для свойств, просмотрите proteinpropplot файл.
proteinpropplot( задает начальную точку для графика от N-концевого конца аминокислотной последовательности SeqAA, ...'Startat', StartatValue, ...)SeqAA. По умолчанию: 1.
proteinpropplot( указывает конечную точку графика от N-концевого конца аминокислотной последовательности SeqAA, ...'Endat', EndatValue, ...)SeqAA. По умолчанию: length(.SeqAA)
proteinpropplot( задает метод сглаживания. Возможны следующие варианты: SeqAA, ...'Smoothing', SmoothingValue, ...)
linear (по умолчанию)
exponential
lowess
proteinpropplot( определяет вес кромки, используемый для методов линейного и экспоненциального сглаживания. Уменьшение этого значения подчеркивает пики на графике. Варианты - это любое значение SeqAA, ...'EdgeWeight', EdgeWeightValue, ...)≥0 и ≤1. По умолчанию: 1.
proteinpropplot( задает длину окна для метода сглаживания. Увеличение этого значения дает более плавный график, который показывает меньшую детализацию. По умолчанию: SeqAA, ...'WindowLength', WindowLengthValue, ...)11.
Используйте getpdb функция для извлечения последовательности белка.
prion = getpdb('1HJM', 'SEQUENCEONLY', true);Постройте график гидрофобности (Kyte and Doolittle, 1982) остатков в последовательности.
proteinpropplot(prion)
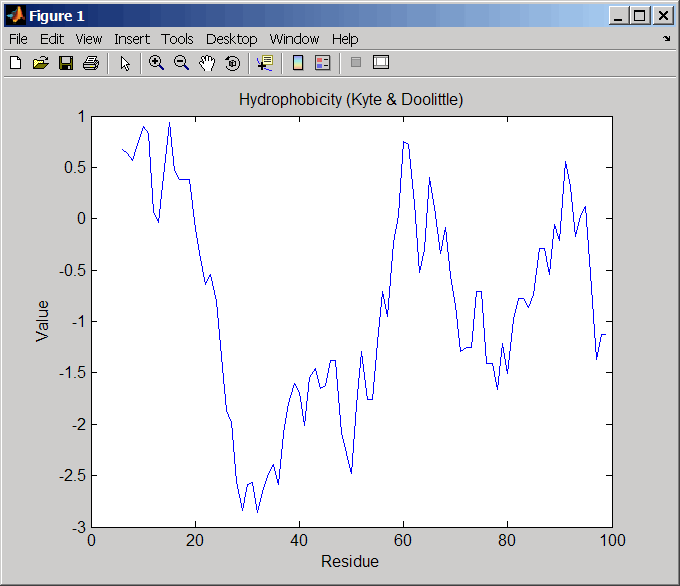
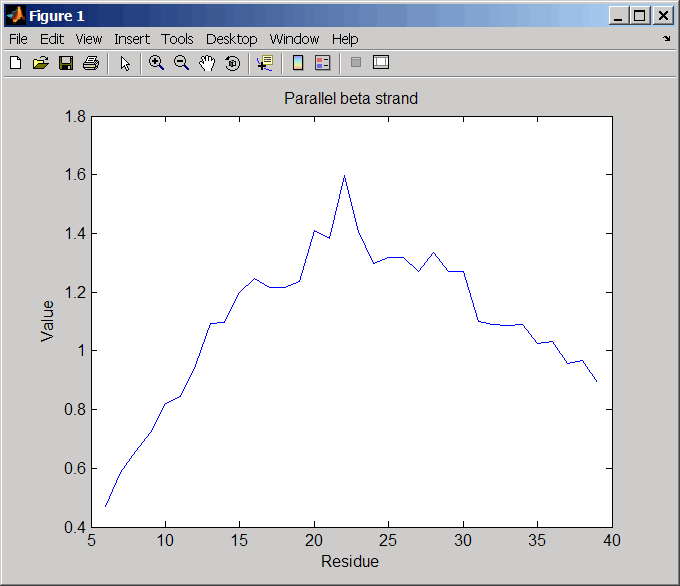
Используйте getgenpept функция для извлечения последовательности белка.
s = getgenpept('aad50640');Постройте график конформационного предпочтения параллельной бета-цепи для остатков в последовательности.
proteinpropplot(s,'propertytitle','Parallel beta strand')
[1] Кайт, Дж., и Дулиттл, Р. Ф. (1982). Простой способ проявления гидропатического характера белка. J Mol Biol 157 (1), 105-132.
aacount | atomiccomp | molviewer | molweight | pdbdistplot | plotyy | proteinplot | ramachandran | seqviewer