Создайте объект phytree
Tree = phytree(B)
Tree = phytree(B, D)
Tree = phytree(B, C)
Tree = phytree(BC)
Tree = phytree(..., N)
Tree = phytree
B | Числовой массив размера |
C | Вектор-столбец с расстояниями для каждого ответвления. |
D | Вектор-столбец с расстояниями от каждого узла до их родительского ответвления. |
BC | Объединенная матрица с указателями на ответвления или листы и расстояния ответвлений. |
| Массив ячеек с именами листов и ответвлений. |
Tree = phytree(B)
B является числовым массивом размера [NUMBRANCHES X 2], в котором каждая строка представляет ответвление дерева, и это содержит два указателя на ответвление или вершины, которые являются его дочерними элементами.
Вершины пронумерованы от 1 до NUMLEAVES, и узлы ответвления пронумерованы от NUMLEAVES + 1 до NUMLEAVES + NUMBRANCHES. Обратите внимание на то, что, потому что только двоичные деревья позволены, NUMLEAVES = NUMBRANCHES + 1.
Ответвления заданы в хронологическом порядке (например, B(i,:) > NUMLEAVES + i). Как следствие первая строка может только иметь указатели на листы, и последняя строка должна представлять корневое ответвление. Родительско-дочерние расстояния установлены в 1, если дочерний элемент не является листом и удовлетворить ультраметрическое условие дерева, его расстояние увеличено.
Учитывая дерево с тремя листами и двумя ответвлениями как пример.
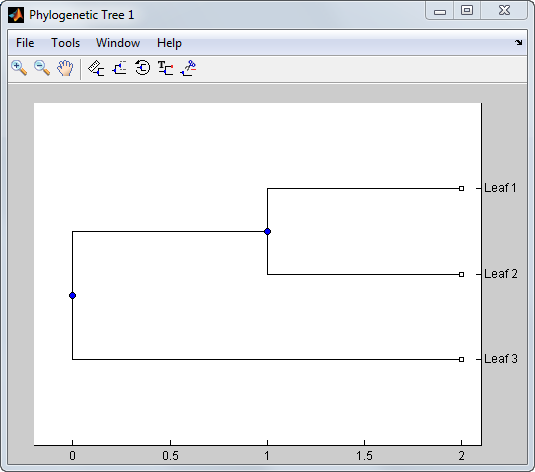
В Командном окне MATLAB® ввести
B = [1 2 ; 3 4]
B =
1 2
3 4
tree = phytree(B)
Phylogenetic tree object with 3 leaves (2 branches)
view(tree)
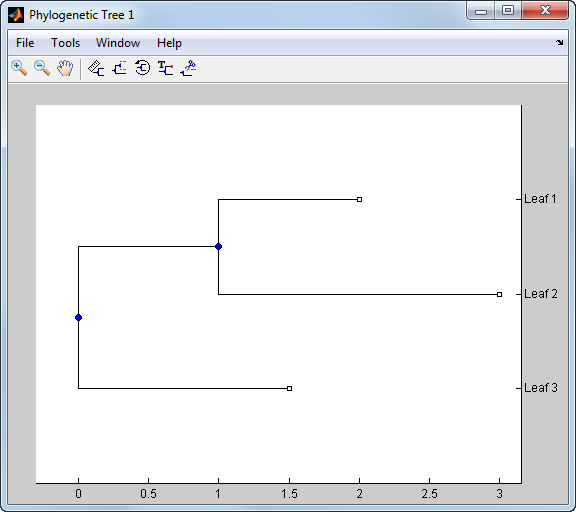
Tree = phytree(B, D)D. D является числовым массивом размера [NUMNODES X 1] с расстояниями каждого дочернего узла (лист или ответвление) к его родительскому ответвлению, равному NUMNODES = NUMLEAVES + NUMBRANCHES. Последнее расстояние в D является расстоянием корневого узла и бессмысленно.
b = [1 2 ; 3 4 ]
b =
1 2
3 4
d = [1; 2; 1.5; 1; 0]
d =
1.0000
2.0000
1.5000
1.0000
0
view(phytree(b,d))
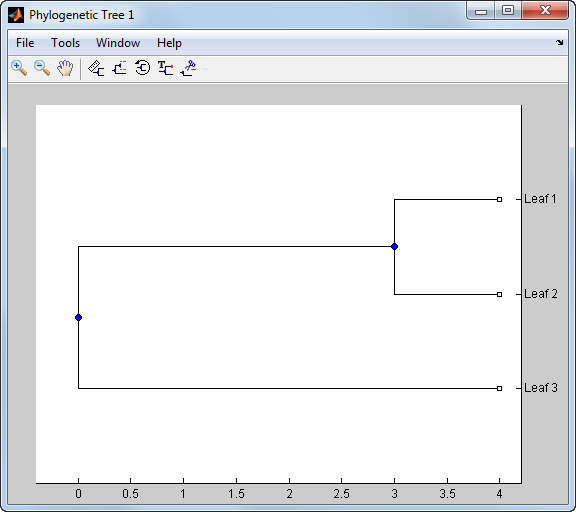
Tree = phytree(B, C)C. C является числовым массивом размера [NUMBRANCHES X 1], который содержит расстояние от каждого ответвления до листов. В ультраметрических деревьях все листы в том же местоположении (то же расстояние до корня).
b = [1 2 ; 3 4]
b =
1 2
3 4
c = [1 4]'
c =
1
4
view(phytree(b,c))
Tree = phytree(BC)BC(:,[1 2]) и координатами ответвления в BC(:,3). То же самое как phytree(B,C).
Tree = phytree(..., N)N является вектором строки или массивом ячеек из символьных векторов. Если NUMEL(N)==NUMLEAVES, то имена присвоены хронологически листам. Если NUMEL(N)==NUMBRANCHES, имена присвоены узлам ответвления. Если NUMEL(N)==NUMLEAVES + NUMBRANCHES, все узлы называют. Неприсвоенное значение по умолчанию имен к 'Leaf #' и/или 'Branch #' как требуется.
Tree = phytree
cluster | get | getbyname | getcanonical | getmatrix | getnewickstr | pdist | phytreeread | phytreeviewer | phytreewrite | plot | prune | reroot | select | seqlinkage | seqneighjoin | seqpdist | subtree | view | weights