Создайте график поля точек интенсивности от отношения микромассива данных
mairplot(DataX, DataY)
[Intensity, Ratio]
= mairplot(DataX, DataY)
[Intensity, Ratio, H]
= mairplot(DataX, DataY)
... = mairplot(...,
'Type', TypeValue, ...)
... = mairplot(...,
'LogTrans', LogTransValue, ...)
... = mairplot(...,
'FactorLines', FactorLinesValue, ...)
... = mairplot(...,
'Title', TitleValue, ...)
... = mairplot(...,
'Labels', LabelsValue, ...)
... = mairplot(...,
'Normalize', NormalizeValue, ...)
... = mairplot(...,
'LowessOptions', LowessOptionsValue,
...)
... = mairplot(...,
'Showplot', ShowplotValue, ...)
... = mairplot(...,
'PlotOnly', PlotOnlyValue, ...)
DataX, DataY | Объект DataMatrix или вектор значений экспрессии генов, где каждая строка соответствует гену. Для примера в двухцветном микромассиве эксперименте DataX могут быть значения интенсивности cy3 и DataY могут быть значения интенсивности cy5. |
TypeValue | Вектор символов или строка, которая задает тип графика. Варианты 'IR' (строит лог10 продукты DataX и DataY интенсивность от лога2 коэффициентов интенсивности) или 'MA' (графики (1/2) log2 продукта DataX и DataY интенсивность от лога2 коэффициентов интенсивности). По умолчанию это 'IR'. |
LogTransValue | Управляет преобразованием данных в X и Y от естественной шкалы до шкалы log2. Задайте LogTransValue на false, когда данные уже являются масштабом log2. По умолчанию это true, что предполагает, что данные являются естественной шкалой. |
FactorLinesValue | Добавляет линии к графику, показывающему коэффициент N изменение. Значение по умолчанию является 2, что соответствует уровню 1 и -1 по шкале log2.Совет Вы также можете изменять линии фактора в интерактивном режиме, после создания графика. |
TitleValue | Вектор символов, задающая заголовок для графика. |
LabelsValue | Массив ячеек из символьных векторов или строкового вектора, содержащий метки для данных. Если метки заданы, то при щелчке по точке на графике отображается метка, соответствующая этой точке. |
NormalizeValue | Управляет отображением низких нормированных значений коэффициента. Введите true отображение и понижение нормированных значений коэффициента. По умолчанию это false. Совет Можно также нормализовать данные из окна MAIR Plot, после создания графика. |
LowessOptionsValue | Массив ячеек из одной, двух или трех пар имя свойства/ значение в любом порядке, который влияет на низкую нормализацию. Варианты для имя свойства/ значение пар:
Для получения дополнительной информации о предыдущих парах имя свойства/ значение см. |
ShowplotValue | Управление отображением графика поля точек. Варианты true (по умолчанию) или false. |
PlotOnlyValue | Управление отображением графика поля точек без компонентов пользовательского интерфейса. Варианты Примечание Если вы задаете |
Intensity | Объект DataMatrix или вектор, содержащий значения интенсивности для данных экспрессии гена микромассива, рассчитанный как:
Примечание Если |
Ratio | Объект DataMatrix или вектор, содержащий отношения данных экспрессии гена микромассива, вычисленные как log2 .Примечание Если |
H | Указатель на график. |
mairplot( создает график поля точек, который строит лог10 продукты DataX, DataY)DataX и DataY интенсивность от лога2 коэффициентов интенсивности.
[ возвращает значения интенсивности и коэффициента. Если вы задаете Intensity, Ratio]
= mairplot(DataX, DataY)'Normalize' на trueвозвращенные значения коэффициента нормированы.
[ возвращает указатель на график.Intensity, Ratio, H]
= mairplot(DataX, DataY)
... = mairplot (..., 'PropertyName', PropertyValue, ...)mairplot с необязательными свойствами, которые используют пары имя/значение свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должны быть заключены в одинарные кавычки и нечувствительны к регистру. Эти имена свойства/пары значения свойств следующие:
задает тип графика. Варианты ... = mairplot(...,
'Type', TypeValue, ...)'IR' (строит лог10 продукты DataX и DataY интенсивность от лога2 коэффициентов интенсивности) или 'MA' (графики (1/2) log2 продукта DataX и DataY интенсивность от лога2 коэффициентов интенсивности). По умолчанию это 'IR'.
... = mairplot(...,
'LogTrans', LogTransValue, ...)X и Y от натурального до логарифмической шкалы 2. Задайте LogTransValue на false, когда данные уже имеют шкалу 2 журнала. По умолчанию это true, что предполагает, что данные являются естественной шкалой.
... = mairplot(...,
'FactorLines', FactorLinesValue, ...)N
Совет
Вы также можете изменять линии фактора в интерактивном режиме, после создания графика.
... = mairplot(...,
'Title', TitleValue, ...)
... = mairplot(...,
'Labels', LabelsValue, ...)
... = mairplot(...,
'Normalize', NormalizeValue, ...)true отображение и понижение нормированных значений коэффициента. По умолчанию это false.
Совет
Можно также нормализовать данные из окна MAIR Plot, после создания графика.
... = mairplot(...,
'LowessOptions', LowessOptionsValue,
...)
'Порядок', OrderValue
'Прочный', RobustValue
'Промежуток', SpanValue
Для получения дополнительной информации о предыдущих трех парах имя свойства/ значение, см. malowess функция.
... = mairplot(...,
'Showplot', ShowplotValue, ...)true (по умолчанию) или false.
... = mairplot(...,
'PlotOnly', PlotOnlyValue, ...)true или false (по умолчанию).
Примечание
Если вы задаете 'PlotOnly' свойство к trueМетки для точек данных можно отобразить, щелкнув на точке данных, и можно по-прежнему регулировать горизонтальные линии изменения сгиба, перетаскивая мышью линии.
Далее приведен ИК- график нормализованных данных.
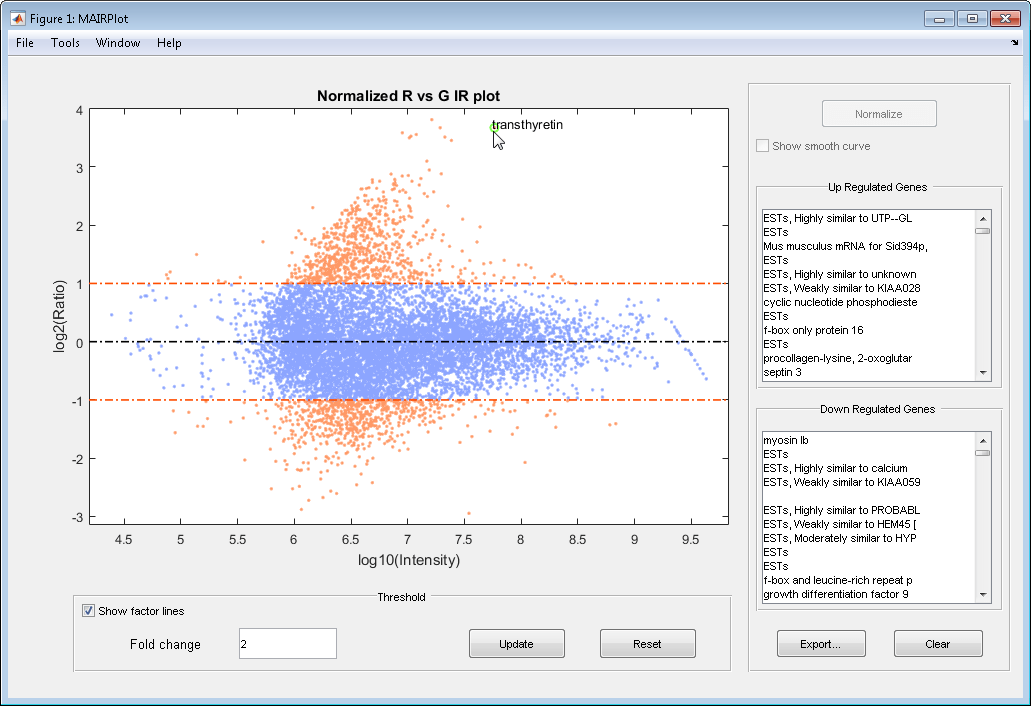
Далее приведен график MA ненормализованных данных.
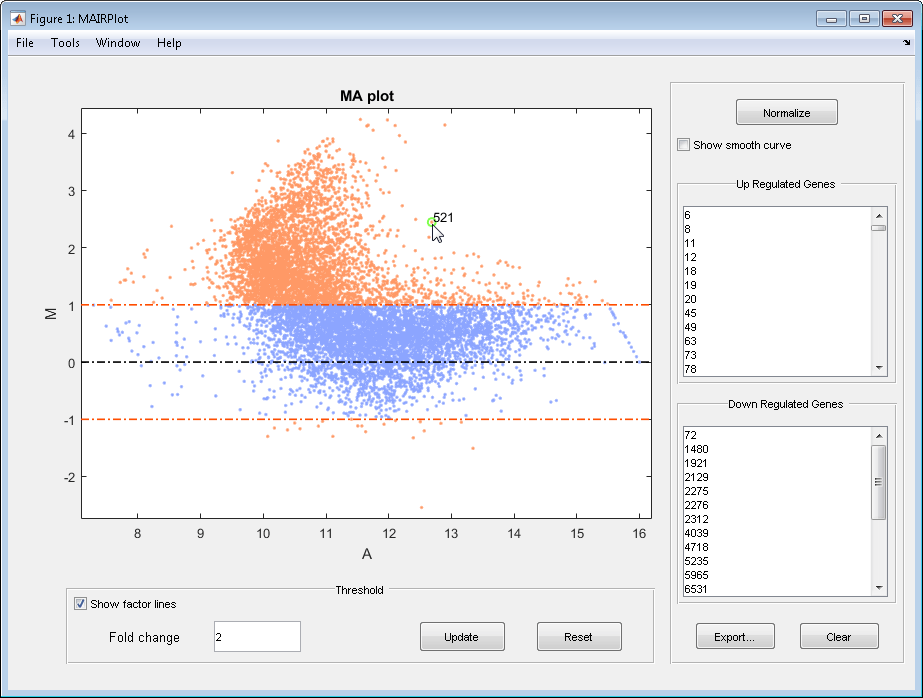
Зависимости интенсивности от графика поля точек отображают следующее:
log10 (Интенсивность) от log2 (Отношение) график поля точек генов.
Две горизонтальные линии изменения складки на уровне изменения складки 2, что соответствует отношению 1 и -1 по шкале log 2 (Ratio). (Линии будут на разных уровнях изменения сгиба, если вы использовали 'FactorLines' свойство.)
Точки данных для генов, которые считаются дифференциально экспрессированными (за пределами линий изменения складки), появляются в оранжевом цвете.
После отображения графика зависимости интенсивности от графика поля точек в интерактивном режиме сделать следующее:
Отрегулируйте горизонтальные линии поворота путем перетаскивания одной строки или ввода значения в Fold Change текстовое поле, а затем нажатия кнопки Update.
Отображение меток для точек данных путем щелчка по точке данных.
Выберите ген из списка Up Regulated или Down Regulated, чтобы выделить соответствующую точку данных на графике. Нажмите и удерживайте Ctrl или Shift, чтобы выбрать несколько генов.
Изменение масштаба графика путем выбора Tools > Zoom In или Tools > Zoom Out.
Просмотрите списки значительно повышенных и пониженных генов и, необязательно, экспортируйте метки и индексы генов в структуру в MATLAB® Рабочая область, нажав Export.
Нормализуйте данные, нажав кнопку Normalize, а затем выбрав, показывать ли нормализованный график в отдельном окне. Если вы показываете нормированный график в отдельном окне, флажок Show smooth curve становится доступным на исходном (ненормализованном) графике.
Совет
Чтобы выбрать различные опции пониженной нормализации перед нормализацией, выберите Tools > Set LOWESS Normalization Options, а затем введите опции в диалоговом окне Опции для LOWESS (Options for LOWESS).
Используйте gprread функция для создания структуры, содержащей данные микромассивов.
maStruct = gprread('mouse_a1wt.gpr');Используйте magetfield функция для извлечения зеленых (cy3) и красных (cy5) сигналов из структуры.
cy5data = magetfield(maStruct,'F635 Median'); cy3data = magetfield(maStruct,'F532 Median');
Создайте зависимость интенсивности от графика поля точек данных cy3 и cy5. Нормализуйте данные и добавьте заголовок и метки:
mairplot(cy5data, cy3data, 'Normalize', true, ... 'Title','Normalized R vs G IR plot', ... 'Labels', maStruct.Names)
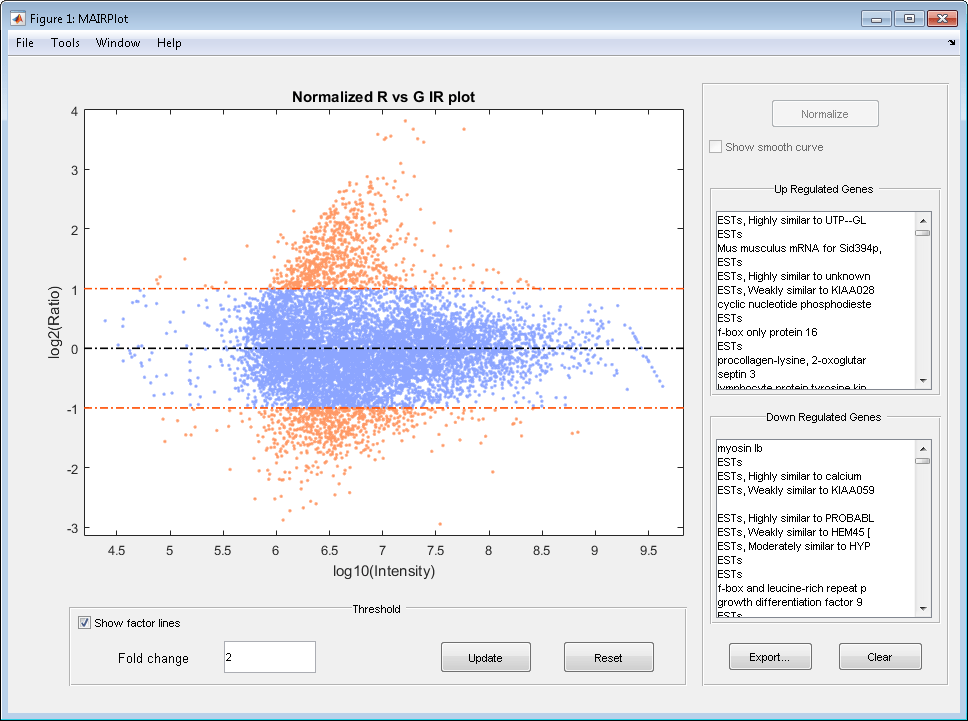
Возвращает значения интенсивности и коэффициенты без отображения графика.
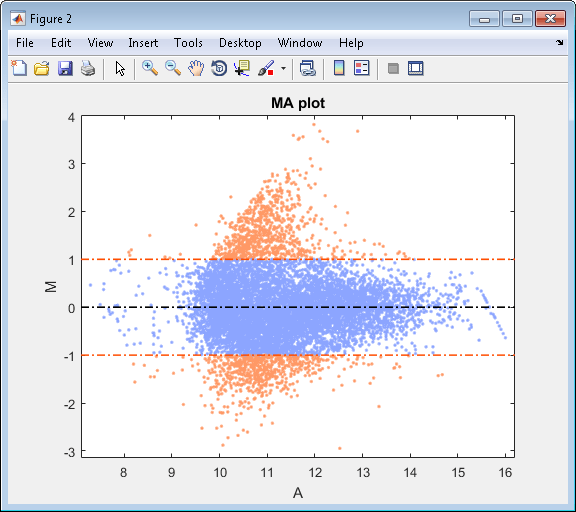
[intensities, ratios] = mairplot(cy5data, cy3data, 'Showplot', false);Создайте нормированный график MA данных cy3 и cy5 без компонентов пользовательского интерфейса.
mairplot(cy5data, cy3data, 'Normalize', true, ... 'Type','MA','PlotOnly',true)
[1] Quackenbush, J. (2002). Нормализация и преобразование данных микромассивов. Nature Genetics Suppl. 32, 496-501.
[2] Dudoit, S., Yang, Y.H., Callow, M.J., and Speed, T.P. (2002). Статистические методы для идентификации дифференциально экспрессируемых генов в экспериментах с реплицированным микромассивом кДНК. Статистика Синица 12, 111-139.
maboxplot | magetfield | maimage | mainvarsetnorm | maloglog | malowess | manorm | mattest | mavolcanoplot