Выполните t-тест с двумя образцами, чтобы оценить дифференциальную экспрессию генов из двух экспериментальных условий или фенотипов
PValues = mattest(DataX,
DataY)
[PValues, TScores] = mattest(DataX, DataY)
[PValues, TScores, DFs]
= mattest(DataX, DataY)
... = mattest(..., 'VarType', VarTypeValue, ...)
... = mattest(..., 'Permute', PermuteValue, ...)
... = mattest(..., 'Bootstrap', BootstrapValue, ...)
... = mattest(..., 'Showhist', ShowhistValue, ...)
... = mattest(..., 'Showplot', ShowplotValue, ...)
... = mattest(..., 'Labels', LabelsValue, ...)
DataX, DataY | Объект DataMatrix или матрица значений экспрессии генов, где каждая строка соответствует гену, и каждый столбец соответствует репликации.
|
VarTypeValue | Вектор символов, который задает тип отклонения теста. VarTypeValue можно 'equal' или 'unequal' (по умолчанию). Если установлено значение 'equal', mattest выполняет тест, предполагая, что две выборки имеют равные отклонения. Если установлено значение 'unequal', mattest выполняет тест, предполагая, что две выборки имеют неизвестные и неравные отклонения. |
PermuteValue | Управляет, выполняются ли тесты сочетания, и, если да, то сколько. Варианты true, false (по умолчанию), или любое целое число, больше 2. Если установлено значение true, количество сочетаний 1000. |
BootstrapValue | Определяет, выполняются ли тесты bootstrap, и, если да, то сколько. Варианты true, false (по умолчанию), или любое целое число, больше 2. Если установлено значение true, количество тестов bootstrap 1000. |
ShowhistValue | Управляет отображением гистограмм распределений t-значений и распределений p-значений. Варианты |
ShowplotValue | Управляет отображением нормального графика квантиля t-балла. Варианты |
LabelsValue | Массив ячеек из символьных векторов или строкового вектора, содержащего метки (обычно имена генов или идентификаторы набора зондов) для каждой строки в DataX и DataY. Метки отображаются, если щелкнуть точку данных на графике квантиля t-балла. |
PValues | Одно из следующих:
|
TScores | Вектор-столбец t-оценок для каждого гена в DataX и DataY. |
DFs | Вектор-столбец, содержащая степень свободы для каждого гена в DataX и DataY. |
PValues = mattest(DataX,
DataY)DataX и DataY и возвращает p-значение для каждого гена. DataX и DataY являются либо объектом DataMatrix, либо матрицей значений экспрессии генов, в которой каждая строка соответствует гену, и каждый столбец соответствует репликации. DataX содержит данные одного экспериментального условия и DataY содержит данные другого экспериментального условия. DataX и DataY должны иметь одинаковое число строк и считаются нормально распределенными в каждом классе. PValues является вектором-столбцом с p-значениями для каждого гена или, если хотя бы один из входов является объектом DataMatrix, объектом DataMatrix с именами строк, такими же как и первый входной объект DataMatrix и именем столбца p-values.
[ также возвращает t-счет для каждого гена в PValues, TScores] = mattest(DataX, DataY)DataX и DataY. TScores представляет собой вектор-столбец t-оценок для каждого гена.
[ также возвращается PValues, TScores, DFs]
= mattest(DataX, DataY)DFs, вектор-столбец, содержащая степень свободы для каждого гена в обоих наборах данных, DataX и DataY.
... = mattest (..., вызывает 'PropertyName', PropertyValue, ...)mattest с необязательными свойствами, которые используют пары имя/значение свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должны быть заключены в одинарные кавычки и нечувствительны к регистру. Эти имена свойства/пары значения свойств следующие:
... = mattest(..., 'VarType', задает тип отклонения теста. VarTypeValue, ...)VarTypeValue можно 'equal' или 'unequal' (по умолчанию). Если установлено значение 'equal', mattest выполняет тест, предполагая, что две выборки имеют равные отклонения. Если установлено значение 'unequal', mattest выполняет тест, предполагая, что две выборки имеют неизвестные и неравные отклонения.
... = mattest(..., 'Permute', управляет, выполняются ли тесты сочетания, и если да, то сколько. PermuteValue, ...)PermuteValue можно true, false (по умолчанию), или любое целое число, больше 2. Если установлено значение true, количество сочетаний 1000.
... = mattest(..., 'Bootstrap', определяет, выполняются ли тесты bootstrap, и если да, то сколько. BootstrapValue, ...)BootstrapValue можно true, false (по умолчанию), или любое целое число, больше 2. Если установлено значение true, количество тестов bootstrap 1000.
... = mattest(..., 'Showhist', управляет отображением гистограмм распределений t-значений и распределений p-значений. Когда ShowhistValue, ...)ShowhistValue является true, mattest отображает гистограммы. По умолчанию это false.
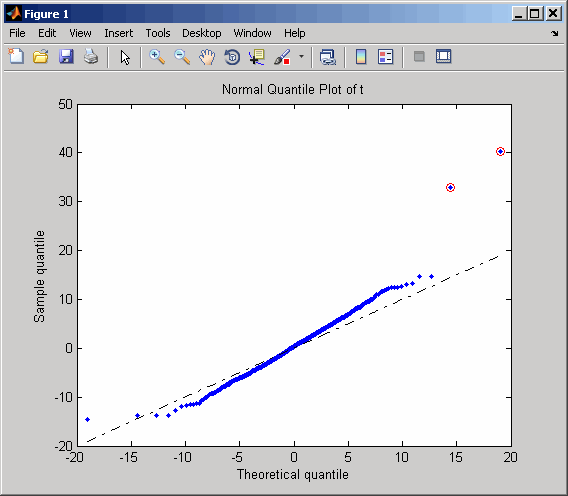
... = mattest(..., 'Showplot', управляет отображением нормального графика квантиля t-балла. Когда ShowplotValue, ...)ShowplotValue является true, mattest отображает график квантиль-квантиль. По умолчанию это false. На графике квантиля t-балла черная диагональная линия представляет собой выборочную квантилю, равную теоретической квантиле. Точки данных генов, считающихся дифференциально экспрессированными, находятся дальше от этой линии. В частности, точки данных с t-оценками > (1 - 1/(2N)) или < 1/(2N) отображение с красными кругами. N - общее количество генов.
... = mattest(..., 'Labels', управляет отображением меток при клике по точке данных на графике квантиля t-балла. LabelsValue, ...)LabelsValue - массив ячеек из векторов символов или строкового вектора, содержащего метки (обычно имена генов или идентификаторы набора зондов) для каждой строки в DataX и DataY.
Загрузите MAT-файл, включенный в программное обеспечение Bioinformatics Toolbox™, которое содержит Affymetrix® данные исследования рака предстательной железы, в частности данные о интенсивности зондирования из Affymetrix HG-U133A GeneChip® массивы. Две переменные в MAT-файле, dependentData и independentData, являются двумя матрицами значений экспрессии генов из двух экспериментальных условий.
load prostatecancerexpdataВычислите значения p и t-оценки для значений экспрессии генов в двух матрицах и отобразите нормальный график квантиля t-балла.
[pvalues,tscores] = mattest(dependentData, independentData,... 'showplot',true);
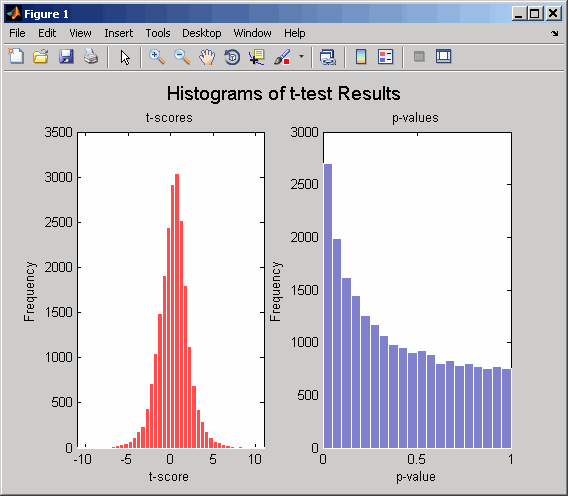
Снова вычислите значения p и t-оценки с помощью тестов сочетания (1000 сочетания) и отображения гистограмм распределений t-оценок и распределений p-значений.
[pvalues,tscores] = mattest(dependentData,independentData,... 'permute',true,'showhist',true,... 'showplot',true);
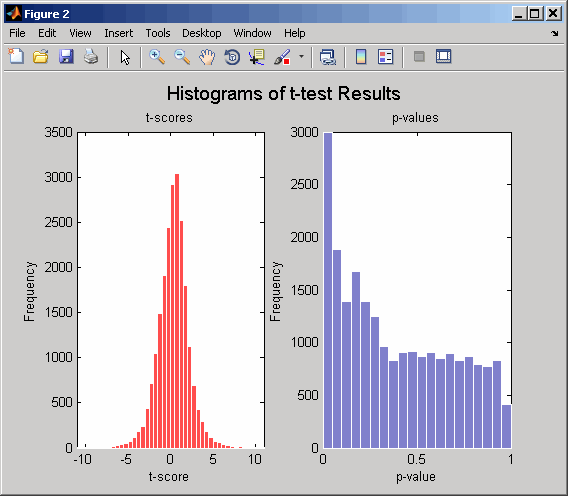
Снова вычислите значения p и t-оценки с помощью тестов bootstrap (2000 тестов) и отображения гистограмм распределений t-баллов и распределений p-значений.
[pvalues,tscores] = mattest(dependentData,independentData,... 'bootstrap',2000,'showhist',true,... 'showplot',true);
The prostatecancerexpdata.mat файл, используемый в этом примере, содержит данные Best et al., 2005.
[1] Review Literature: Huber, W., von Heydebreck, A., Sültmann, H., Poustka, A., and Vingron, M. (2002). Стабилизация отклонения, применяемая к калибровке данных микромассивов и к количественной оценке дифференциальной экспрессии. Биоинформатика 18 (Дополнение 1), S96-S104.
[2] Best, C.J.M., Gillespie, J.W., Yi, Y., Chandramouli, G.V.R., Perlmutter, M.A., Собери, Я., Эриксон, Х.С., Георгиевич, Л., Тангрея, М.А., Duray, P.H., Gonsalez, S., Velasco, A., Linehan, W.M., Matusik, R.J., Price, D.K., Figg, W.D., Emmert-Buck, M.R., and Chuaqui, R.F. (2005). Молекулярные изменения при первичном раке предстательной железы после андрогенной абляции. Клинические исследования рака 11, 6823-6834.
affygcrma | affyrma | maboxplot | mafdr | mainvarsetnorm | mairplot | maloglog | malowess | manorm | mavolcanoplot | rmasummary