Выполните настройку фона GC Robust Multi-array Average (GCRMA) на данных уровня зонда микромассива Affymetrix с использованием информации о последовательности
PMMatrix_Adj = gcrmabackadj(PMMatrix, MMMatrix, AffinPM, AffinMM)
[PMMatrix_Adj, nsbStruct]
= gcrmabackadj(PMMatrix, MMMatrix, AffinPM, AffinMM)
... = gcrmabackadj( ...'OpticalCorr', OpticalCorrValue,
...)
... = gcrmabackadj( ...'CorrConst', CorrConstValue,
...)
... = gcrmabackadj( ...'Method', MethodValue,
...)
... = gcrmabackadj( ...'TuningParam', TuningParamValue,
...)
... = gcrmabackadj( ...'AddVariance', AddVarianceValue,
...)
... = gcrmabackadj( ...'GSBCorr', GSBCorrValue,
...)
... = gcrmabackadj( ...'Showplot', ShowplotValue,
...)
... = gcrmabackadj( ...'Verbose', VerboseValue,
...)
PMMatrix | Матрица значений интенсивности, где каждая строка соответствует зонду совершенного соответствия (PM), и каждый столбец соответствует Affymetrix® Файл CEL. (Каждый файл CEL генерируется из отдельного чипа. Все чипы должны быть одного типа.) Совет Можно использовать
|
MMMatrix | Матрица значений интенсивности, где каждая строка соответствует зонду несоответствия (MM), и каждый столбец соответствует файлу Affymetrix CEL. (Каждый файл CEL генерируется из отдельного чипа. Все чипы должны быть одного типа.) Совет Можно использовать
|
AffinPM | Вектор-столбец сродства зонда PM, такие как возвращенные affyprobeaffinities функция. Каждая строка соответствует зонду. |
AffinMM | Вектор-столбец сродства зонда MM, такие как возвращенные affyprobeaffinities функция. Каждая строка соответствует зонду. |
OpticalCorrValue | Управляет использованием оптической коррекции фона на значениях интенсивности зонда PM и MM в PMMatrix и MMMatrix. Варианты true (по умолчанию) или false. |
CorrConstValue | Значение, которое задает константу корреляции, rho, для интенсивности фона журнала для каждой пары зондов PM/MM. Варианты являются любым значением ≥ 0 и ≤ 1. По умолчанию это 0.7. |
MethodValue | Вектор символов или строка, которая задает метод для оценки сигнала. Варианты 'MLE', более быстрый, специальный метод оценки максимальных вероятностей или 'EB', более медленный, формальный, эмпирический метод Байеса. По умолчанию это 'MLE'. |
TuningParamValue | Значение, которое задает параметр настройки, используемый методом оценки. Эта настройка наборов параметров нижнюю границу значений сигналов с положительной вероятностью. Выбор является положительным значением. По умолчанию это 5 (MLE) или 0.5 (EB).Совет Для получения информации об определении настройки для этого параметра см. Wu et al., 2004. |
AddVarianceValue | Управляет тем, добавлено ли отклонение сигнала к функции веса для сглаживания низкого ребра сигнала. Варианты true или false (по умолчанию). |
GSBCorrValue | Определяет, выполнять ли коррекцию геноспецифического связывания (GSB) с помощью данных о сродстве зондов. Варианты true (по умолчанию) или false. Если нет информации о сродстве зонда, это свойство игнорируется. |
ShowplotValue | Управляет отображением графика, показывающего лог2 значений интенсивности зонда из заданного столбца (чипа) в MMMatrix, от сродства зонда к AffinMM. Варианты true, false, или I, целое число, задающее столбец в MMMatrix. Если установлено значение true, первый столбец в MMMatrix нанесен на график. По умолчанию это:
|
VerboseValue | Управляет отображением отчета о прогрессе, показывающего количество каждого чипа по мере его завершения. Варианты true (по умолчанию) или false. |
PMMatrix_Adj | Матрица значений интенсивности скорректированного фона PM (идеальное соответствие). |
nsbStruct | Структура, содержащая неспецифические фоновые параметры связывания, оцененные из интенсивности и сродства зондов на Affymetrix GeneChip® массив.
|
PMMatrix_Adj = gcrmabackadj(PMMatrix, MMMatrix, AffinPM, AffinMM)PMMatrix_Adj, матрица скорректированных по фону значений интенсивности PM (идеально соответствующих).
Примечание
Если AffinPM и AffinMM данные недоступны, вы все еще можете использовать gcrmabackadj функция путем ввода пустых векторов-столбцов для обоих из этих входов в синтаксисе.
[ возвращает PMMatrix_Adj, nsbStruct]
= gcrmabackadj(PMMatrix, MMMatrix, AffinPM, AffinMM)nsbStruct, структуру, содержащую неспецифические параметры фона связывания, оцененные по интенсивности и аффинности зондов на массиве Affymetrix GeneChip. nsbStruct включает следующие поля:
sigma
mu_pm
mu_mm
... = gcrmabackadj (... вызывает 'PropertyName', PropertyValue, ...)gcrmabackadj с необязательными свойствами, которые используют пары имя/значение свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должны быть заключены в одинарные кавычки и нечувствительны к регистру. Эти имена свойства/пары значения свойств следующие:
... = gcrmabackadj( ...'OpticalCorr', управляет использованием оптической коррекции фона на значениях интенсивности зонда PM и MM в OpticalCorrValue,
...)PMMatrix и MMMatrix. Варианты true (по умолчанию) или false.
... = gcrmabackadj( ...'CorrConst', задает константу корреляции rho для интенсивности фона журнала для каждой пары зондов PM/MM. Варианты являются любым значением CorrConstValue,
...)≥ 0 и ≤ 1. По умолчанию это 0.7.
... = gcrmabackadj( ...'Method', задает метод оценки сигнала. Варианты MethodValue,
...)MLE, более быстрый, специальный метод оценки максимальных вероятностей или EB, более медленный, формальный, эмпирический метод Байеса. По умолчанию это MLE.
... = gcrmabackadj( ...'TuningParam', задает параметр настройки, используемый методом оценки. Эта настройка наборов параметров нижнюю границу значений сигналов с положительной вероятностью. Выбор является положительным значением. По умолчанию это TuningParamValue,
...)5 (MLE) или 0.5 (EB).
Совет
Для получения информации об определении настройки для этого параметра см. Wu et al., 2004.
... = gcrmabackadj( ...'AddVariance', управляет, добавляется ли отклонение сигнала к весовой функции для сглаживания низкого ребра сигнала. Варианты AddVarianceValue,
...)true или false (по умолчанию).
... = gcrmabackadj( ...'GSBCorr', определяет, выполнять ли коррекцию генно-специфического связывания (GSB) с помощью данных о сродстве зонда. Варианты GSBCorrValue,
...)true (по умолчанию) или false. Если нет информации о сродстве зонда, это свойство игнорируется.
... = gcrmabackadj( ...'Showplot', управляет отображением графика, показывающего лог2 значений интенсивности зонда из заданного столбца (микросхемы) в ShowplotValue,
...)MMMatrix, от сродства зонда к AffinMM. Варианты true, false, или I, целое число, задающее столбец в MMMatrix. Если установлено значение true, первый столбец в MMMatrix нанесен на график. По умолчанию это:
false - Когда заданы возвращаемые значения.
true - Когда значения возврата не заданы.
... = gcrmabackadj( ...'Verbose', управляет отображением отчета о прогрессе, показывающего количество каждого чипа по мере его завершения. Варианты VerboseValue,
...)true (по умолчанию) или false.
Загрузите MAT-файл, включенный в программное обеспечение Bioinformatics Toolbox™, которое содержит данные Affymetrix из исследования рака предстательной железы. Переменные в MAT-файле включают seqMatrix, матрица, содержащая информацию о последовательности для PM-зондов, pmMatrix и mmMatrixматрицы, содержащие значения интенсивности зонда PM и MM, и probeIndicesВектор-столбец, содержащая информацию индексации зонда.
load prostatecancerrawdataВычислите сходство зондов Affymetrix PM и MM из их последовательностей и интенсивности зондов MM.
[apm, amm] = affyprobeaffinities(seqMatrix, mmMatrix(:,1),... 'ProbeIndices', probeIndices);
Выполните настройку фона GCRMA на данных уровня зонда микромассива Affymetrix, создав матрицу скорректированных значений интенсивности ТЧ фона. Кроме того, отобразите график, показывающий лог2 значений интенсивности зонда из столбца 3 (микросхема 3) в mmMatrix, от сродства зонда к amm.
pms_adj = gcrmabackadj(pmMatrix, mmMatrix, apm, amm, 'showplot', 3);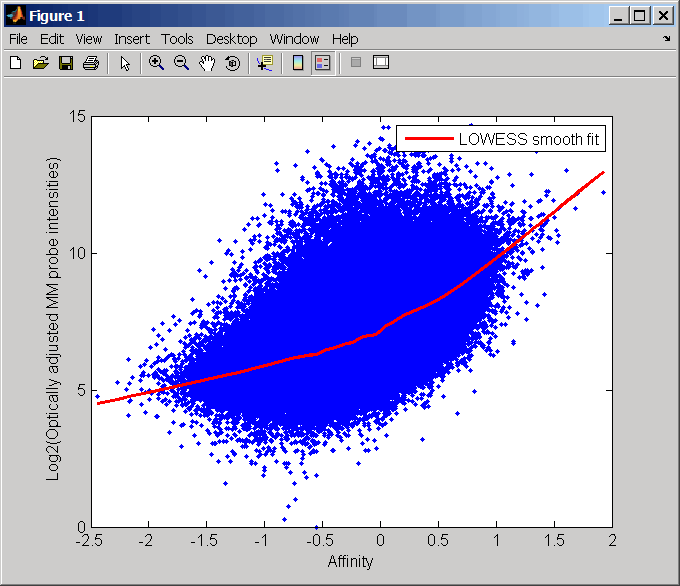
Снова выполните настройку фона GCRMA, используя более медленный, формальный, эмпирический метод Байеса.
pms_adj2 = gcrmabackadj(pmMatrix, mmMatrix, apm, amm, 'method', 'EB');
The prostatecancerrawdata.mat файл, используемый в этом примере, содержит данные Best et al., 2005.
[1] Wu, Z., Irizarry, R.A., Gentleman, R., Murillo, F.M., and Spencer, F. (2004). Модельная корректировка фона для массивов экспрессии олигонуклеотидов. Журнал Американской статистической ассоциации 99 (468), 909-917.
[2] Wu, Z., and Irizarry, R.A. (2005). Стохастические модели, вдохновленные теорией гибридизации для коротких олигонуклеотидных массивов. Материалы RECOMB 2004. J Comput Biol. 12 (6), 882-93.
[3] Wu, Z., and Irizarry, R.A. (2005). Статистическая среда для анализа данных уровня зонда микромассивов. Университет Джона Хопкинса, рабочие документы по биостатистике 73.
[4] Wu, Z., and Irizarry, R.A. (2003). Модельная корректировка фона для массивов экспрессии олигонуклеотидов. Семинар RSS по экспрессии генов, Уай, Англия, http://biosun01.biostat.jhsph.edu/%7Eririzarr/Talks/gctalk.pdf.
[5] Abd Rabbo, N.A., and Barakat, H.M. (1979). Оценочные задачи при двухмерном логнормальном распределении. Индиец J. Pure Appl. Math 10 (7), 815-825.
[6] Best, C.J.M., Gillespie, J.W., Yi, Y., Chandramouli, G.V.R., Perlmutter, M.A., Собери, Я., Эриксон, Х.С., Георгиевич, Л., Тангрея, М.А., Duray, P.H., Gonsalez, S., Velasco, A., Linehan, W.M., Matusik, R.J., Price, D.K., Figg, W.D., Emmert-Buck, M.R., and Chuaqui, R.F. (2005). Молекулярные изменения при первичном раке предстательной железы после андрогенной абляции. Клинические исследования рака 11, 6823-6834.
affygcrma | affyprobeseqread | affyread | celintensityread | probelibraryinfo