Выполните настройку фона на данных уровня зонда микромассивов Affymetrix с помощью зонального метода
BackAdjustedData = zonebackadj(Data)
[BackAdjustedData, ZoneStruct]
= zonebackadj(Data)
[BackAdjustedData, ZoneStruct, Background]
= zonebackadj(Data)
... = zonebackadj(Data,
...'NumZones', NumZonesValue, ...)
... = zonebackadj(Data,
...'Percent', PercentValue, ...)
... = zonebackadj(Data,
...'SmoothFactor', SmoothFactorValue, ...)
... = zonebackadj(Data,
...'NoiseFrac', NoiseFracValue, ...)
... = zonebackadj(Data,
...'CDF', CDFValue, ...)
... = zonebackadj(Data,
...'Mask', MaskValue, ...)
... = zonebackadj(Data,
...'Showplot', ShowplotValue, ...)
Data | Одно из следующих:
|
NumZonesValue | Скалярный или двухэлементный вектор, который задает количество зон для использования в настройке фона. Если скаляр, это должно быть квадратное число. Если вектор с двумя элементами, первый элемент задает количество строк, а второй элемент задает количество столбцов в неквадратичной сетке. По умолчанию это 16. |
PercentValue | Значение, которое задает процент, P, таким образом, чтобы самый низкий P для оценки фона для этой зоны используется процент значений ранжированной интенсивности из каждой зоны. По умолчанию это 2. |
SmoothFactorValue | Значение, задающее коэффициент сглаживания, используемый при вычислении взвешенного среднего значения вкладов каждой зоны в фон точки. По умолчанию это 100. |
NoiseFracValue | Значение, которое задает долю шума, NF, таким образом, значение, скорректированное по фону, определяется max((Intensity - WeightedBackground), NF*LocalNoiseEstimate). По умолчанию это 0.5. |
CDFValue | Одно из следующих:
|
MaskValue | Логический вектор, который определяет, какие камеры маскировать и не использовать в фоновых оценках. В векторе 0 = not masked и 1 = masked. По умолчанию это значения в Masked столбец Probes поле файла CEL. |
ShowplotValue | Управляет графическим изображением изображения фоновых оценок. Варианты |
BackAdjustedData | Матрица или массив ячеек из векторов, содержащих скорректированные по фону значения интенсивности зонда. |
ZoneStruct | Структура MATLAB, содержащая центры зон, используемых для настройки фона, и оценки значений фона в центре каждой зоны. |
Background | Матрица или массив ячеек из векторов, содержащих предполагаемые фоновые значения для каждого зонда. |
BackAdjustedData = zonebackadj(Data)Data, который содержит интенсивность зондирования из файлов Affymetrix CEL. Детали фоновой корректировки описаны в документе «Описание статистических алгоритмов».
[ также возвращает структуру, содержащую центры зон, используемых для настройки фона, и оценки значений фона в центре каждой зоны. BackAdjustedData, ZoneStruct]
= zonebackadj(Data)
[ также возвращает матрицу или массив ячеек из векторов, содержащих предполагаемые значения фона для каждого зонда. BackAdjustedData, ZoneStruct, Background]
= zonebackadj(Data)
... = zonebackadj вызывает (Data... 'PropertyName', PropertyValue, ...)zonebackadj с необязательными свойствами, которые используют пары имя/значение свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должны быть заключены в одинарные кавычки и нечувствительны к регистру. Эти имена свойства/пары значения свойств следующие:
... = zonebackadj( задает количество зон, используемых в настройке фона. Data,
...'NumZones', NumZonesValue, ...)NumZonesValue может быть либо скаляром, который является квадратным числом, либо массивом с двумя элементами, в котором первый элемент задает количество строк, а второй элемент задает количество столбцов в нескверной сетке. По умолчанию это 16.
... = zonebackadj( задает процент, Data,
...'Percent', PercentValue, ...)P, таким образом, чтобы самый низкий P для оценки фона для этой зоны используется процент значений ранжированной интенсивности из каждой зоны. По умолчанию это 2.
... = zonebackadj( задает коэффициент сглаживания, используемый при вычислении взвешенного среднего значения вкладов каждой зоны в фон точки, обеспечивая таким образом плавный переход между зонами. По умолчанию это Data,
...'SmoothFactor', SmoothFactorValue, ...)100.
... = zonebackadj( задает долю шума, так что скорректированное по фону значение задается как Data,
...'NoiseFrac', NoiseFracValue, ...)max((Intensity - WeightedBackground), NF*LocalNoiseEstimate), где NF является NoiseFracValue. По умолчанию это 0.5.
... = zonebackadj( задает файл или структуру библиотеки Affymetrix CDF, которая задает камеры, не используемые в фоновых оценках.Data,
...'CDF', CDFValue, ...)
... = zonebackadj( задает логический вектор, который определяет, какие камеры маскировать и не использовать в фоновых оценках. В векторе Data,
...'Mask', MaskValue, ...)0 = not masked и 1 = masked. По умолчанию это значения в Masked столбец Probes поле файла CEL.
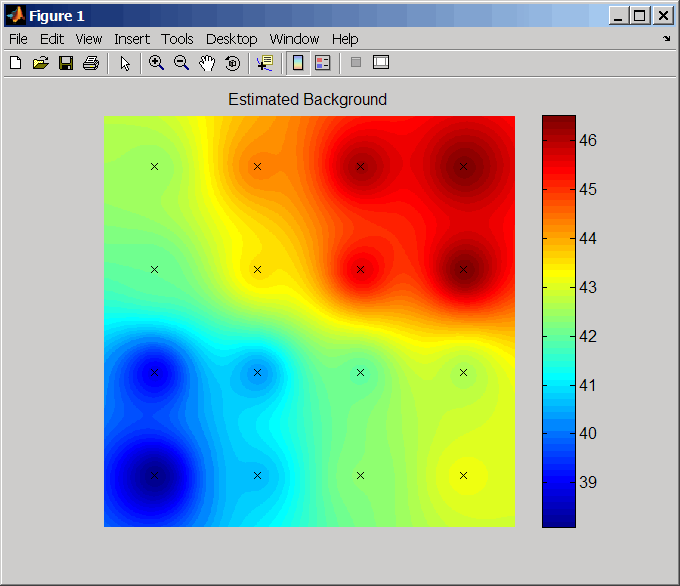
... = zonebackadj( строит график изображения фоновых оценок. Варианты Data,
...'Showplot', ShowplotValue, ...)true или false (по умолчанию).
[1] Описание статистических алгоритмов Документ, https://www.affymetrix.com/support/technical/whitepapers/ sadd_whitepaper.pdf
affyinvarsetnorm | affyread | celintensityread | gcrma | gcrmabackadj | probelibraryinfo | probesetlink | probesetlookup | probesetvalues | quantilenorm | rmabackadj | rmasummary