Глобально выровнять две последовательности с помощью алгоритма Нидлемана-Вунша
Score = nwalign(Seq1,Seq2)
[Score, Alignment] = nwalign(Seq1,Seq2)
[Score, Alignment, Start]
= nwalign(Seq1,Seq2)
... = nwalign(Seq1,Seq2,
...'Alphabet', AlphabetValue, ...)
... = nwalign(Seq1,Seq2,
...'ScoringMatrix', ScoringMatrixValue,
...)
... = nwalign(Seq1,Seq2,
...'Scale', ScaleValue, ...)
... = nwalign(Seq1,Seq2,
...'GapOpen', GapOpenValue, ...)
... = nwalign(Seq1,Seq2,
...'ExtendGap', ExtendGapValue, ...)
... = nwalign(Seq1,Seq2,
...'Glocal', GlocalValue, ...)
... = nwalign(Seq1,Seq2,
...'Showscore', ShowscoreValue, ...)
Seq1, Seq2 | Аминокислотные или нуклеотидные последовательности. Введите любое из следующих значений: Совет Для помощи с буквенными и целочисленными представлениями аминокислот и нуклеотидов, смотрите Amino Acid Lookup или Nucleotide Lookup. |
AlphabetValue | Вектор символов или строка, задающая тип последовательности. Варианты 'AA' (по умолчанию) или 'NT'. |
ScoringMatrixValue | Одно из следующих:
Примечание Если вам нужно скомпилироваться |
ScaleValue | Положительное значение, которое задает коэффициент шкалы, который применяется к выходу счета. Например, если выходной счет первоначально определен в битах, и вы вводите По умолчанию это Примечание Если на Совет Перед сравнением счетов выравнивания из нескольких трасс убедитесь, что счета указаны в тех же модулях. Можно использовать |
GapOpenValue | Положительное значение, определяющее штраф за открытие зазора в выравнивании. По умолчанию это 8. |
ExtendGapValue | Положительное значение, определяющее штраф за расширение разрыва с помощью схемы штрафа за аффинную разрыв. Примечание Если вы задаете это значение, |
GlocalValue | Управляет возвратом полуглобального или «глокального» выравнивания. В полуглобальном выравнивании штрафы за разрыв в конце последовательностей являются нулевыми. Варианты true или false (по умолчанию). |
ShowscoreValue | Управление отображением пространства подсчета очков и выигрышного пути выравнивания. Варианты true или false (по умолчанию). |
Score | Оптимальный глобальный счет выравнивания в битах. |
Alignment | 3-by-N символьный массив, показывающий две последовательности, Seq1 и Seq2, в первой и третьей строках и символы, представляющие для них оптимальное глобальное выравнивание во второй строке. |
Start | Вектор 2 на 1 индексов, указывающих начальную точку в каждой последовательности для выравнивания. Потому что это глобальное выравнивание, Start всегда [1;1]. |
Score = nwalign(Seq1,Seq2)
[ возвращает 3-by-N символьный массив, показывающий две последовательности, Score, Alignment] = nwalign(Seq1,Seq2)Seq1 и Seq2, в первой и третьей строках и символы, представляющие для них оптимальное глобальное выравнивание во второй строке. Символ | указывает аминокислоты или нуклеотиды, которые точно совпадают. Символ : указывает аминокислоты или нуклеотиды, которые связаны, как определено матрицей оценки (несовпадения с нулевым или положительным значением матрицы оценки).
[ возвращает вектор 2 на 1 индексов, указывающих начальную точку в каждой последовательности для выравнивания. Потому что это глобальное выравнивание, Score, Alignment, Start]
= nwalign(Seq1,Seq2)Start всегда [1;1].
... = nwalign вызывает (Seq1, Seq2... 'PropertyName', PropertyValue, ...)nwalign с необязательными свойствами, которые используют пары имя/значение свойства. Можно задать одно или несколько свойств в любом порядке. Каждый PropertyName должны быть заключены в одинарные кавычки и нечувствительны к регистру. Эти имена свойства/пары значения свойств следующие:
... = nwalign( задает тип последовательностей. Варианты Seq1,Seq2,
...'Alphabet', AlphabetValue, ...)'AA' (по умолчанию) или 'NT'.
... = nwalign( задает матрицу скоринга, которая будет использоваться для глобального выравнивания. По умолчанию это:Seq1,Seq2,
...'ScoringMatrix', ScoringMatrixValue,
...)
'BLOSUM50' - Когда AlphabetValue равен 'AA'
'NUC44' - Когда AlphabetValue равен 'NT'
... = nwalign( задает масштабный коэффициент, который применяется к выходному баллу, таким образом управляя единицами измерения выходного балла. Выбор является любым положительным значением.Seq1,Seq2,
...'Scale', ScaleValue, ...)
... = nwalign( определяет штраф за открытие зазора в выравнивании. Выбор является любым положительным значением. По умолчанию это Seq1,Seq2,
...'GapOpen', GapOpenValue, ...)8.
... = nwalign( определяет штраф за расширение промежутка с помощью схемы аффинного штрафа за разрыв. Выбор является любым положительным значением. Seq1,Seq2,
...'ExtendGap', ExtendGapValue, ...)
... = nwalign( управляет возвратом полуглобального или «глокального» выравнивания. В полуглобальном выравнивании штрафы за разрыв в конце последовательностей являются нулевыми. Варианты Seq1,Seq2,
...'Glocal', GlocalValue, ...)true или false (по умолчанию).
... = nwalign( управляет отображением оценочного пространства и выигрышного пути выравнивания. Варианты Seq1,Seq2,
...'Showscore', ShowscoreValue, ...)true или false (по умолчанию).
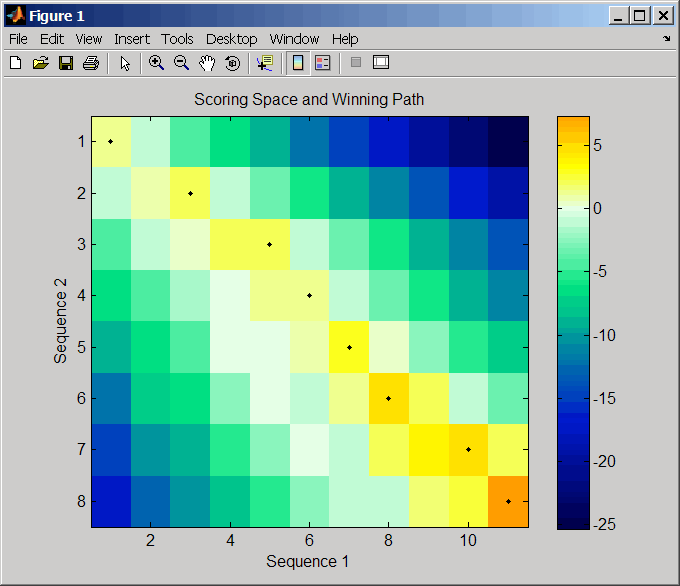
Пространство оценки является тепловой картой, отображающей лучшие счета для всех частичных выравниваний двух последовательностей. Цвет каждого (n1,n2) координата в пространстве подсчета представляет собой лучший счет для сопряжения подпоследовательностей Seq1(1:n1) и Seq2(1:n2), где n1 - положение в Seq1 и n2 - положение в Seq2. Лучший счет для сопряжения определенных подпоследовательностей определяется путем оценки всех возможных выравниваний подпоследовательностей путем суммирования совпадений и штрафов за разрыв.
Выигрышный путь представлен черными точками в скоринговом пространстве, и он иллюстрирует сопряжение позиций в оптимальном глобальном выравнивании. Цвет последней точки (нижняя правая) выигрышного пути представляет оптимальный глобальный счет выравнивания для двух последовательностей и является Score выход возвращен nwalign.
Примечание
Пространство оценки визуально указывает, существуют ли потенциальные альтернативные пути выигрыша, что полезно при выравнивании последовательностей с большими погрешностями. Визуальные шаблоны в пространстве оценки могут также указывать на возможную перестройку последовательности.
Глобально выровнять две аминокислотные последовательности с помощью BLOSUM50 (по умолчанию) матрица оценки и значения по умолчанию для GapOpen и ExtendGap свойства. Верните оптимальный счет глобального выравнивания в битах и символьном массиве выравнивания.
[Score, Alignment] = nwalign('VSPAGMASGYD','IPGKASYD') Score = 7.3333 Alignment = VSPAGMASGYD : | | || || I-P-GKAS-YD
Глобально выровняйте две аминокислотные последовательности, определяющие PAM250 матрица скоринга и разрыв открытый штраф 5.
[Score, Alignment] = nwalign('IGRHRYHIGG','SRYIGRG',... 'scoringmatrix','pam250',... 'gapopen',5) Score = 2.3333 Alignment = IGRHRYHIG-G : || || | -S--RY-IGRG
Глобально выравнивает две аминокислотные последовательности, возвращая Score в единицах nat (nats) путем определения масштабного коэффициента log(2).
[Score, Alignment] = nwalign('HEAGAWGHEE','PAWHEAE','Scale',log(2)) Score = 0.2310 Alignment = HEAGAWGHE-E || || | --P-AW-HEAE
[1] Durbin, R., Eddy, S., Krogh, A. and Mitchison, G. (1998). Анализ биологической последовательности (Cambridge University Press).
aa2int | aminolookup | baselookup | blosum | dayhoff | gonnet | int2aa | int2nt | localalign | multialign | nt2aa | nt2int | nuc44 | pam | profalign | seqdotplot | swalign